โครงงานนี้ประกอบด้วยวิธีการต่างๆ ในการสุ่มตัวอย่างประชากรและการประเมินวิธีการต่างๆ เรารวมสถานการณ์ต่างๆ มากมายที่อาจทำให้เกิดอคติต่อการประมาณระดับการติดเชื้อตามกลุ่มตัวอย่าง รวมถึงผู้ไม่ตอบสนอง อัตราผลบวกลวง/ลบ ความสามารถในการแพร่เชื้อของผู้ป่วยในช่วงระยะเวลาของการติดเชื้อ ตามแบบจำลอง EpiABM แพ็คเกจนี้ยังสามารถส่งออกวิธีการสุ่มตัวอย่างที่ดีที่สุด โดยการรันการจำลองการแพร่กระจายของโรค เพื่อดูข้อผิดพลาดในการทำนายของวิธีการสุ่มตัวอย่างแต่ละวิธี
EpiOS ยังไม่พร้อมใช้งานบน PyPI แต่สามารถติดตั้ง pip ในเครื่องได้ ควรดาวน์โหลดไดเร็กทอรีลงในเครื่องของคุณก่อน จากนั้นจึงสามารถติดตั้งได้โดยใช้คำสั่ง:
pip install -e .เราขอแนะนำให้คุณติดตั้งโมเดล EpiABM เพื่อสร้างข้อมูลการจำลองการติดเชื้อ ขั้นแรก คุณสามารถดาวน์โหลด pyEpiabm ไปยังตำแหน่งใดก็ได้บนเครื่องของคุณ จากนั้นจึงสามารถติดตั้งได้โดยใช้คำสั่ง:
pip install -e path/to/pyEpiabm สามารถเข้าถึงเอกสารได้ผ่านทางป้าย docs ด้านบน
นี่คือแผนภาพคลาส UML สำหรับโครงการของเรา: 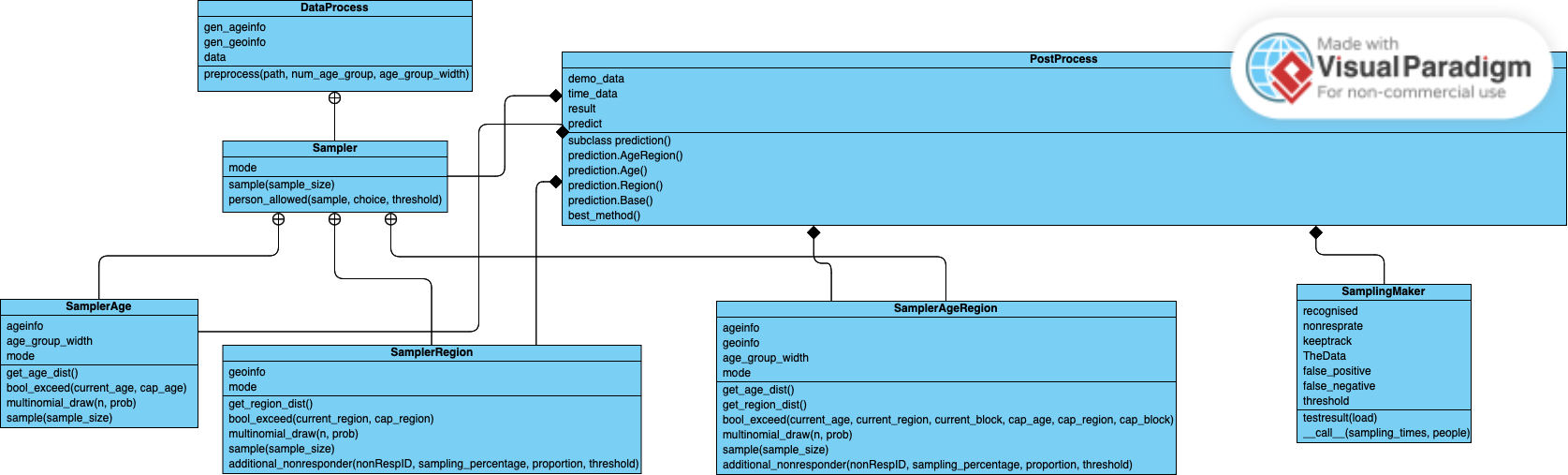
ไฟล์ params.py มีพารามิเตอร์ทั้งหมดที่จำเป็นสำหรับโมเดลนี้ นอกจากนี้ ไฟล์ในโฟลเดอร์ input ยังเป็นตัวอย่างของไฟล์ชั่วคราวที่สร้างขึ้นระหว่างการประมวลผลข้อมูลล่วงหน้า มันจะถูกใช้โดยคลาสตัวอย่าง พารามิเตอร์ data_store_path ในแต่ละคลาสแซมเพลอร์คือพาธในการจัดเก็บไฟล์เหล่านี้
PostProcess เพื่อสร้างพล็อต ประการแรก คุณต้องกำหนดออบเจ็กต์ PostProcess ใหม่และป้อนข้อมูล demodata ข้อมูลประชากรและข้อมูล timedata การติดเชื้อที่สร้างจาก pyEpiabm ประการที่สอง คุณสามารถใช้ PostProcess.predict เพื่อทำการคาดการณ์ตามวิธีการสุ่มตัวอย่างต่างๆ คุณสามารถเรียกวิธีการสุ่มตัวอย่างที่คุณต้องการใช้เป็นวิธีการได้โดยตรง จากนั้นระบุจุดเวลาให้กับตัวอย่างและขนาดตัวอย่าง ในที่นี้ เราจะใช้ AgeRegion เป็นวิธีสุ่มตัวอย่าง [0, 1, 2, 3, 4, 5] เป็นจุดเวลาที่จะสุ่มตัวอย่าง และ 3 เป็นขนาดตัวอย่าง สุดท้ายนี้ คุณสามารถระบุว่าคุณต้องการพิจารณาผู้ไม่ตอบกลับหรือไม่ และคุณต้องการเปรียบเทียบผลลัพธ์กับข้อมูลจริงหรือไม่ โดยระบุพารามิเตอร์ non_responder และ comparison
สำหรับตัวอย่างโค้ด คุณสามารถดูได้ดังต่อไปนี้:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)ตอนนี้ คุณจะมีรูปของคุณบันทึกไว้ในเส้นทางที่กำหนด!
PostProcess เพื่อเลือกวิธีการสุ่มตัวอย่างที่ดีที่สุด ประการแรก คุณต้องกำหนดออบเจ็กต์ PostProcess ใหม่และป้อนข้อมูล demodata ข้อมูลประชากรและข้อมูล timedata การติดเชื้อที่สร้างจาก pyEpiabm ประการที่สอง คุณสามารถใช้ PostProcess.best_method เพื่อเปรียบเทียบประสิทธิภาพของวิธีการสุ่มตัวอย่างต่างๆ คุณสามารถระบุวิธีการที่คุณต้องการเปรียบเทียบได้ จากนั้นระบุช่วงเวลาการสุ่มตัวอย่างให้กับตัวอย่างและขนาดตัวอย่าง ประการที่สาม คุณสามารถระบุว่าคุณต้องการพิจารณาผู้ไม่ตอบกลับหรือไม่ และคุณต้องการเปรียบเทียบผลลัพธ์กับข้อมูลจริงหรือไม่ โดยระบุพารามิเตอร์ non_responder และ comparison นอกจากนี้ เนื่องจากวิธีการสุ่มตัวอย่างเป็นแบบสุ่ม คุณจึงสามารถระบุจำนวนการวนซ้ำเพื่อให้ได้ประสิทธิภาพโดยเฉลี่ยได้ นอกจากนี้ parallel_computation ยังสามารถเปิดเพื่อเพิ่มความเร็วได้อีกด้วย สุดท้าย คุณสามารถเปิด hyperparameter_autotune อัตโนมัติเพื่อค้นหาชุดค่าผสมของไฮเปอร์พารามิเตอร์ที่ดีที่สุดโดยอัตโนมัติ
สำหรับตัวอย่างโค้ด คุณสามารถดูได้ดังต่อไปนี้:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed