
พื้นที่เก็บข้อมูลนี้เป็นการดำเนินการอย่างเป็นทางการของ CELL-E 2: การแปลโปรตีนเป็นรูปภาพและย้อนกลับด้วยตัวแปลงข้อความเป็นรูปภาพแบบสองทิศทาง
สร้างสภาพแวดล้อมเสมือนและติดตั้งแพ็คเกจที่จำเป็นผ่าน:
pip install -r requirements.txt
ถัดไป ติดตั้ง torch = 2.0.0 ด้วยเวอร์ชัน CUDA ที่เหมาะสม
มีโมเดลให้เลือกบน Hugging Face
นอกจากนี้เรายังมีช่องว่างสองช่องที่คุณสามารถเรียกใช้การคาดการณ์ข้อมูลของคุณเองได้!
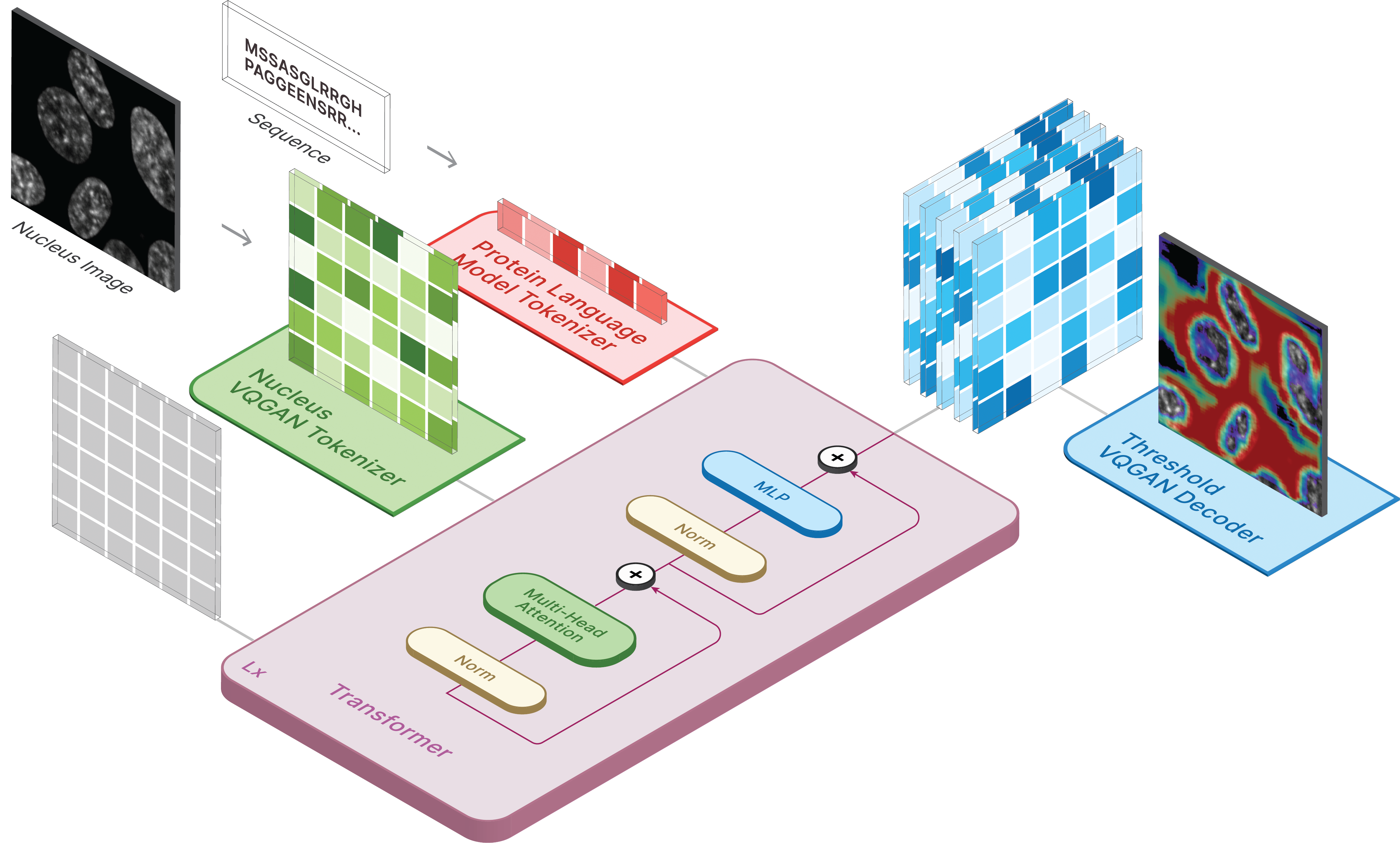
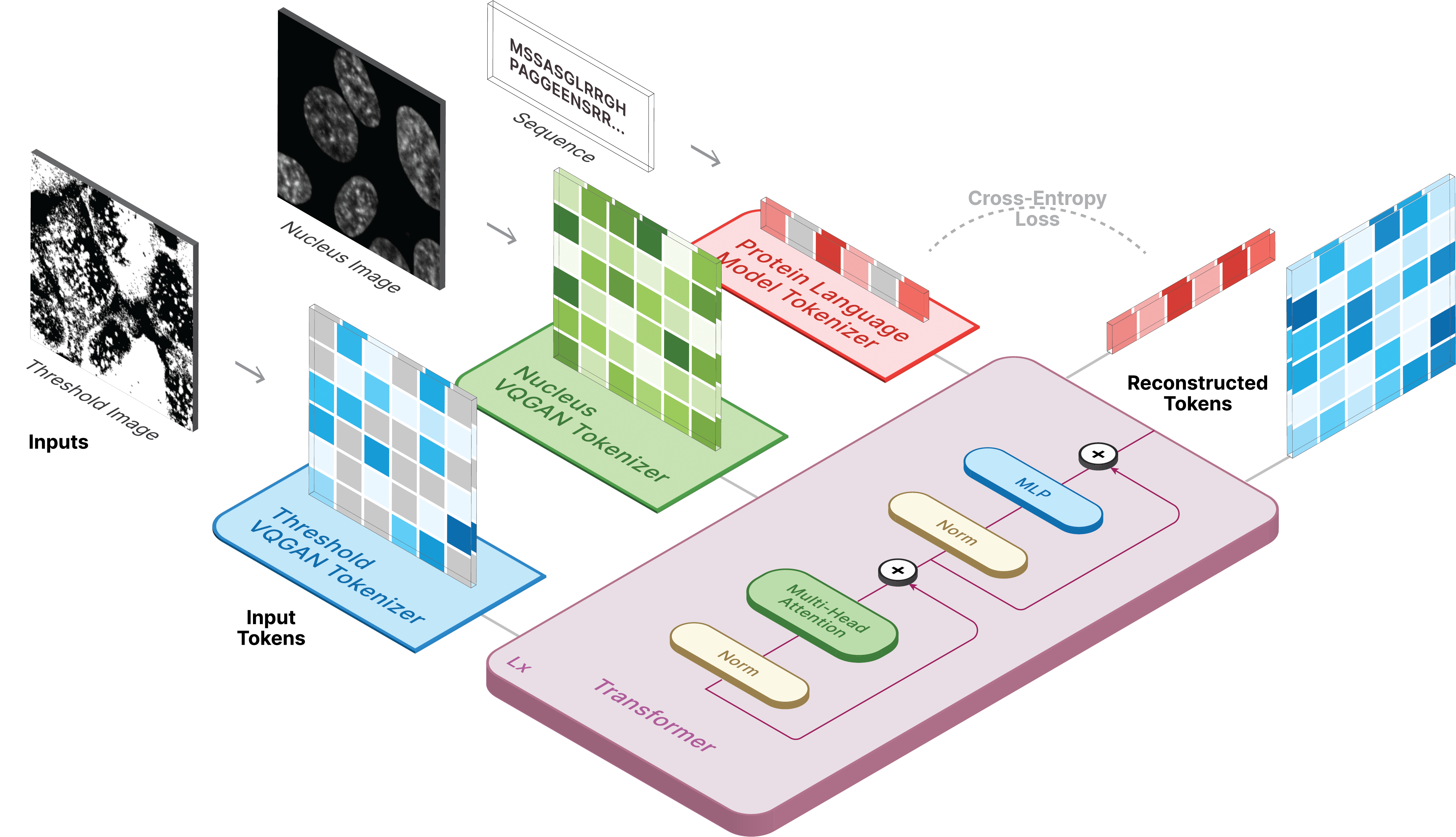
หากต้องการสร้างรูปภาพ ให้ตั้งค่าโมเดลที่บันทึกไว้เป็น ckpt_path วิธีการนี้อาจไม่เสถียร ดังนั้นโปรดดู Demo.ipynb เพื่อดูวิธีการโหลดแบบอื่น
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
progress = True )การฝึกอบรม CELL-E เกิดขึ้นใน 3 ขั้นตอน:
หากใช้อิมเมจขีดจำกัดโปรตีน ให้ตั้ง threshold: True สำหรับชุดข้อมูล
เราใช้โค้ด taming-transformers เวอร์ชันที่ได้รับการปรับเปลี่ยนเล็กน้อย
หากต้องการฝึก ให้รันสคริปต์ต่อไปนี้:
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
โปรดดู repo ดั้งเดิมสำหรับแฟล็กเพิ่มเติม เช่น --gpus
เรามีสคริปต์สำหรับการดาวน์โหลดรูปภาพ Human Protein Atlas และ OpenCell ในโฟลเดอร์สคริปต์ จำเป็นต้องมี data_csv สำหรับ dataloader คุณต้องสร้างไฟล์ CSV ซึ่งมีคอลัมน์ nucleus_image_path , protein_image_path , metadata_path , split (train หรือ val) และ sequence (ไม่บังคับ) สันนิษฐานว่าไฟล์นี้มีอยู่ในโฟลเดอร์ data ทั่วไปเดียวกันกับไฟล์รูปภาพและข้อมูลเมตา
ข้อมูลเมตาเป็น JSON ที่ควรมาพร้อมกับลำดับโปรตีนทุกลำดับ หากลำดับไม่ปรากฏใน data_csv ลำดับนั้นจะต้องปรากฏใน metadata.json โดยมีคีย์ชื่อ protein_sequence
การเพิ่มข้อมูลเพิ่มเติมที่นี่อาจเป็นประโยชน์ในการสืบค้นโปรตีนแต่ละตัว สามารถดึงข้อมูลได้ผ่านทาง retrieve_metadata ซึ่งสร้างตัวแปร self.metadata ภายในออบเจ็กต์ชุดข้อมูล
หากต้องการฝึก ให้รันสคริปต์ต่อไปนี้:
python celle_main.py --base configs/celle.yaml -t True
ระบุ --gpus ในรูปแบบเดียวกับ VQGAN
CELL-E มีตัวเลือกต่อไปนี้:
ckpt_path : ดำเนินการฝึกอบรม CELL-E 2 ก่อนหน้าต่อ โมเดลที่บันทึกไว้ด้วย state_dictvqgan_model_path : โมเดลอิมเมจโปรตีนที่บันทึกไว้ (พร้อม state_dict) สำหรับตัวเข้ารหัสอิมเมจโปรตีนvqgan_config_path : โมเดลอิมเมจโปรตีนที่บันทึกไว้ yamlcondition_model_path : แบบจำลองเงื่อนไข (นิวเคลียส) ที่บันทึกไว้ (พร้อม state_dict) สำหรับตัวเข้ารหัสภาพโปรตีนcondition_config_path : โมเดล yaml เงื่อนไข (นิวเคลียส) ที่บันทึกไว้num_images : 1 หากใช้ตัวเข้ารหัสภาพโปรตีนเท่านั้น และ 2 หากรวมตัวเข้ารหัสภาพสภาพimage_key : nucleus , target หรือ thresholddim : มิติของการฝังโมเดลภาษาnum_text_tokens : จำนวนโทเค็นทั้งหมดในรูปแบบภาษา (33 สำหรับ ESM-2)text_seq_len : จำนวนกรดอะมิโนทั้งหมดที่พิจารณาdepth : ความลึกของโมเดลหม้อแปลง ความลึกมักจะดีกว่าด้วยต้นทุนของ VRAMheads : จำนวนหัวที่ใช้ในการสนใจแบบหลายหัวdim_head : ขนาดของหัวความสนใจattn_dropout : อัตราความสนใจออกจากกลางคันในการฝึกอบรมff_dropout : อัตราการออกกลางคันของ Feed-Forward ในการฝึกอบรมloss_img_weight : การถ่วงน้ำหนักที่ใช้กับการสร้างภาพใหม่ น้ำหนักข้อความ = 1loss_text_weight : การถ่วงน้ำหนักที่ใช้กับการสร้างภาพตามเงื่อนไขใหม่stable : น้ำหนักมาตรฐาน (สำหรับเมื่อเกิดการไล่ระดับสีแบบระเบิด)learning_rate : อัตราการเรียนรู้สำหรับเครื่องมือเพิ่มประสิทธิภาพ Adammonitor : พารามิเตอร์ที่ใช้ในการบันทึกโมเดล โปรดอ้างอิงถึงเราหากคุณตัดสินใจที่จะใช้โค้ดของเราสำหรับส่วนใดส่วนหนึ่งของการวิจัยของคุณ
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}