แพ็คเกจนี้ประกอบด้วยโมเดล U-net ที่ได้รับการฝึกอบรมสำหรับการแบ่งส่วนปอด สำหรับตอนนี้มีทั้งหมด 4 รุ่นด้วยกัน:
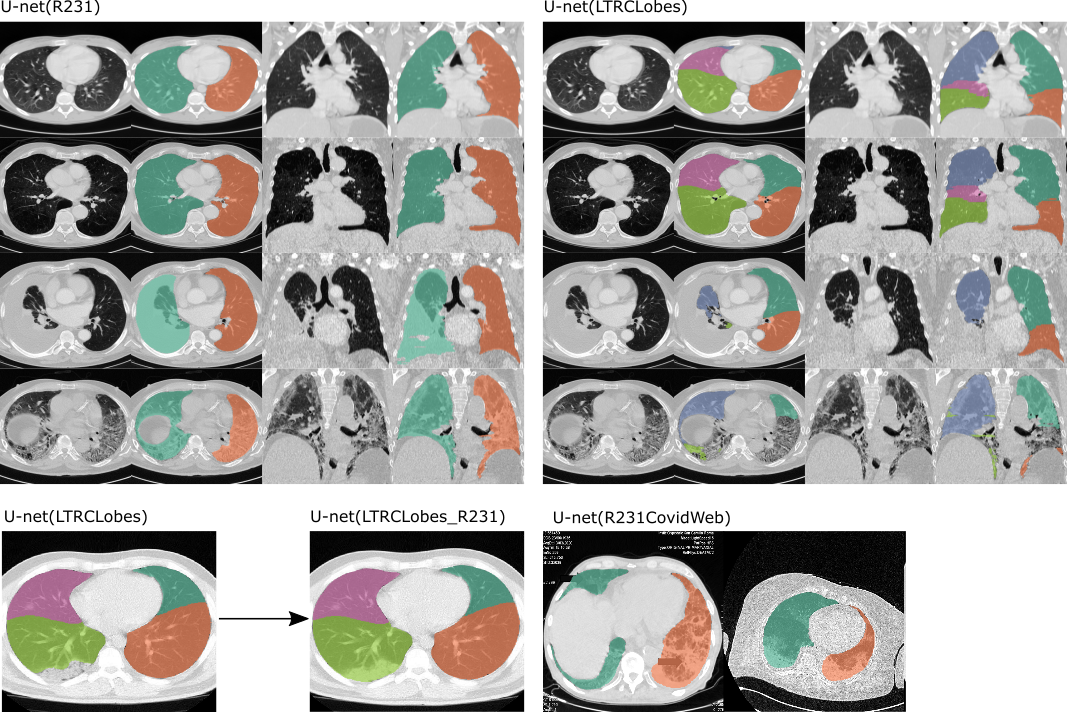
U-net(R231): โมเดลนี้ได้รับการฝึกอบรมเกี่ยวกับชุดข้อมูลขนาดใหญ่และหลากหลาย ซึ่งครอบคลุมความแปรปรวนทางการมองเห็นที่หลากหลาย แบบจำลองดำเนินการแบ่งส่วนในแต่ละชิ้น แยกปอดขวา-ซ้ายแยกกัน รวมถึงช่องลม เนื้องอก และน้ำไหลออก หลอดลมจะไม่รวมอยู่ในการแบ่งส่วนปอด https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRCLobes): โมเดลนี้ได้รับการฝึกฝนบนชุดย่อยของชุดข้อมูล LTRC แบบจำลองดำเนินการแบ่งส่วนของปอด-กลีบแต่ละส่วน แต่ให้ประสิทธิภาพที่จำกัด เมื่อมีโรคที่หนาแน่นหรือเมื่อมองไม่เห็นรอยแยกในทุกชิ้น
U-net(LTRCLobes_R231): สิ่งนี้จะรันโมเดล R231 และ LTRCLobes และหลอมรวมผลลัพธ์ ผลลบลวงจาก LTRCLobes จะถูกเติมด้วยการคาดการณ์ R231 และแมปกับป้ายกำกับเพื่อนบ้าน ผลบวกลวงจาก LTRCLobes จะถูกลบออก กระบวนการหลอมรวมมีความเข้มข้นในการคำนวณและอาจใช้เวลานานหลายนาทีต่อปริมาตร ทั้งนี้ขึ้นอยู่กับข้อมูลและผลลัพธ์
ยูเน็ต(R231CovidWeb)
ตัวอย่างของทั้งสองรุ่นที่นำมาใช้ ซ้าย: U-net (R231) จะแยกปอดซ้ายและขวา และรวมถึงบริเวณที่มีความหนาแน่นมาก เช่น ของเหลวไหลออกมา (แถวที่สาม) เนื้องอก หรือพังผืดรุนแรง (แถวที่สี่) ขวา: U-net(LTRLobes) จะแยกแยะระหว่างกลีบปอด แต่จะไม่รวมบริเวณที่หนาแน่นมาก LTRCLobes_R231 จะรวมผลลัพธ์ LTRCLobes และ R231 R231CovidWeb ได้รับการฝึกอบรมเกี่ยวกับข้อมูล COVID-19 เพิ่มเติม
ความหมายของเอาท์พุท :
ฉลากสองรุ่น (ซ้าย-ขวา):
1 = ปอดขวา
2 = ปอดซ้าย
แบบจำลองฉลากห้าแบบ (กลีบปอด):
1 = กลีบบนซ้าย
2 = กลีบล่างซ้าย
3 = กลีบบนขวา
4 = กลีบกลางขวา
5 = กลีบล่างขวา
หากต้องการการวิจัยที่น่าตื่นเต้นเพิ่มเติมเกี่ยวกับข้อมูล CT ปอด โปรดดูเว็บไซต์ของกลุ่มการวิจัยของเรา: https://www.cir.meduniwien.ac.at/research/lung/
หากคุณใช้รหัสนี้หรือหนึ่งในโมเดลที่ผ่านการฝึกอบรมในงานของคุณ โปรดดูที่:
Hofmanninger, J. , การอธิษฐาน, F. , Pan, J. และคณะ การแบ่งส่วนปอดอัตโนมัติในการถ่ายภาพตามปกติเป็นปัญหาด้านความหลากหลายของข้อมูลเป็นหลัก ไม่ใช่ปัญหาด้านระเบียบวิธี Eur Radiol Exp 4, 50 (2020) https://doi.org/10.1186/s41747-020-00173-2
บทความนี้ประกอบด้วยคำอธิบายโดยละเอียดของชุดข้อมูลที่ใช้ การประเมินแบบจำลอง U-net (R231) อย่างละเอียด และการเปรียบเทียบกับวิธีอ้างอิง
pip install lungmask
หรือ
pip install git+https://github.com/JoHof/lungmask
บน Windows อาจจำเป็นต้องติดตั้ง torch ล่วงหน้า: https://pytorch.org ขึ้นอยู่กับการตั้งค่าของคุณ
รันไทม์ระหว่างการอนุมานที่สนับสนุน CPU เท่านั้นและ GPU นั้นแตกต่างกันอย่างมาก การใช้ GPU การประมวลผลโวลุ่มใช้เวลาเพียงไม่กี่วินาที การใช้ CPU เพียงอย่างเดียวจะใช้เวลาหลายนาที หากต้องการใช้ GPU ตรวจสอบให้แน่ใจว่าการติดตั้งคบเพลิงของคุณรองรับ CUDA ในกรณีที่ข้อผิดพลาดหน่วยความจำ cuda ไม่เพียงพอ ให้ลดขนาดแบตช์เป็น 1 ด้วยอาร์กิวเมนต์ทางเลือก --batchsize 1
lungmask INPUT OUTPUT
หาก INPUT ชี้ไปที่ไฟล์ ไฟล์นั้นจะถูกประมวลผล หาก INPUT ชี้ไปที่ไดเร็กทอรี ไดเร็กทอรีนั้นจะถูกค้นหาสำหรับซีรี่ส์ DICOM ปริมาตรที่ใหญ่ที่สุดที่พบ (ในแง่ของจำนวนวอเซล) จะถูกนำมาใช้ในการคำนวณหน้ากากปอด OUTPUT คือชื่อไฟล์เอาต์พุต รองรับรูปแบบ ITK ทั้งหมด
เลือกรุ่น:
U-net(R231) จะถูกใช้เป็นค่าเริ่มต้น อย่างไรก็ตาม คุณสามารถระบุรุ่นอื่นได้ เช่น LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
สำหรับตัวเลือกเพิ่มเติม ให้พิมพ์:
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image จะต้องเป็นวัตถุ SimpleITK
โหลดโมเดลทางเลือกดังนี้:
inferer = LMInferer(modelname="R231CovidWeb")
หากต้องการใช้ความสามารถในการหลอมรวมแบบจำลองสำหรับ (เช่น LTRCLobes_R231) ให้ใช้:
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
ในเวอร์ชัน 0.2.9 รองรับอาร์เรย์ numpy เป็นโวลุ่มอินพุต โหมดนี้จะถือว่าอาร์เรย์อินพุต numpy มีรูปแบบต่อไปนี้สำหรับแต่ละแกน:
โมเดลนี้ใช้งานได้กับสไลซ์เต็มเท่านั้น ชิ้นที่จะแปรรูปต้องแสดงให้เห็นปอดทั้งหมด และปอดจะต้องมีเนื้อเยื่อล้อมรอบเพื่อที่จะแบ่งส่วน อย่างไรก็ตาม แบบจำลองนี้ค่อนข้างเสถียรสำหรับกรณีที่มีขอบเขตการมองเห็นแบบครอบตัด ตราบใดที่ปอดถูกล้อมรอบด้วยเนื้อเยื่อ
lungmask INPUT OUTPUT --modelname R231CovidWeb
โมเดล U-net (R231) ปกติทำงานได้ดีมากสำหรับการสแกน CT สแกนโควิด-19 อย่างไรก็ตาม คอลเลกชันของชิ้นส่วนและรายงานเคสจากเว็บมักจะถูกครอบตัด ใส่คำอธิบายประกอบ หรือเข้ารหัสในรูปแบบรูปภาพปกติ เพื่อให้สามารถประมาณค่าหน่วย hounsfield ดั้งเดิม (HU) ได้เท่านั้น ข้อมูลการฝึกอบรมของโมเดล U-net (R231CovidWeb) ได้รับการเสริมด้วยส่วนข้อมูลโควิด-19 ที่แมปกลับจากรูปแบบการถ่ายภาพปกติไปยัง HU ข้อมูลถูกรวบรวมและจัดเตรียมโดย MedSeg (http://medicalsegmentation.com/covid19/) แม้ว่า U-net ทั่วไป (R231) จะแสดงผลลัพธ์ที่ดีมากสำหรับภาพเหล่านี้ แต่อาจมีบางกรณีที่โมเดลนี้ให้การแบ่งส่วนที่ดีขึ้นเล็กน้อย โปรดทราบว่าคุณต้องแมปรูปภาพกลับไปที่ HU เมื่อใช้รูปภาพจากเว็บ โพสต์ในบล็อกนี้จะอธิบายวิธีที่คุณสามารถทำได้ หรือคุณสามารถตั้งค่าแท็ก --noHU ได้ 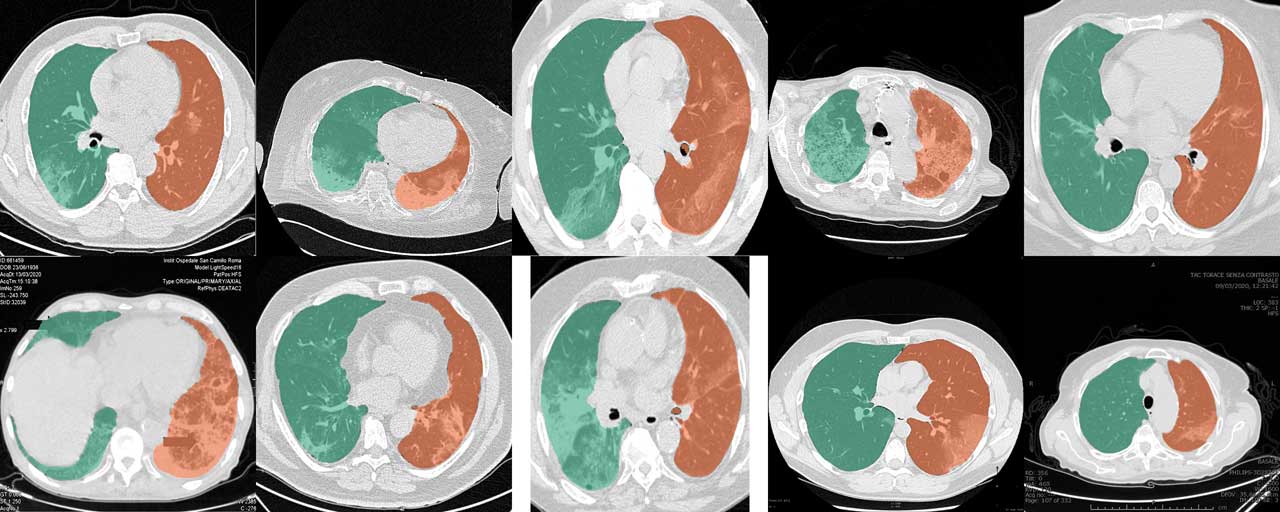
คุณลักษณะนี้มีเฉพาะในเวอร์ชันระหว่าง 0.2.5 ถึง 0.2.14 ตั้งแต่เวอร์ชัน 0.2.5 รองรับอิมเมจเหล่านี้ ใช้แท็ก --noHU หากคุณประมวลผลรูปภาพที่ไม่ได้เข้ารหัสใน HU โปรดทราบว่าแบบจำลองดังกล่าวได้รับการฝึกอบรมเกี่ยวกับการสแกน CT ที่เหมาะสมซึ่งเข้ารหัสใน HU ผลลัพธ์ของรูปภาพที่มีการครอบตัด ใส่คำอธิบายประกอบ สูงและมีการเลื่อนความเข้มต่ำมากอาจไม่น่าเชื่อถือมากนัก เมื่อใช้แท็ก --noHU จะสามารถประมวลผลได้เพียงชิ้นเดียวเท่านั้น