MedSegDiff กรอบงานที่ใช้โมเดลความน่าจะเป็นการแพร่กระจาย (DPM) สำหรับการแบ่งส่วนภาพทางการแพทย์ อัลกอริทึมได้รับการอธิบายอย่างละเอียดในเอกสารของเรา MedSegDiff: การแบ่งส่วนภาพทางการแพทย์ด้วยแบบจำลองความน่าจะเป็นการแพร่กระจาย และ MedSegDiff-V2: การแบ่งส่วนภาพทางการแพทย์ที่ใช้การแพร่กระจายด้วยหม้อแปลงไฟฟ้า
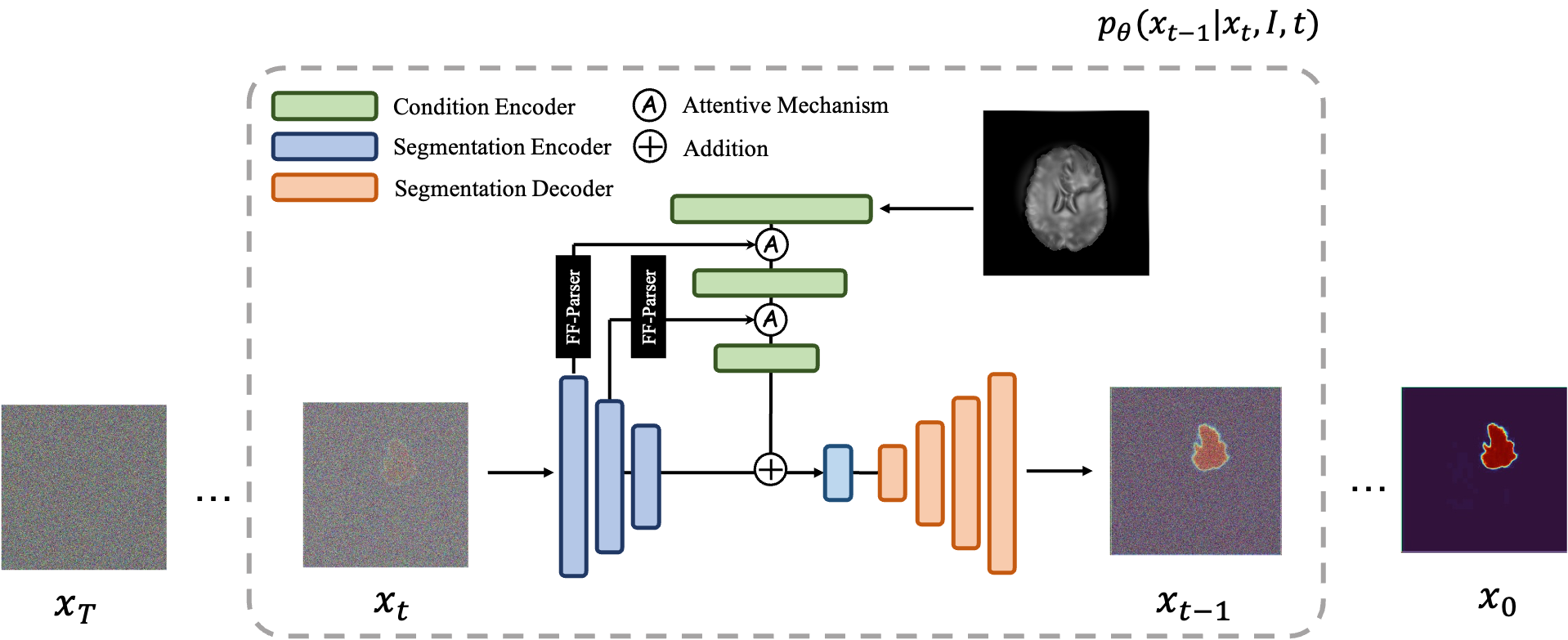
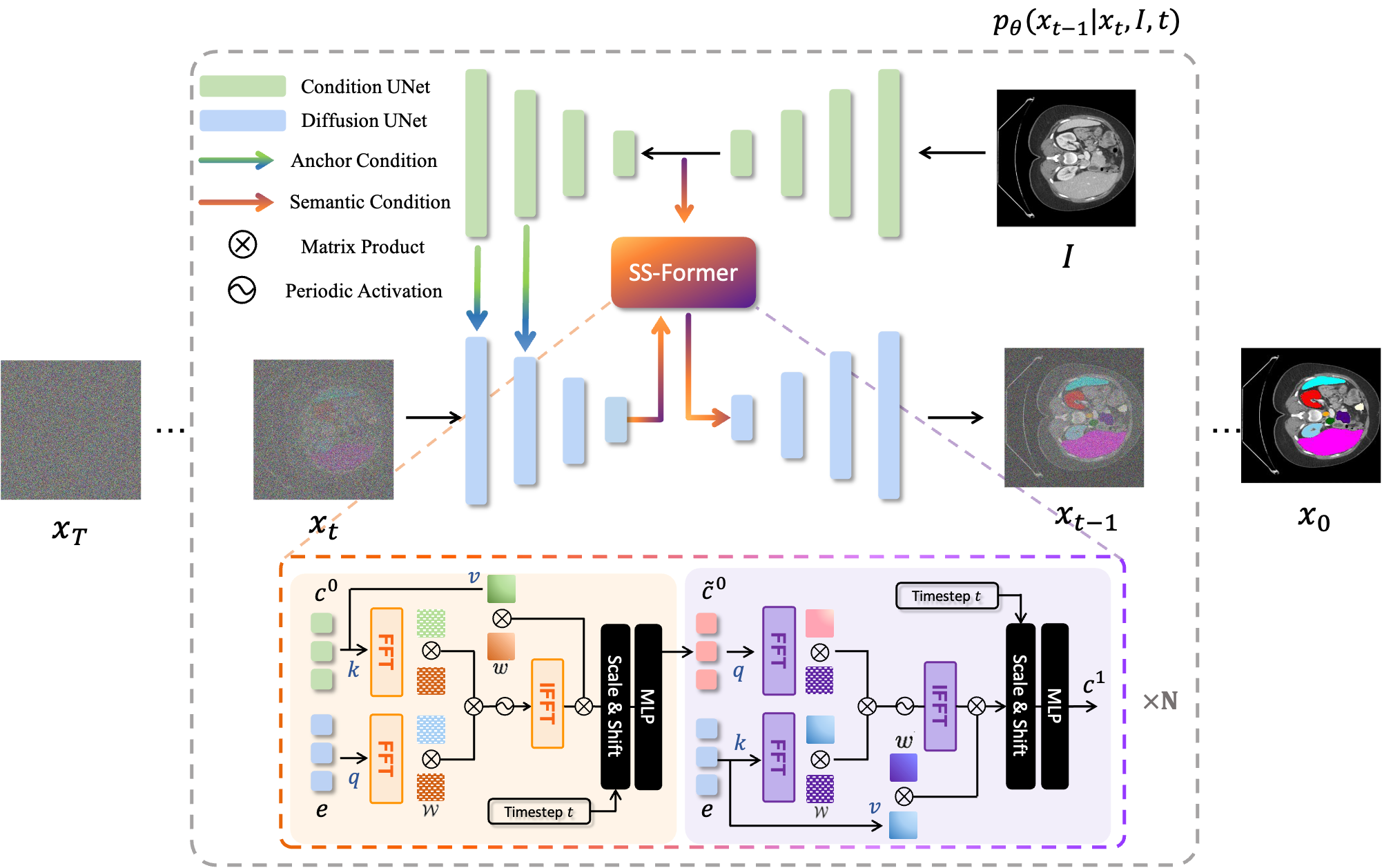
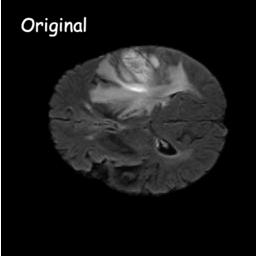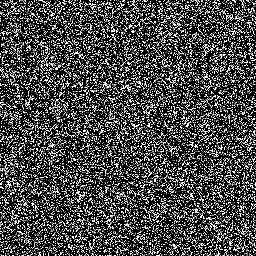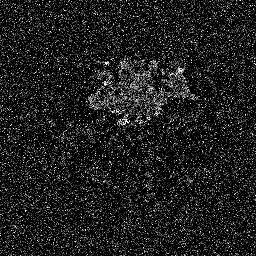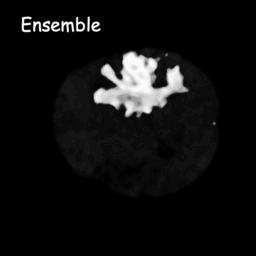 แบบจำลองการแพร่กระจายทำงานโดยการทำลายข้อมูลการฝึกด้วยการเพิ่มสัญญาณรบกวนแบบเกาส์เซียนอย่างต่อเนื่อง จากนั้นเรียนรู้ที่จะกู้คืนข้อมูลโดยการย้อนกลับกระบวนการรบกวนนี้ หลังการฝึกอบรม เราสามารถใช้แบบจำลองการแพร่กระจายเพื่อสร้างข้อมูลโดยเพียงแค่ส่งสัญญาณรบกวนตัวอย่างแบบสุ่มผ่านกระบวนการลดสัญญาณรบกวนที่เรียนรู้ ในโปรเจ็กต์นี้ เราขยายแนวคิดนี้ไปสู่การแบ่งส่วนภาพทางการแพทย์ เราใช้ภาพต้นฉบับเป็นเงื่อนไข และสร้างแผนที่การแบ่งส่วนหลายส่วนจากสัญญาณรบกวนแบบสุ่ม จากนั้นจึงประกอบภาพเหล่านั้นเข้าด้วยกันเพื่อให้ได้ผลลัพธ์สุดท้าย วิธีการนี้จะจับภาพความไม่แน่นอนทางการแพทย์และทำได้ดีกว่าวิธีการก่อนหน้านี้ในเกณฑ์มาตรฐานต่างๆ
แบบจำลองการแพร่กระจายทำงานโดยการทำลายข้อมูลการฝึกด้วยการเพิ่มสัญญาณรบกวนแบบเกาส์เซียนอย่างต่อเนื่อง จากนั้นเรียนรู้ที่จะกู้คืนข้อมูลโดยการย้อนกลับกระบวนการรบกวนนี้ หลังการฝึกอบรม เราสามารถใช้แบบจำลองการแพร่กระจายเพื่อสร้างข้อมูลโดยเพียงแค่ส่งสัญญาณรบกวนตัวอย่างแบบสุ่มผ่านกระบวนการลดสัญญาณรบกวนที่เรียนรู้ ในโปรเจ็กต์นี้ เราขยายแนวคิดนี้ไปสู่การแบ่งส่วนภาพทางการแพทย์ เราใช้ภาพต้นฉบับเป็นเงื่อนไข และสร้างแผนที่การแบ่งส่วนหลายส่วนจากสัญญาณรบกวนแบบสุ่ม จากนั้นจึงประกอบภาพเหล่านั้นเข้าด้วยกันเพื่อให้ได้ผลลัพธ์สุดท้าย วิธีการนี้จะจับภาพความไม่แน่นอนทางการแพทย์และทำได้ดีกว่าวิธีการก่อนหน้านี้ในเกณฑ์มาตรฐานต่างๆ
 |  |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver Truepython scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
สำหรับการฝึก ให้รัน: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
สำหรับการสุ่มตัวอย่าง ให้รัน: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
สำหรับการประเมิน ให้รัน python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
ตามค่าเริ่มต้น ตัวอย่างจะถูกบันทึกไว้ที่ ./results/
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
สำหรับการฝึก ให้รัน: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
สำหรับการสุ่มตัวอย่าง ให้รัน: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
-
การรัน MedSegDiff บนชุดข้อมูลอื่นๆ เป็นเรื่องง่าย เพียงเขียนไฟล์ตัวโหลดข้อมูลอื่นตาม ./guided_diffusion/isicloader.py หรือ . ./guided_diffusion/bratsloader.py ยินดีต้อนรับสู่ปัญหาที่เปิดอยู่หากคุณประสบปัญหาใด ๆ จะยินดีเป็นอย่างยิ่งหากคุณสามารถสนับสนุนส่วนขยายชุดข้อมูลของคุณได้ ภาพทางการแพทย์ต่างจากภาพธรรมชาติ โดยจะมีความแตกต่างกันอย่างมากขึ้นอยู่กับงานที่แตกต่างกัน การขยายวิธีการทั่วไปต้องใช้ความพยายามของทุกคน
หากต้องการฝึกโมเดลที่ดี เช่น MedSegDiff-B ในกระดาษ ให้ตั้งค่าไฮเปอร์พารามิเตอร์ของโมเดลเป็น:
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
ไฮเปอร์พารามิเตอร์การแพร่กระจายเป็น:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
เพื่อเร่งการสุ่มตัวอย่าง:
--diffusion_steps 50 --dpm_solver True
ทำงานบน GPU หลายตัว:
--multi-gpu 0,1,2 (for example)
การฝึกอบรมไฮเปอร์พารามิเตอร์เป็น:
--lr 5e-5 --batch_size 8
และตั้งค่า --num_ensemble 5 ในการสุ่มตัวอย่าง
การวิ่งประมาณ 100,000 ก้าวในการฝึกจะถูกรวมเข้ากับชุดข้อมูลส่วนใหญ่ โปรดทราบว่าแม้ว่าการสูญเสียจะไม่ลดลงในขั้นตอนต่อๆ ไปส่วนใหญ่ แต่คุณภาพของผลลัพธ์ก็ยังคงดีขึ้น กระบวนการดังกล่าวยังพบเห็นได้ในแอปพลิเคชัน DPM อื่นๆ เช่น การสร้างภาพ หวังว่าคนฉลาดสามารถบอกฉันได้ว่าทำไม?.
เร็วๆ นี้ ฉันจะเผยแพร่ประสิทธิภาพภายใต้ขนาดแบตช์ที่เล็กลง (เหมาะกับการทำงานบน GPU 24GB) เพื่อการเปรียบเทียบ
การตั้งค่าเพื่อปลดปล่อยศักยภาพทั้งหมดคือ (MedSegDiff++):
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
จากนั้นฝึกด้วยขนาดแบตช์ --batch_size 64 และสุ่มตัวอย่างด้วย ensemble number --num_ensemble 25
ยินดีต้อนรับสู่การมีส่วนร่วมกับ MedSegDiff เทคนิคใดๆ ที่สามารถปรับปรุงประสิทธิภาพหรือเพิ่มความเร็วของอัลกอริทึมได้นั้นเป็นสิ่งที่น่าชื่นชม ฉันกำลังเขียน MedSegDiff V2 โดยมุ่งเป้าไปที่วารสาร Nature/CVPR เช่นเดียวกับสิ่งพิมพ์ ฉันดีใจที่ได้ระบุรายชื่อผู้มีส่วนร่วมเป็นผู้เขียนร่วมของฉัน?
คัดลอกโค้ดจำนวนมากจาก openai/การปรับปรุงการแพร่กระจาย, WuJunde/ MrPrism, WuJunde/ DiagnosisFirst, LuChengTHU/dpm-solver, JuliaWolleb/Diffusion-based-Segmentation, hojonathanho/diffusion, guided-diffusion, bigmb/Unet-Segmentation-Pytorch-Nest -of-Unets, nnUnet, ลูซิเดรน/วิต-ไพทอร์ช
กรุณาอ้างอิง
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu