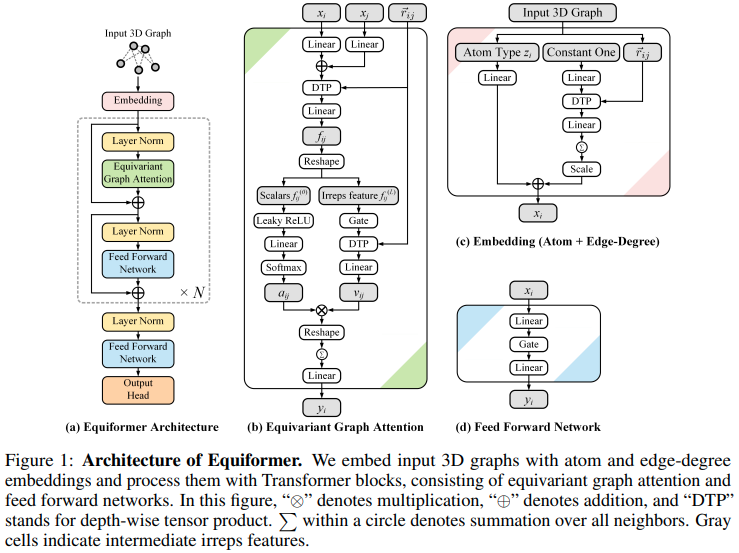
การใช้งาน Equiformer ซึ่งเป็นเครือข่ายความสนใจเทียบเท่า SE3/E3 ที่เข้าถึง SOTA ใหม่ และนำมาใช้โดย EquiFold (Prescient Design) สำหรับการพับโปรตีน
การออกแบบนี้ดูเหมือนจะสร้างจาก SE3 Transformers โดยแทนที่ความสนใจแบบ dot product ด้วย MLP Attention และข้อความที่ไม่ใช่เชิงเส้นที่ส่งผ่านจาก GATv2 นอกจากนี้ยังใช้ผลิตภัณฑ์เทนเซอร์เชิงลึกเพื่อประสิทธิภาพที่เพิ่มขึ้นอีกเล็กน้อย หากคุณคิดว่าฉันเข้าใจผิด โปรดส่งอีเมลถึงฉัน
อัปเดต: มีการพัฒนาใหม่ที่ทำให้การปรับขนาดจำนวนองศาสำหรับเครือข่ายที่เทียบเท่ากับ SE3 ดีขึ้นอย่างมาก! บทความนี้ตั้งข้อสังเกตเป็นครั้งแรกว่าโดยการจัดแนวการแสดงไปตามแกน z (หรือแกน y ด้วยรูปแบบอื่น) ฮาร์โมนิกทรงกลมจะกระจัดกระจาย วิธีนี้จะลบมิติ m f ออกจากสมการ บทความติดตามผลจาก Passaro และคณะ สังเกตเห็นว่าเมทริกซ์ Clebsch Gordan ก็มีน้อยลงเช่นกัน ซึ่งนำไปสู่การกำจัด m i และ l f พวกเขายังทำการเชื่อมต่อว่าปัญหาลดลงจาก SO(3) เป็น SO(2) หลังจากจัดตำแหน่งตัวแทนไปที่แกนเดียว Equiformer v2 (พื้นที่เก็บข้อมูลอย่างเป็นทางการ) ใช้ประโยชน์จากสิ่งนี้ในเฟรมเวิร์กที่คล้ายกับหม้อแปลงเพื่อเข้าถึง SOTA ใหม่
จะต้องทุ่มเททำงาน/สำรวจให้มากกว่านี้อย่างแน่นอน สำหรับตอนนี้ ฉันได้รวมเทคนิคจากรายงานสองฉบับแรกสำหรับ Equiformer v1 แล้ว บันทึกไว้สำหรับการแปลงเป็น SO(2) โดยสมบูรณ์
อัปเดต 2: ดูเหมือนว่าจะมี SOTA ใหม่โดยไม่มีการโต้ตอบระหว่างตัวแทนระดับที่สูงกว่า (กล่าวอีกนัยหนึ่งผลิตภัณฑ์เทนเซอร์ทั้งหมด / คณิตศาสตร์ clebsch gordan หายไป) GotenNet ซึ่งดูเหมือนว่าจะเป็นเวอร์ชัน Transformer ของ HEGNN
$ pip install equiformer-pytorch import torch
from equiformer_pytorch import Equiformer
model = Equiformer (
num_tokens = 24 ,
dim = ( 4 , 4 , 2 ), # dimensions per type, ascending, length must match number of degrees (num_degrees)
dim_head = ( 4 , 4 , 4 ), # dimension per attention head
heads = ( 2 , 2 , 2 ), # number of attention heads
num_linear_attn_heads = 0 , # number of global linear attention heads, can see all the neighbors
num_degrees = 3 , # number of degrees
depth = 4 , # depth of equivariant transformer
attend_self = True , # attending to self or not
reduce_dim_out = True , # whether to reduce out to dimension of 1, say for predicting new coordinates for type 1 features
l2_dist_attention = False # set to False to try out MLP attention
). cuda ()
feats = torch . randint ( 0 , 24 , ( 1 , 128 )). cuda ()
coors = torch . randn ( 1 , 128 , 3 ). cuda ()
mask = torch . ones ( 1 , 128 ). bool (). cuda ()
out = model ( feats , coors , mask ) # (1, 128)
out . type0 # invariant type 0 - (1, 128)
out . type1 # equivariant type 1 - (1, 128, 3)พื้นที่เก็บข้อมูลนี้ยังรวมถึงวิธีแยกการใช้หน่วยความจำจากความลึกโดยใช้เครือข่ายแบบพลิกกลับได้ กล่าวอีกนัยหนึ่ง หากคุณเพิ่มความลึก ต้นทุนหน่วยความจำจะคงที่เมื่อใช้บล็อกหม้อแปลงอีคฟอร์เมอร์หนึ่งบล็อก (ความสนใจและฟีดไปข้างหน้า)
import torch
from equiformer_pytorch import Equiformer
model = Equiformer (
num_tokens = 24 ,
dim = ( 4 , 4 , 2 ),
dim_head = ( 4 , 4 , 4 ),
heads = ( 2 , 2 , 2 ),
num_degrees = 3 ,
depth = 48 , # depth of 48 - just to show that it runs - in reality, seems to be quite unstable at higher depths, so architecture stil needs more work
reversible = True , # just set this to True to use https://arxiv.org/abs/1707.04585
). cuda ()
feats = torch . randint ( 0 , 24 , ( 1 , 128 )). cuda ()
coors = torch . randn ( 1 , 128 , 3 ). cuda ()
mask = torch . ones ( 1 , 128 ). bool (). cuda ()
out = model ( feats , coors , mask )
out . type0 . sum (). backward ()มีขอบ เช่น พันธะอะตอม
import torch
from equiformer_pytorch import Equiformer
model = Equiformer (
num_tokens = 28 ,
dim = 64 ,
num_edge_tokens = 4 , # number of edge type, say 4 bond types
edge_dim = 16 , # dimension of edge embedding
depth = 2 ,
input_degrees = 1 ,
num_degrees = 3 ,
reduce_dim_out = True
)
atoms = torch . randint ( 0 , 28 , ( 2 , 32 ))
bonds = torch . randint ( 0 , 4 , ( 2 , 32 , 32 ))
coors = torch . randn ( 2 , 32 , 3 )
mask = torch . ones ( 2 , 32 ). bool ()
out = model ( atoms , coors , mask , edges = bonds )
out . type0 # (2, 32)
out . type1 # (2, 32, 3)ด้วยเมทริกซ์ที่อยู่ติดกัน
import torch
from equiformer_pytorch import Equiformer
model = Equiformer (
dim = 32 ,
heads = 8 ,
depth = 1 ,
dim_head = 64 ,
num_degrees = 2 ,
valid_radius = 10 ,
reduce_dim_out = True ,
attend_sparse_neighbors = True , # this must be set to true, in which case it will assert that you pass in the adjacency matrix
num_neighbors = 0 , # if you set this to 0, it will only consider the connected neighbors as defined by the adjacency matrix. but if you set a value greater than 0, it will continue to fetch the closest points up to this many, excluding the ones already specified by the adjacency matrix
num_adj_degrees_embed = 2 , # this will derive the second degree connections and embed it correctly
max_sparse_neighbors = 8 # you can cap the number of neighbors, sampled from within your sparse set of neighbors as defined by the adjacency matrix, if specified
)
feats = torch . randn ( 1 , 128 , 32 )
coors = torch . randn ( 1 , 128 , 3 )
mask = torch . ones ( 1 , 128 ). bool ()
# placeholder adjacency matrix
# naively assuming the sequence is one long chain (128, 128)
i = torch . arange ( 128 )
adj_mat = ( i [:, None ] <= ( i [ None , :] + 1 )) & ( i [:, None ] >= ( i [ None , :] - 1 ))
out = model ( feats , coors , mask , adj_mat = adj_mat )
out . type0 # (1, 128)
out . type1 # (1, 128, 3) การทดสอบความเท่าเทียมกัน ฯลฯ
$ python setup.py test ขั้นแรกให้ติดตั้ง sidechainnet
$ pip install sidechainnetจากนั้นรันงาน denoising โปรตีนแบ็คโบน
$ python denoise.pyย้าย xi และ xj แยกโปรเจ็กต์และตรรกะรวมลงในคลาส Conv
ย้ายการผลิตคีย์ / ค่าที่โต้ตอบด้วยตนเองไปยัง Conv แก้ไขไม่รวมใน Conv ด้วยการโต้ตอบด้วยตนเอง
ใช้วิธีการไร้เดียงสาเพื่อแยกการมีส่วนร่วมจากระดับอินพุตสำหรับ DTP
สำหรับความสนใจของผลิตภัณฑ์ดอทในประเภทที่สูงกว่า ให้ลองใช้ระยะทางแบบยูคลิด
พิจารณาชั้นความสนใจของเพื่อนบ้านทั้งหมดสำหรับประเภท 0 โดยใช้ความสนใจเชิงเส้น
รวมการค้นพบใหม่จากกระดาษช่องทรงกลมตามด้วยกระดาษ so(3) -> so(2) ซึ่งจะลดการคำนวณจาก O(L^6) -> O(L^3)!
@article { Liao2022EquiformerEG ,
title = { Equiformer: Equivariant Graph Attention Transformer for 3D Atomistic Graphs } ,
author = { Yi Liao and Tess E. Smidt } ,
journal = { ArXiv } ,
year = { 2022 } ,
volume = { abs/2206.11990 }
} @article { Lee2022.10.07.511322 ,
author = { Lee, Jae Hyeon and Yadollahpour, Payman and Watkins, Andrew and Frey, Nathan C. and Leaver-Fay, Andrew and Ra, Stephen and Cho, Kyunghyun and Gligorijevic, Vladimir and Regev, Aviv and Bonneau, Richard } ,
title = { EquiFold: Protein Structure Prediction with a Novel Coarse-Grained Structure Representation } ,
elocation-id = { 2022.10.07.511322 } ,
year = { 2022 } ,
doi = { 10.1101/2022.10.07.511322 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2022/10/08/2022.10.07.511322 } ,
eprint = { https://www.biorxiv.org/content/early/2022/10/08/2022.10.07.511322.full.pdf } ,
journal = { bioRxiv }
} @article { Shazeer2019FastTD ,
title = { Fast Transformer Decoding: One Write-Head is All You Need } ,
author = { Noam M. Shazeer } ,
journal = { ArXiv } ,
year = { 2019 } ,
volume = { abs/1911.02150 }
} @misc { ding2021cogview ,
title = { CogView: Mastering Text-to-Image Generation via Transformers } ,
author = { Ming Ding and Zhuoyi Yang and Wenyi Hong and Wendi Zheng and Chang Zhou and Da Yin and Junyang Lin and Xu Zou and Zhou Shao and Hongxia Yang and Jie Tang } ,
year = { 2021 } ,
eprint = { 2105.13290 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CV }
} @inproceedings { Kim2020TheLC ,
title = { The Lipschitz Constant of Self-Attention } ,
author = { Hyunjik Kim and George Papamakarios and Andriy Mnih } ,
booktitle = { International Conference on Machine Learning } ,
year = { 2020 }
} @article { Zitnick2022SphericalCF ,
title = { Spherical Channels for Modeling Atomic Interactions } ,
author = { C. Lawrence Zitnick and Abhishek Das and Adeesh Kolluru and Janice Lan and Muhammed Shuaibi and Anuroop Sriram and Zachary W. Ulissi and Brandon C. Wood } ,
journal = { ArXiv } ,
year = { 2022 } ,
volume = { abs/2206.14331 }
} @article { Passaro2023ReducingSC ,
title = { Reducing SO(3) Convolutions to SO(2) for Efficient Equivariant GNNs } ,
author = { Saro Passaro and C. Lawrence Zitnick } ,
journal = { ArXiv } ,
year = { 2023 } ,
volume = { abs/2302.03655 }
} @inproceedings { Gomez2017TheRR ,
title = { The Reversible Residual Network: Backpropagation Without Storing Activations } ,
author = { Aidan N. Gomez and Mengye Ren and Raquel Urtasun and Roger Baker Grosse } ,
booktitle = { NIPS } ,
year = { 2017 }
} @article { Bondarenko2023QuantizableTR ,
title = { Quantizable Transformers: Removing Outliers by Helping Attention Heads Do Nothing } ,
author = { Yelysei Bondarenko and Markus Nagel and Tijmen Blankevoort } ,
journal = { ArXiv } ,
year = { 2023 } ,
volume = { abs/2306.12929 } ,
url = { https://api.semanticscholar.org/CorpusID:259224568 }
} @inproceedings { Arora2023ZoologyMA ,
title = { Zoology: Measuring and Improving Recall in Efficient Language Models } ,
author = { Simran Arora and Sabri Eyuboglu and Aman Timalsina and Isys Johnson and Michael Poli and James Zou and Atri Rudra and Christopher R'e } ,
year = { 2023 } ,
url = { https://api.semanticscholar.org/CorpusID:266149332 }
}