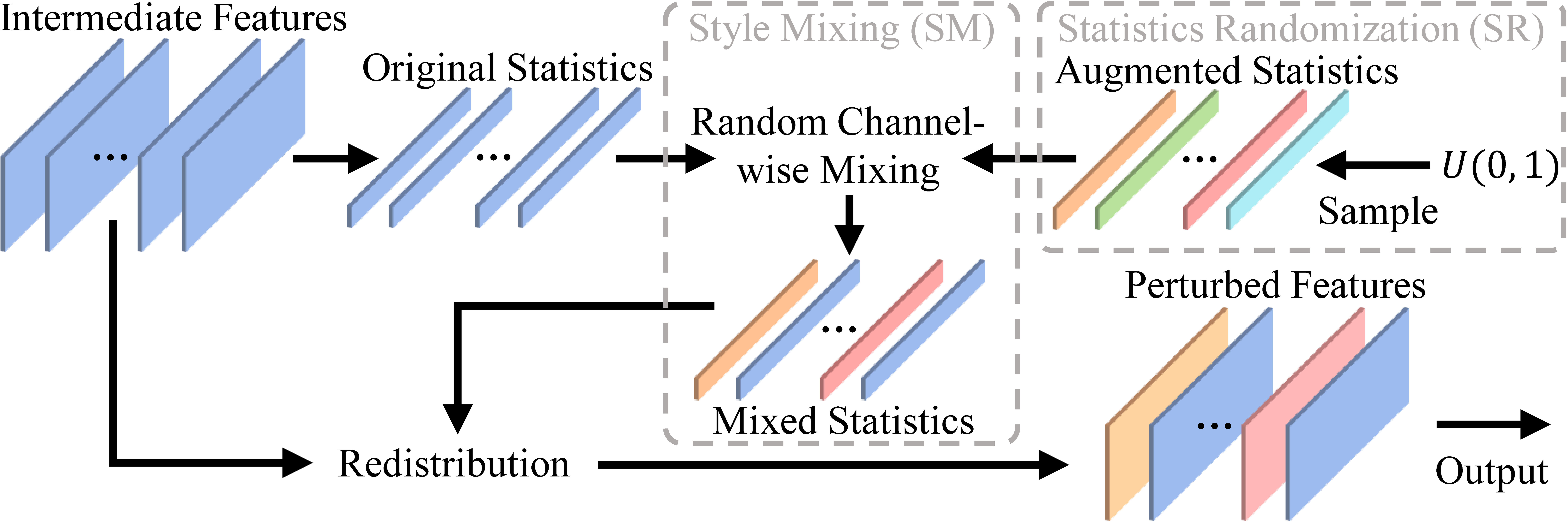
นี่คือการใช้งาน Pytorch อย่างเป็นทางการของ MICCAI 2023 กระดาษ "สมบัติในการกระจาย: การสุ่มโดเมนตามโดเมนหลายแหล่งโดเมนโดยทั่วไปสำหรับการแบ่งส่วนภาพการแพทย์ 2D" ในบทความนี้เราเสนอวิธีการวางนัยทั่วไปของโดเมนหลายแหล่งที่เรียกว่าสมบัติในการกระจาย (TRID) เพื่อสร้างพื้นที่การค้นหาที่ไม่เคยเกิดขึ้นมาก่อนสำหรับการสุ่มโดเมนคุณสมบัติพื้นที่คุณสมบัติ
Python 3.7
Pytorch 1.8.0
git clone https://github.com/Chen-Ziyang/TriD.git
cd TriD/TriD-master
การแบ่งส่วน OD/OC
การแบ่งส่วนต่อมลูกหมาก
เราใช้สถานการณ์โดยใช้ binrushed (โดเมนเป้าหมาย) และชุดข้อมูลอีกสี่ชุด (โดเมนแหล่งที่มา) เป็นตัวอย่าง
cd OPTIC
# Training
CUDA_VISIBLE_DEVICES=0 python main.py --mode train_DG --mixstyle_layers layer1 layer2 --random_type TriD --Target_Dataset BinRushed --Source_Dataset Magrabia REFUGE ORIGA Drishti_GS
# Test
CUDA_VISIBLE_DEVICES=0 python main.py --mode single_test --load_time TIME_OF_MODEL --Target_Dataset BinRushed
เราใช้สถานการณ์โดยใช้ BMC (โดเมนเป้าหมาย) และชุดข้อมูลอีกห้าชุด (โดเมนต้นทาง) เป็นตัวอย่าง
cd PROSTATE
# Training
CUDA_VISIBLE_DEVICES=0 python main.py --mode train_DG --mixstyle_layers layer1 layer2 --random_type TriD --Target_Dataset BMC --Source_Dataset BIDMC HK I2CVB RUNMC UCL
# Test
CUDA_VISIBLE_DEVICES=0 python main.py --mode single_test --load_time TIME_OF_MODEL --Target_Dataset BMC
ส่วนหนึ่งของรหัสได้รับการแก้ไขจากการใช้ Pytorch ของ DCR
หากคุณพบว่า repo นี้มีประโยชน์สำหรับการวิจัยของคุณโปรดพิจารณาอ้างถึงบทความดังนี้:
@inproceedings{chen2023treasure,
title={Treasure in distribution: a domain randomization based multi-source domain generalization for 2d medical image segmentation},
author={Chen, Ziyang and Pan, Yongsheng and Ye, Yiwen and Cui, Hengfei and Xia, Yong},
booktitle={International Conference on Medical Image Computing and Computer-Assisted Intervention},
pages={89--99},
year={2023},
organization={Springer}
}