เรายินดีที่จะประกาศว่าขนาดดัชนีบนดิสก์ลดลง 8 เท่าและในหน่วยความจำ 4 ครั้งเนื่องจากย้ายไปยังดัชนี FM เพียงประเภทเดียว (2 บิต 64 แทนที่จะเป็น 2 บิต 64 และ 8bit.32) และการบีบอัด 8x ของอาร์เรย์ต่อท้าย ตัวอย่างเช่นสำหรับจีโนมมนุษย์ขนาดดัชนีบนดิสก์จะลดลงเหลือ ~ 10GB จาก ~ 80GB และรอยเท้าหน่วยความจำลดลงเหลือ ~ 10GB จาก ~ 40GB มีการลดลงอย่างมากในเวลาดัชนี IO เนื่องจากการลดลงและแทบจะไม่ส่งผลกระทบต่อประสิทธิภาพการทำงานในการทำแผนที่อ่าน เนื่องจากการเปลี่ยนแปลงโครงสร้างดัชนีนี้ (ใน commit #4B59796, 10 ตุลาคม 2020) คุณจะต้องสร้างดัชนีใหม่
เพิ่ม MC Flag ในไฟล์ SAM เอาต์พุตใน commit A591E22 เอาต์พุตควรตรงกับ BWA-MEM ดั้งเดิมเวอร์ชัน 0.7.17
ในฐานะที่กระทำ E0AC59E เรามี safestringLib git submodule ในการรับให้ใช้ -recursive ในขณะที่โคลนหรือใช้ "Git Submodule Init" และ "Git Submodule Update" ในที่เก็บโคลนแล้ว (ดูรายละเอียดเพิ่มเติมด้านล่าง)
# ใช้ Binaries Precompiled (แนะนำ) Curl -l https://github.com/bwa-mem2/bwa-mem2/releases/download/v2.2.1/bwa-mem2-2.2.1_x64-linux.tar.bz2.bz2 - tar jxf - BWA-MEM2-2.2.1_X64-LINUX/BWA-MEM2 INDEX REF.FA bwa-mem2-2.2.1_x64-linux/bwa-mem2 mem ref.fa read1.fq read2.fq> out.sam# รวบรวมจากแหล่งที่มา (ไม่แนะนำสำหรับผู้ใช้ทั่วไป)# รับ clone sourcegit github.com/bwa-mem2/bwa-mem2cd bwa-mem2# orgit โคลน https://github.com/bwa-mem2/bwa-mem2cd bwa-mem2 git submodule init Git Submodule Update# Compile and Runmake ./BWA-MEM2
เครื่องมือ BWA-MEM2 เป็นอัลกอริทึม BWA-MEM รุ่นต่อไปใน BWA มันสร้างการจัดตำแหน่งเหมือนกับ BWA และ ~ 1.3-3.1x เร็วขึ้นขึ้นอยู่กับกรณีการใช้งานชุดข้อมูลและเครื่องที่กำลังทำงาน
BWA ดั้งเดิมได้รับการพัฒนาโดย Heng Li (@LH3) การเพิ่มประสิทธิภาพการทำงานใน BWA-MEM2 นั้นส่วนใหญ่ทำโดย Vasimuddin MD (@Yuk12) และ Sanchit Misra (@Sanchit-Misra) จาก Lab คอมพิวเตอร์ขนาน, Intel BWA-MEM2 มีการแจกจ่ายภายใต้ใบอนุญาต MIT
สำหรับผู้ใช้ทั่วไปขอแนะนำให้ใช้ไบนารีที่รวบรวมไว้ล่วงหน้าจากหน้ารุ่น ไบนารีเหล่านี้ถูกรวบรวมด้วยคอมไพเลอร์ Intel และทำงานได้เร็วกว่าไบนารีคอมไพล์ GCC ไบนารีที่คอมไพล์ล่วงหน้ายังสนับสนุนการจัดส่ง CPU ทางอ้อม ไบนารี bwa-mem2 สามารถเลือกการใช้งานที่มีประสิทธิภาพมากที่สุดโดยอัตโนมัติตามชุดคำสั่ง SIMD ที่มีอยู่ในเครื่องที่ทำงาน ไบนารีที่คอมไพล์ล่วงหน้าถูกสร้างขึ้นบนเครื่อง CentOS7 โดยใช้บรรทัดคำสั่งต่อไปนี้:
ทำ cxx = icpc multi
การใช้งานเหมือนกับเครื่องมือ BWA MEM ดั้งเดิม นี่คือ synopsys สั้น ๆ Run ./BWA-MEM2 สำหรับคำสั่งที่มีอยู่
# การจัดทำดัชนีลำดับการอ้างอิง (ต้องใช้หน่วยความจำ 28N GB โดยที่ n คือขนาดของลำดับการอ้างอิง) ../ ดัชนี BWA-MEM2 [-p คำนำหน้า] <in.fasta> โดยที่ <in.fasta> เป็นพา ธ ไปยังไฟล์อ้างอิงลำดับ Fasta และ <คำนำหน้า> เป็นคำนำหน้าของชื่อของไฟล์ที่เก็บดัชนีผลลัพธ์ ค่าเริ่มต้นคือ in.fasta. # การแมป # run "./bwa-mem2 mem" เพื่อรับตัวเลือกทั้งหมด/bwa-mem2 mem -t <num_threads> <คำนำหน้า> <reads.fq/fa >> out.sam โดยที่ <คำนำหน้า> เป็นคำนำหน้าที่ระบุเมื่อสร้างดัชนีหรือพา ธ ไปยังไฟล์ Fasta อ้างอิงในกรณีที่ไม่มีคำนำหน้า
ชุดข้อมูล:
จีโนมอ้างอิง: human_g1k_v37.fasta
| นามแฝง | แหล่งข้อมูล | จำนวนการอ่าน | อ่านความยาว |
|---|---|---|---|
| D1 | สถาบันกว้าง | 2 x 2.5m bp | 151bp |
| D2 | SRA: SRR77733443 | 2 x 2.5m bp | 151bp |
| D3 | SRA: SRR9932168 | 2 x 2.5m bp | 151bp |
| D4 | SRA: SRX6999918 | 2 x 2.5m bp | 151bp |
รายละเอียดของเครื่อง:
โปรเซสเซอร์: Intel (R) Xeon (R) 8280 CPU @ 2.70GHz
OS: Centos Linux Release 7.6.1810
หน่วยความจำ: 100GB
เราทำตามขั้นตอนด้านล่างเพื่อรวบรวมผลการปฏิบัติงาน:
A. ข้อมูลดาวน์โหลดข้อมูล:
ดาวน์โหลด SRA Toolkit จาก https://trace.ncbi.nlm.nih.gov/traces/sra/sra.cgi?view=software#header-global
tar xfzv sratoolkit.2.10.5-centos_linux64.tar.gz
ดาวน์โหลด d2: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump-split-files srr77733443
ดาวน์โหลด d3: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump-split-files srr9932168
ดาวน์โหลด d4: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump--split-files srx6999918
B. ขั้นตอนการจัดตำแหน่ง:
git clone https://github.com/bwa-mem2/bwa-mem2.git
CD BWA-MEM2
make CXX=icpc (ใช้คอมไพเลอร์ Intel C/C ++)
หรือ make (ใช้คอมไพเลอร์ GCC)
./bwa-mem2 index <ref.fa>
./bwa-mem2 mem [-t <#threads>] <ref.fa> <in_1.fastq> [<in_2.fastq>]> <output.sam>
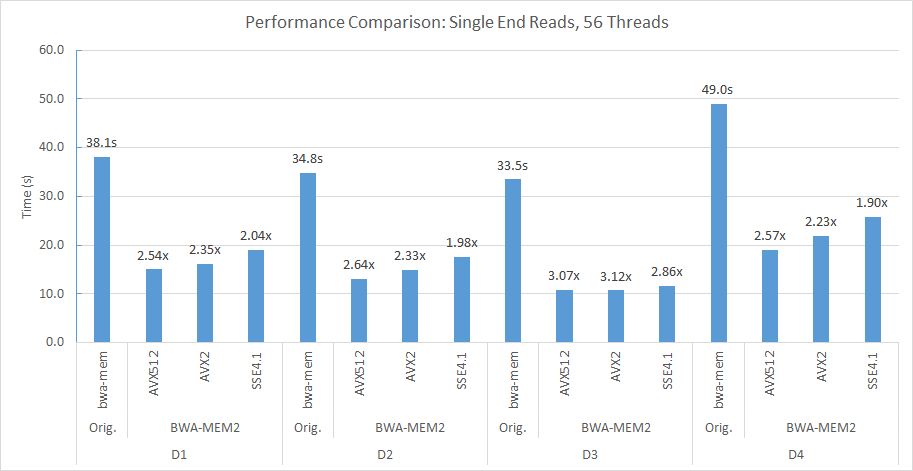
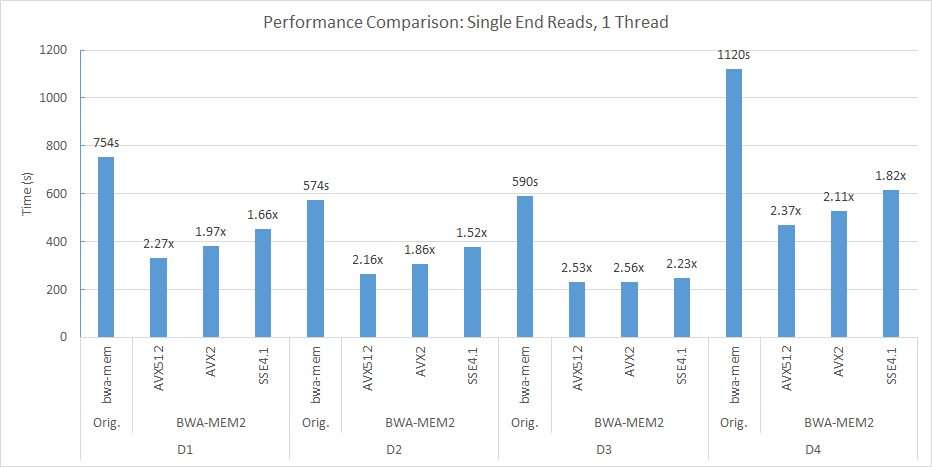
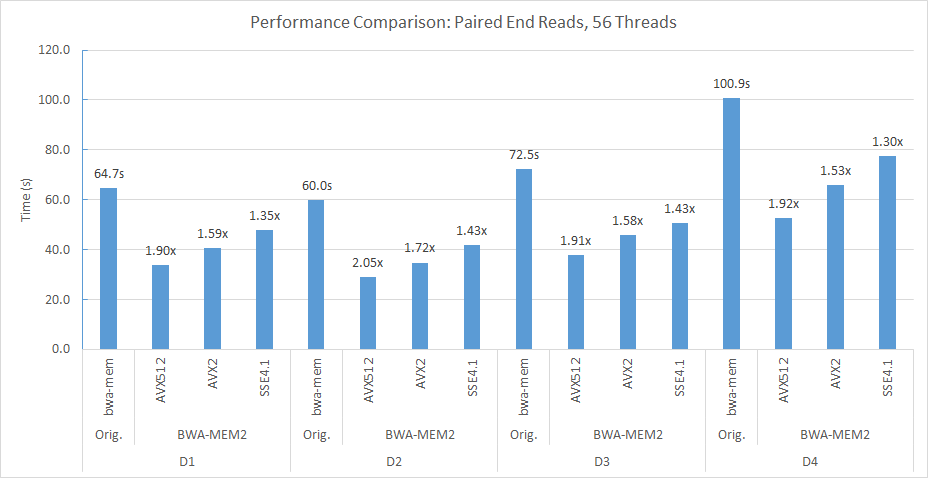
ตัวอย่างเช่นในซ็อกเก็ตคู่ของเรา (56 เธรดแต่ละอัน) และโหนดการคำนวณ NUMA สองครั้งเราใช้บรรทัดคำสั่งต่อไปนี้เพื่อจัดตำแหน่ง D2 เป็น Human_G1K_V37.Fasta อ้างอิงจีโนม
numactl -m 0 -C 0-27,56-83 ./bwa-mem2 index human_g1k_v37.fasta numactl -m 0 -C 0-27,56-83 ./bwa-mem2 mem -t 56 human_g1k_v37.fasta SRR7733443_1.fastq SRR7733443_2.fastq > d2_align.sam

BWA-MEM2-LISA เป็นรุ่นเร่งความเร็วของ BWA-MEM2 ซึ่งเราใช้ดัชนีที่เรียนรู้กับขั้นตอนการเพาะ สาขา BWA-MEM2-LISA มีซอร์สโค้ดของการใช้งาน ต่อไปนี้เป็นคุณสมบัติของ BWA-MEM2-LISA:
เอาต์พุตที่แน่นอนเช่น BWA-MEM2
สายคำสั่งทั้งหมดสำหรับการสร้างดัชนีและการแมปอ่านจะเหมือนกับ BWA-MEM2
BWA-MEM2-LISA เร่งระยะการเพาะ (หนึ่งในคอขวดที่สำคัญใน BWA-MEM2) สูงถึง 4.5x เมื่อเทียบกับ BWA-MEM2
รอยเท้าหน่วยความจำของดัชนี BWA-MEM2-LISA คือ ~ 120GB สำหรับจีโนมมนุษย์
รหัสมีอยู่ในสาขา BWA-MEM2-LISA: https://github.com/bwa-mem2/bwa-mem2/tree/bwa-mem2-lisa
สาขา ERT ของที่เก็บ BWA-MEM2 มี codebase ของการเร่งความเร็วตาม Radix Tree ที่เป็น enuerated ของ BWA-MEM2 รหัส ERT ถูกสร้างขึ้นที่ด้านบนของ BWA-MEM2 (ขอบคุณการทำงานอย่างหนักโดย @Arun-Sub) ต่อไปนี้เป็นไฮไลท์ของเครื่องมือ BWA-MEM2 ที่ใช้ ERT:
เอาต์พุตที่แน่นอนเช่นเดียวกับ BWA-MEM (2)
เครื่องมือนี้มีธงเพิ่มเติมสองหมายเพื่อเปิดใช้งานการใช้โซลูชัน ERT (สำหรับการสร้างดัชนีและการแมป) มิฉะนั้นจะทำงานในโหมดวานิลลา BWA-MEM2
ใช้ 1 แฟล็กเพิ่มเติมเพื่อสร้างดัชนี ERT (แตกต่างจากดัชนี BWA-MEM2) และ 1 ธงเพิ่มเติมสำหรับการใช้ดัชนี ERT นั้น (โปรดดู readMe ของสาขา ERT)
โซลูชัน ERT นั้นเร็วขึ้น 10% -30% (ทดสอบในการกำหนดค่าเครื่องด้านบน) เมื่อเปรียบเทียบกับวานิลลา BWA -MEM2 -ผู้ใช้ได้รับคำแนะนำให้ใช้ตัวเลือก -K 1000000 เพื่อดูการเร่งความเร็ว
การพิมพ์เท้าหน่วยความจำของดัชนี ERT คือ ~ 60GB
รหัสมีอยู่ในสาขา ERT: https://github.com/bwa-mem2/bwa-mem2/tree/ert
Vasimuddin MD, Sanchit Misra, Heng Li, Srinivas Aluru การเร่งความเร็วด้วยสถาปัตยกรรมที่มีประสิทธิภาพของ BWA-MEM สำหรับระบบมัลติคอร์ IEEE Parallel และ Distributed Processing Symposium (IPDPS), 2019. 10.1109/IPDPS.2019.00041