นักวิจัยจากสถาบันเทคโนโลยีแมสซาชูเซตส์ (MIT) ได้เปิดตัวแบบจำลองปัญญาประดิษฐ์แบบโอเพ่นซอร์สอันทรงพลังที่เรียกว่า Boltz-1 ซึ่งมีความแม่นยำเท่ากับ AlphaFold3 ของ Google DeepMind ในการทำนายโครงสร้างชีวโมเลกุล ความก้าวหน้าครั้งนี้คาดว่าจะช่วยเร่งกระบวนการวิจัยชีวการแพทย์และการพัฒนายาได้อย่างมีนัยสำคัญ โดยถือเป็นเครื่องมือที่สะดวกและมีประสิทธิภาพมากขึ้นสำหรับนักวิจัยทางวิทยาศาสตร์ทั่วโลก ลักษณะโอเพ่นซอร์สโดยสมบูรณ์ของ Boltz-1 ยังส่งเสริมการทำงานร่วมกันในวงกว้างและการมีส่วนร่วมของชุมชน ซึ่งจะช่วยพัฒนาความก้าวหน้าของการค้นพบทางวิทยาศาสตร์
นักวิทยาศาสตร์จากสถาบันเทคโนโลยีแมสซาชูเซตส์ (MIT) เพิ่งเปิดตัวโมเดลปัญญาประดิษฐ์แบบโอเพ่นซอร์สอันทรงพลังที่เรียกว่า Boltz-1 นวัตกรรมนี้สัญญาว่าจะเร่งการวิจัยทางชีวการแพทย์และการพัฒนายาได้อย่างมีนัยสำคัญ
Boltz-1 เป็นโมเดลโอเพ่นซอร์สเต็มรูปแบบรุ่นแรกที่สามารถไปถึงระดับขั้นสูงเช่นเดียวกับ AlphaFold3 ของ Google DeepMind ในการทำนายโครงสร้างชีวโมเลกุล แบบจำลองนี้ได้รับการพัฒนาโดยทีมงานจาก Jameel Machine Learning Health Clinic ของ MIT ซึ่งนำโดยนักศึกษาระดับบัณฑิตศึกษา Jeremy Wolvind และ Gabriel Corso และรวมถึงนักวิจัยของ MIT Salo Passaro และศาสตราจารย์วิศวกรรมไฟฟ้า พร้อมด้วยอาจารย์ด้านวิทยาการคอมพิวเตอร์ Regina Bardsley และ Tommy Akara
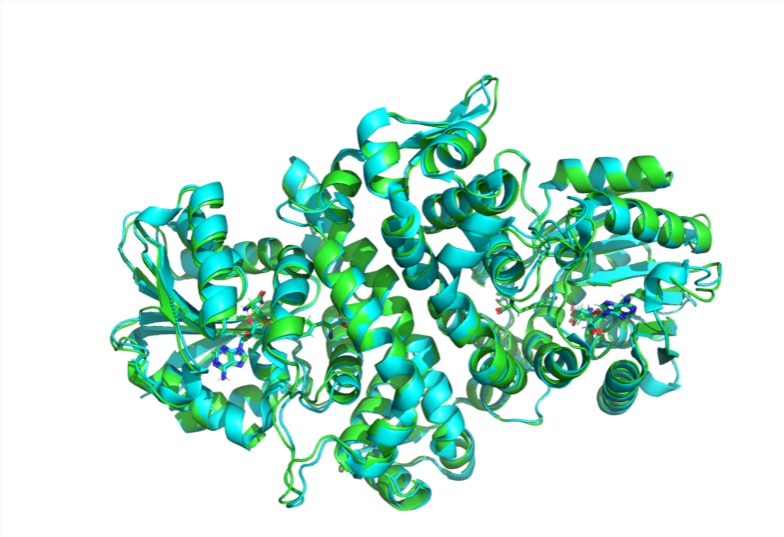
ในการเปิดตัวเมื่อวันที่ 5 ธันวาคม Wohlwind และ Corso กล่าวว่าเป้าหมายสูงสุดของพวกเขาคือการส่งเสริมความร่วมมือระดับโลก เร่งการค้นพบทางวิทยาศาสตร์ และจัดหาแพลตฟอร์มที่แข็งแกร่งเพื่อพัฒนาการสร้างแบบจำลองชีวโมเลกุล “เราหวังว่านี่จะเป็นจุดเริ่มต้นสำหรับชุมชน” คอร์โซกล่าว โดยเน้นว่าการตั้งชื่อ “โบลต์ซ-1” แทนที่จะเป็น “โบลต์ซ” มีจุดประสงค์เพื่อส่งเสริมการมีส่วนร่วมของชุมชน
โปรตีนมีบทบาทสำคัญในกระบวนการทางชีวภาพเกือบทั้งหมด และรูปร่างของโปรตีนมีความสัมพันธ์อย่างใกล้ชิดกับหน้าที่ของมัน ดังนั้นการทำความเข้าใจโครงสร้างของโปรตีนจึงเป็นสิ่งสำคัญสำหรับการออกแบบยาใหม่หรือวิศวกรรมโปรตีนใหม่ด้วยหน้าที่เฉพาะ เนื่องจากกระบวนการพับสายโซ่ยาวของกรดอะมิโนในโปรตีนให้เป็นโครงสร้างสามมิตินั้นซับซ้อนมาก การทำนายโครงสร้างของพวกมันได้อย่างแม่นยำจึงเป็นความท้าทายที่สำคัญสำหรับชุมชนวิทยาศาสตร์มาโดยตลอด
AlphaFold2 ของ DeepMind ใช้แมชชีนเลิร์นนิงเพื่อทำนายโครงสร้างโปรตีน 3 มิติอย่างรวดเร็วด้วยความแม่นยำสูงจนยากที่นักวิทยาศาสตร์ทดลองจะแยกแยะความแตกต่างได้ AlphaFold3 ได้รับการปรับปรุงบนพื้นฐานนี้และนำโมเดล AI เชิงสร้างสรรค์มาใช้ อย่างไรก็ตาม เนื่องจากมันไม่ใช่โอเพ่นซอร์สอย่างสมบูรณ์ จึงถูกวิพากษ์วิจารณ์จากชุมชนวิทยาศาสตร์ ดังนั้น ทีมวิจัยของ MIT จึงเริ่มพัฒนา Boltz-1 ตามแนวคิดพื้นฐานของ AlphaFold3 และทำการปรับปรุงบนพื้นฐานนี้เพื่อปรับปรุงความแม่นยำและประสิทธิภาพการทำนายของแบบจำลอง
ทีมวิจัยใช้เวลาสี่เดือนและทำการทดลองหลายครั้งเพื่อเอาชนะปัญหาความคลุมเครือและความหลากหลายที่พบในคลังข้อมูลโปรตีน ท้ายที่สุดแล้ว การทดลองของพวกเขาแสดงให้เห็นว่า Boltz-1 มีความแม่นยำเช่นเดียวกับ AlphaFold3 ในการทำนายโครงสร้างของชีวโมเลกุลที่ซับซ้อน
นักวิจัยวางแผนที่จะปรับปรุงประสิทธิภาพของ Boltz-1 ต่อไป และลดระยะเวลาการคาดการณ์ให้สั้นลง พวกเขายังเชิญชวนนักวิจัยให้ลองใช้ Boltz-1 บน GitHub และสื่อสารกับผู้ใช้รายอื่นผ่านช่องทาง Slack ทีมวิจัยหวังว่า Boltz-1 จะส่งเสริมการทำงานร่วมกันในวงกว้างขึ้น และสร้างแรงบันดาลใจให้เกิดการใช้งานเชิงสร้างสรรค์ในชุมชน
โครงการ: https://jclinic.mit.edu/democratizing-science-boltz-1/
ไฮไลท์:
Boltz-1 เป็นโมเดลทำนายโครงสร้างชีวโมเลกุลแบบโอเพ่นซอร์สรุ่นแรกที่มีประสิทธิภาพเทียบเท่ากับ AlphaFold3
แบบจำลองนี้ได้รับการพัฒนาเพื่อส่งเสริมความร่วมมือระดับโลกและการพัฒนาการวิจัยทางชีวการแพทย์และยาขั้นสูง
ทีมงาน MIT หวังว่าจะใช้ Boltz-1 เพื่อลดความซับซ้อนในการทำนายโครงสร้างโปรตีน เพื่อให้นักวิจัยสามารถใช้เครื่องมืออันทรงพลังนี้ได้มากขึ้น
ลักษณะโอเพ่นซอร์สของ Boltz-1 ช่วยลดเกณฑ์การวิจัยทางชีวการแพทย์ ช่วยให้นักวิทยาศาสตร์ทั่วโลกมีเครื่องมืออันทรงพลัง และเร่งกระบวนการพัฒนายา การมีส่วนร่วมของทีม MIT นี้จะมีผลกระทบอย่างมากต่อการวิจัยทางชีวการแพทย์ในอนาคตอย่างไม่ต้องสงสัย เราหวังว่า Boltz-1 จะประสบความสำเร็จในการใช้งานในอนาคต