最后更新:2024 年 9 月 30 日
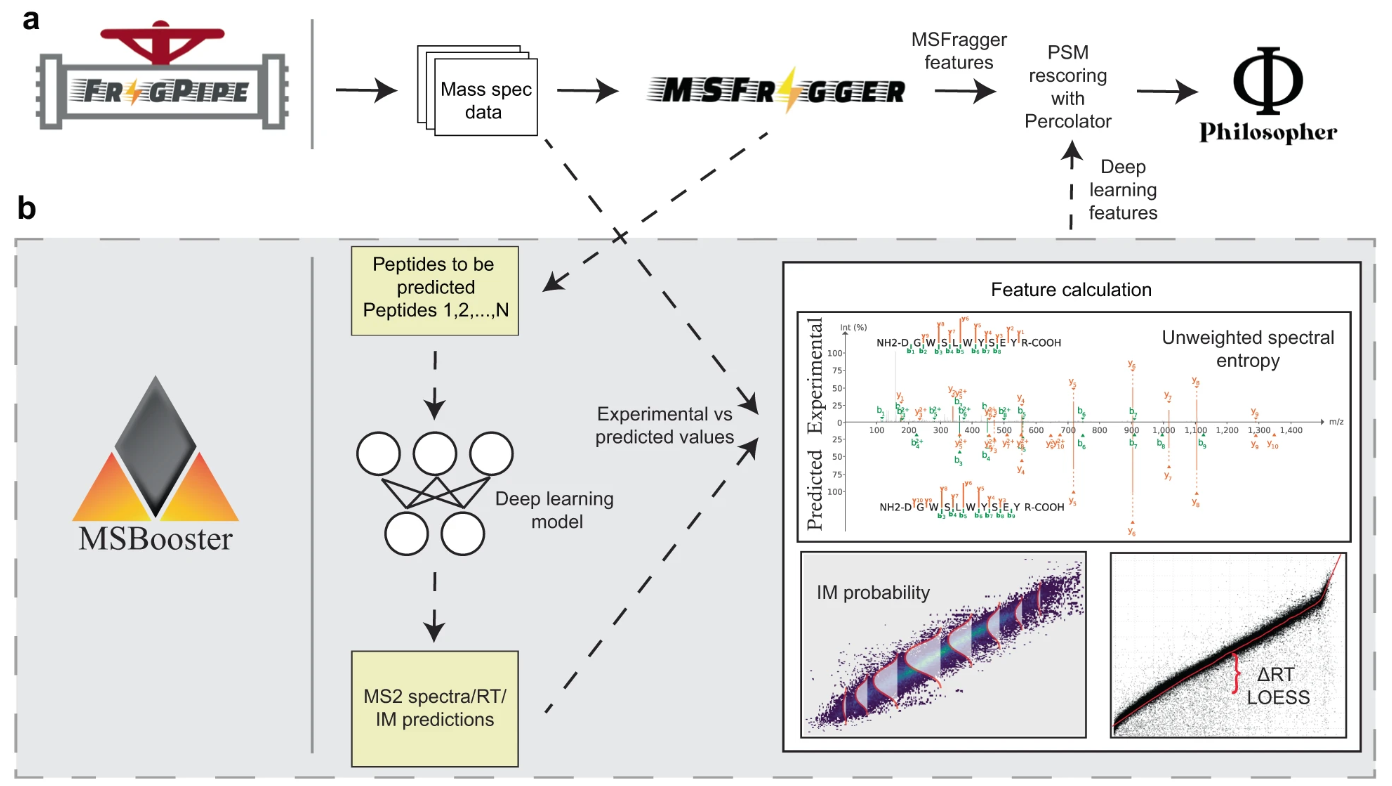
MSBooster 是一种工具,用于将谱库预测纳入自下而上串联液相色谱质谱蛋白质组学数据中的肽谱匹配 (PSM) 重新评分中。大致分为4步:
从搜索结果中的 PSM 中提取肽,并格式化机器/深度学习 (ML/DL) 预测器的输入文件
调用预测模型并保存输出
特征计算
向搜索结果文件添加新功能
MSBooster 与多种类型的数据库搜索兼容,包括 HLA 免疫肽组学、DDA 和 DIA 以及单细胞蛋白质组学。它被合并到 FragPipe 中并包含在其许多工作流程中。 MSBooster 是在开发时考虑了其他 FragPipe 工具,例如 FragPipe-PDV。
MSBooster 可以处理多种输入文件格式和模型:
| 质谱仪输出 |
|---|
| .mzML |
| .mgf |
| PSM文件 |
|---|
| 。别针 |
| .pepXML(正在进行中) |
| 预测模型 |
|---|
| DIA-NN |
| 科伊纳模型 |
MSBooster可以在Windows和Linux系统中运行。如果使用FragPipe,除了安装FragPipe外,不需要其他安装步骤。 MSBooster 位于“验证”选项卡中。选择启用“预测 RT”的保留时间功能和“预测光谱”的 MS/MS 光谱功能。有关如何运行分析的信息,请参阅 FragPipe 文档。
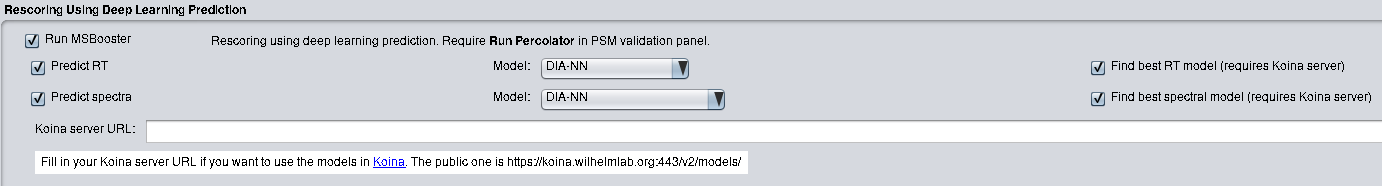
如果使用独立的 MSBooster 在命令行中运行,请从 Releases 下载最新的 jar 文件。 MSBooster 还需要 DIA-NN 进行 MS/MS 和 RT 预测。请安装 DIA-NN 并记下 DIA-NN 可执行文件的路径(例如 Windows 中的 DiaNN.exe,Linux 中的 diann-1.8.1.8)。
您可以使用类似于以下内容的命令运行 MSBooster:
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
需要传递的最少参数是:
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
虽然您可以单独传递这些参数,但在 paramsList 文件的每一行上放置一个参数会更容易。请参阅 msbooster_params.txt 获取模板。
以下参数仅供一般使用。 Koina 特定参数位于 Koina 文档中
paramsList (String) :包含此运行参数的文本文件的位置
fragger (String) :MSFragger 运行中的 fragger.params 文件的文件路径。 MSBooster将读入多个参数并根据它们调整内部参数,例如碎片质量误差容限和质量偏移
outputDirectory (String) :输出新文件的位置
editedPin (String) :MSBooster 将根据提供的文件命名新文件。例如,A.pin 将有一个名为 A_edited.pin 的对应项。要更改默认的“已编辑”,请在此处提供新字符串
renamePin (int) :是否生成新的 pin 文件或重写旧的 pin 文件。这里默认是1,不会覆盖。设置为 0 将覆盖旧的 pin 文件
deletePreds (boolean) :成功运行后是否删除存储模型预测的文件。默认情况下,设置为 false。如果您想删除这些,请设置为 true
loadingPercent (int) :使用进度报告器报告任务进度的频率。默认情况下,设置为 10,表示每 10% 打印一次更新。
numThreads (int) :要使用的线程数。默认设置为 0,即使用所有可用线程减 1
splitPredInputFile (int) :仅在 DIA-NN 预测因内存不足错误而失败时使用 (137)。默认情况下,设置为 1,但您可以增加此值以指定 DIA-NN 输入文件应分解为多少个较小的文件。然后将按顺序预测每个文件,减轻内存负担
plotExtension (String) :绘图应采用什么文件格式。默认为 png,也允许 pdf
features (String) :要计算的特征列表。区分大小写,以逗号分隔,中间没有空格。默认为“predRTrealUnits,unweightedSpectralEntropy,deltaRTLOESS”
spectraPredFile (String) :如果您要重用旧的光谱预测(例如来自 DIA-NN 或 Koina),您可以在此处指定文件位置
RTPredFile (String) :与spectrumPredFile相同,但用于RT预测
IMPredFile (String) :与 SpectrumPredFile 相同,但用于 IM 预测
spectraModel (String) :使用哪个光谱预测模型
rtModel (String) :与spectrumModel相同,但用于RT
imModel (String) :与 SpectrumModel 相同,但用于 IM
useSpectra (boolean) :是否使用基于光谱预测的特征。默认设置为 true
useRT (boolean) :是否使用基于 RT 预测的特征。默认设置为 true
useIM (boolean) :是否使用基于 IM 预测的功能。默认设置为 false
ppmTolerance (float) :片段错误 ppm 容差(默认 20ppm)
matchWithDaltons (boolean) : 是否匹配预测和观察到的片段(以道尔顿为单位)(默认 false)
DaTolerance (float) :在预测峰周围有多少道尔顿来寻找实验峰(默认 0.05)
useTopFragments (boolean) :是否将光谱预测过滤到 N 个最高强度峰值(默认 true)
topFragments (int) :最多应使用多少个预测片段进行特征计算(默认 20)。仅当 useTopFragments 为 true 时才应用
removeRankPeaks (boolean) :默认设置为 true,一旦匹配,就会从实验光谱中过滤掉片段。如果为 false,则实验片段可以通过同一扫描中的多个 PSM 进行匹配
useBasePeak (boolean) :是否应将下限应用于 MS2 预测,以仅使用强度较高的片段(默认 true)
percentBasePeak (float) :相似性计算中包含强度为基峰强度某个百分比的片段的百分比。仅当 useBasePeak 为 true 时才应用(默认 1)
loessEscoreCutoff (float) :用于收集 PSM 以进行 RT/IM 校准的第一遍的期望值截止值。默认值为 10^-3.5,或大约 0.000316
rtLoessRegressionSize (int) :用于 RT LOESS 校准的 PSM 最大数量(默认 5000)
imLoessRegressionSize (int) :与 rtLoessRegressionSize 相同,但用于 IM(默认 1000)
minLoessRegressionSize (int) :尝试 LOESS RT/IM 校准所需的最小 PSM 数量(默认 100)。如果可用的 PSM 数量少于此数量,则使用线性回归
minLinearRegressionSize (int) :尝试线性回归 RT/IM 校准所需的最小 PSM 数量(默认 10)。如果可用的 PSM 数量少于此数量,则不会尝试校准
loessBandwidth (String) :尝试进行 RT/IM LOESS 校准的带宽列表(默认 0.01,0.05,0.1,0.2)。必须以逗号分隔,中间不能有空格
regressionSplits (int) :用于 RT/IM LOESS 校准的交叉验证数量(默认 5)
massesForLoessCalibration (String) :质量偏移的质量应适合其自己的校准曲线。列表以逗号分隔,中间没有空格。质量数应写入与 PIN 文件中相同的位数
loessScatterOpacity (float) : LOESS 校准图中散点图的不透明度,从 0 到 1(默认 0.35)
.pin 文件具有新功能。默认情况下,将生成以“_edited.pin”结尾的新 pin 文件。使用的默认特征是“unweighted_spectral_entropy”、“delta_RT_loess”和“pred_RT_real_units”。如果启用离子淌度功能,还将包括“delta_IM_loess”和“ion_mobility”
SpectrumRT.tsv 和 SpectrumRT_full.tsv:DIA-NN 预测模型的输入文件
SpectrumRT.predicted.bin:包含 DIA-NN 预测的二进制文件,MSBooster 将使用该文件进行特征计算。如果使用 FragPipe-PDV,这些文件用于生成实验和预测光谱的镜像图
MSBooster 生成多个图表,可用于进一步检查数据与模型预测的比较情况。
MSBooster_plots 文件夹:
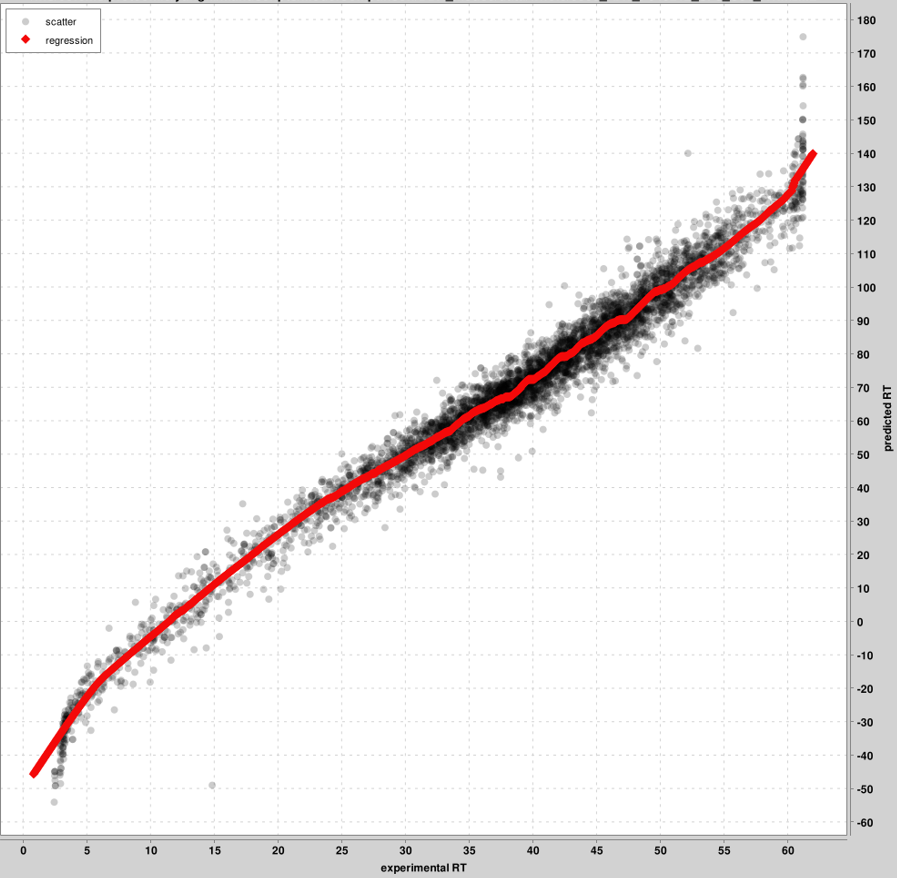
RT_calibration_curves:最多 5000 个 PSM 将用于实验和预测 RT 尺度之间的校准。图中显示了这些顶级 PSM,而不是所有 PSM。每个 pin 文件将生成一张图表
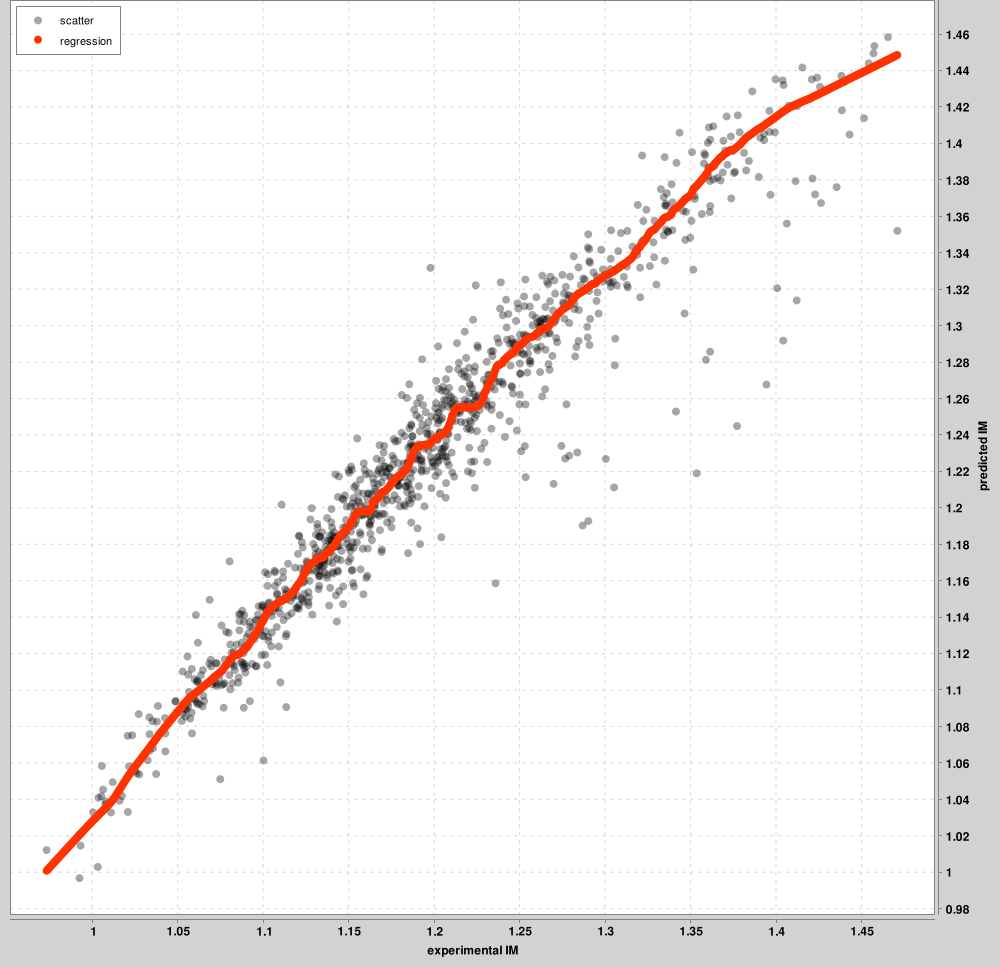
IM_calibration_curves:最多前 1000 个 PSM 将用于实验和预测 IM 尺度之间的校准。图中显示了这些顶级 PSM,而不是所有 PSM。将为每个充电状态学习一条单独的曲线。下图是电荷2前驱体的示例
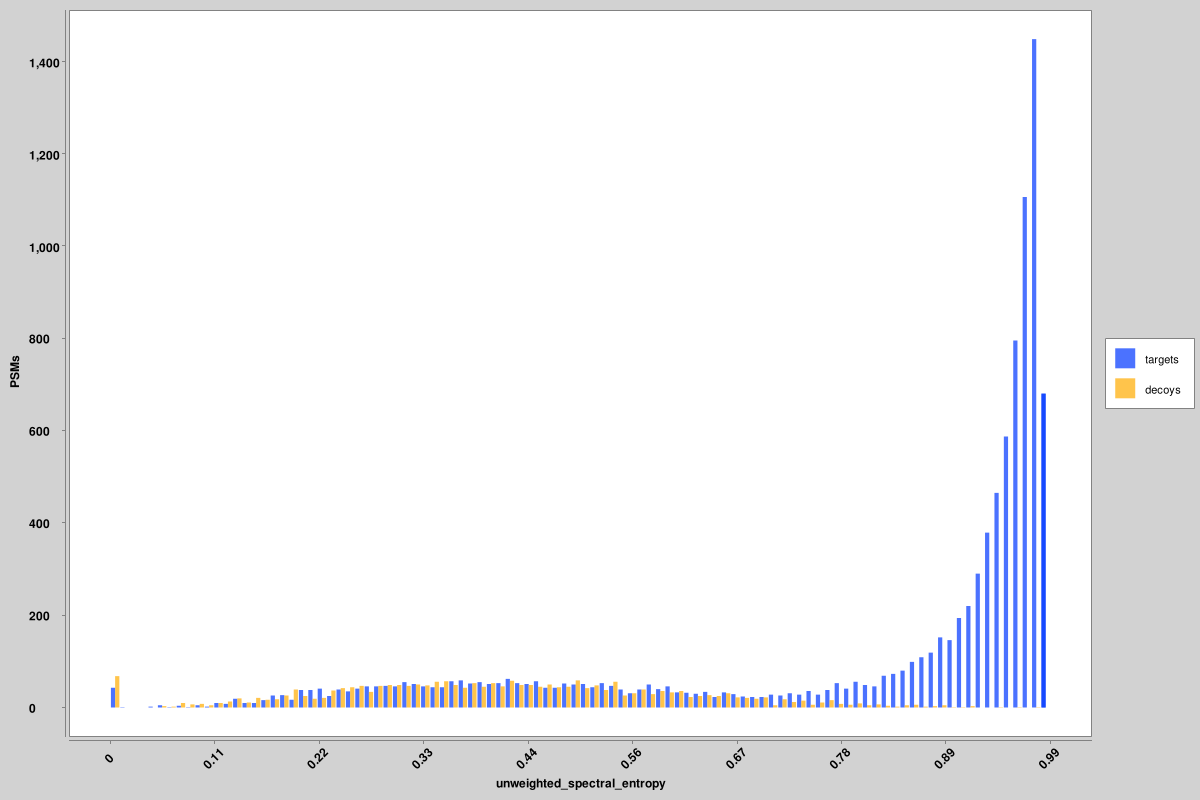
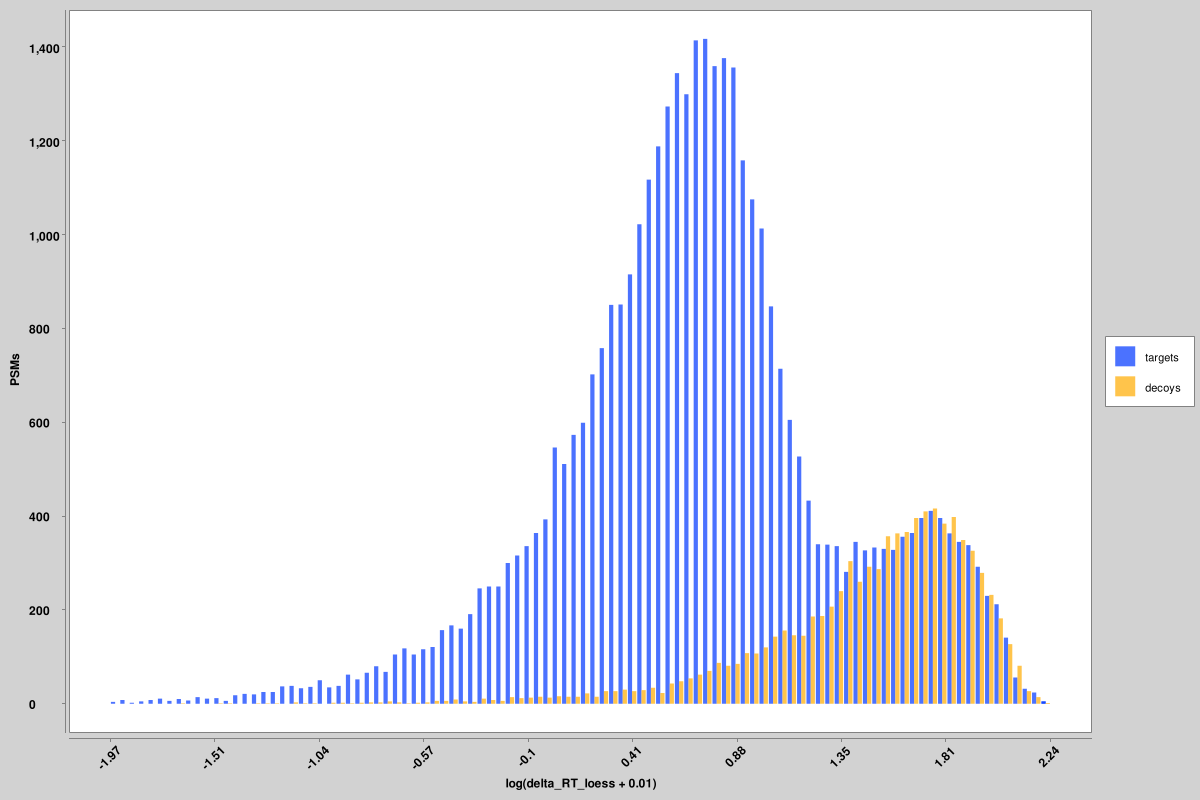
Score_histograms:每个 pin 文件的所有目标和诱饵 PSM 的叠加直方图。此处以对数刻度绘制了一些特征,以便更好地可视化真值和误报的双峰分布,但原始值是 pin 文件中使用的值,而不是对数刻度版本。这里显示的是未加权谱熵和 delta RT 分数的直方图,但所有特征都会生成类似的直方图
使用 Koina 的肽预测模型进行 MSBooster 特征生成:https://fragpipe.nesvilab.org/docs/tutorial_koina.html
通过 MGF 文件读取任何模型的预测
有关所有允许的功能以及如何使用图形输出对其进行质量控制的文档
使用 MSBooster 时请引用以下内容:https://www.nature.com/articles/s41467-023-40129-9