这是“一个模型来统治一切:通过文本提示实现医学图像的通用分割”的官方存储库
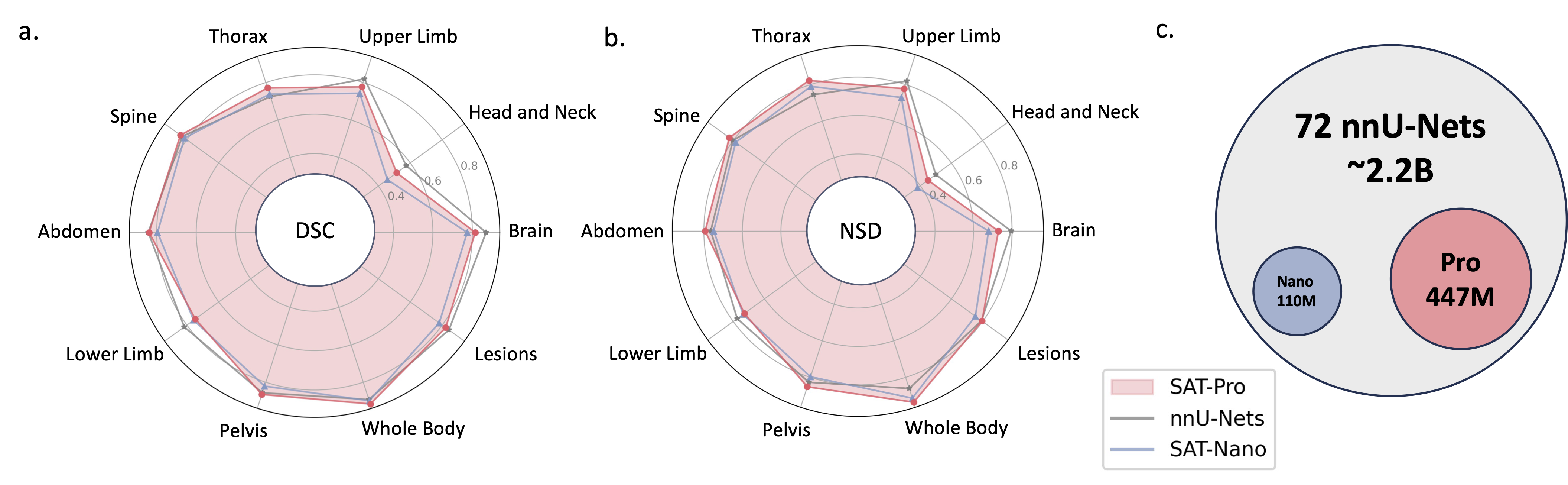
它是一个建立在前所未有的数据收集(72 个公共 3D 医学分割数据集)之上的知识增强型通用分割模型,可以根据文本(解剖学)提示,对来自 3 种不同模式(MR、CT、PET)和 8 个人体区域的 497 个类别进行分割术语)。
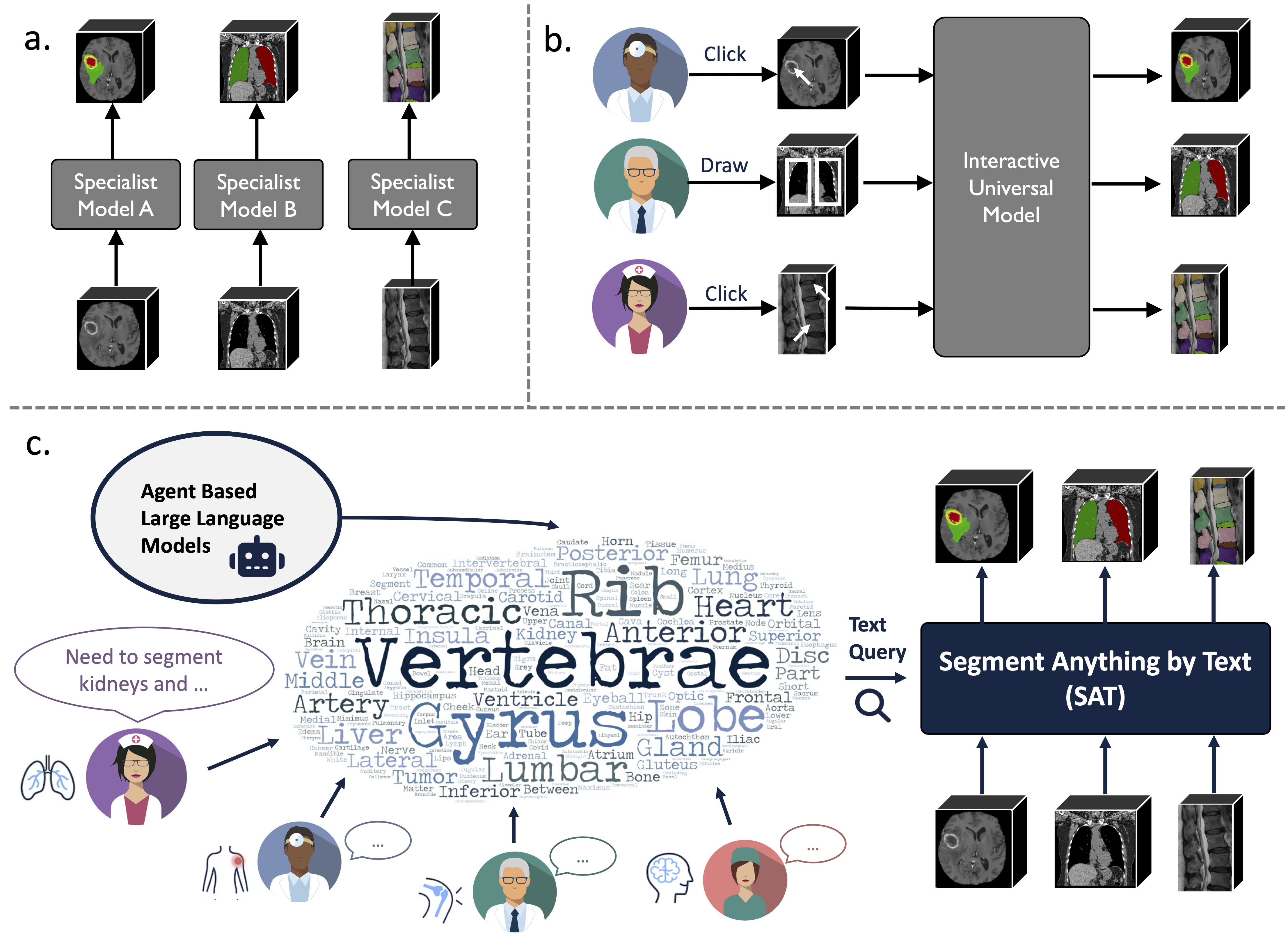
它比训练和部署一系列专业模型更强大、更高效。在我们的网站或报纸上查找更多信息。
2024.08?基于SAT和大语言模型,我们构建了一个全面的、大规模的、区域引导的3D胸部CT判读数据集。它包含 196 个类别的器官级分割和多粒度报告,其中每个句子都基于相应的分割。在“hushingface”上检查一下。
2024.06?我们已经发布了构建SAT-DS 的代码,它是 72 个公共分割数据集的集合,包含超过 22K 3D 图像、302K 分割掩模和来自 3 种不同模式(MRI、CT、PET)和 8 个人体区域的 497 个类别。我们建立SAT。我们还提供 42/72 数据集的快捷下载链接,这些数据集经过我们的预处理和打包,以方便您下载和提取后立即使用。检查此存储库以获取详细信息。
2024.05?我们训练了新版本的 SAT,模型尺寸更大( SAT-Pro ),数据集更多( 72 ),现在支持497 个类别!我们还更新了 SAT-Nano,并基于不同的视觉主干(U-Mamba 和 SwinUNETR)和文本编码器(MedCPT 和 BERT-Base)发布了 SAT-Nano 的一些变体。有关此更新的更多详细信息,请参阅我们的新论文。
U-Net的实现依赖于一个定制版本的dynamic-network-architectures,安装它:
cd model
pip install -e dynamic-network-architectures-main
其他一些关键要求:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
如果您想要 SAT-Nano 的 U-Mamba 变体,您还需要安装mamba_ssm
S1。按照requirements.txt构建环境。
S2。从 Huggingface 下载 SAT 和文本编码器的检查点。
S3。准备 jsonl 文件中的数据。检查data/inference_demo/demo.jsonl中的演示。
每个要分割的样本都需要image (图像路径)、 labe (分割目标名称)、 dataset (样本所属的数据集)和modality (ct、mri 或 pet)。 SAT 支持的模式和类别可以在本文的表 12 中找到。
orientation_code (方向)默认为RAS ,适合大多数轴面图像。对于矢状面中的图像(例如脊柱检查),将其设置为ASR 。输入图像应具有H,W,D形状。我们的数据处理代码将在方向、强度、间距等方面对输入图像进行标准化。 demoprocessed_data中可以找到两张处理成功的图像,请确保正确完成归一化以保证 SAT 的性能。
S4。使用 SAT-Pro 开始推理?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d是输入图像块的批量大小,需要根据 GPU 内存进行调整(查看下表); --max_queries建议设置大于推理数据集中的类,除非你的 GPU 内存非常有限;
| 模型 | 批量大小_3d | 显存 |
|---|---|---|
| 卫星专业版 | 1 | 〜34GB |
| 卫星专业版 | 2 | 〜62GB |
| SAT-纳米 | 1 | 〜24GB |
| SAT-纳米 | 2 | 〜36GB |
S5。检查--rcd_dir的输出。结果按数据集组织。对于每种情况,都会找到输入图像、聚合分割结果和包含每个类别分割的文件夹。所有输出都存储为 nifiti 文件。您可以使用 ITK-SNAP 将它们可视化。
如果您想使用在 72 个数据集上训练的 SAT-Nano,只需将--vision_backbone修改为“UNET”,并相应地更改--checkpoint和--text_encoder_checkpoint即可。
对于其他 SAT-Nano 变体(在 49 个数据集上训练):
UNET-我们的:设置--vision_backbone 'UNET'和--text_encoder 'ours' ;
UNET-CPT:设置--vision_backbone 'UNET'和--text_encoder 'medcpt' ;
UNET-BB:设置--vision_backbone 'UNET'和--text_encoder 'basebert' ;
UMamba-CPT:设置--vision_backbone 'UMamba'和--text_encoder 'medcpt' ;
SwinUNETR-CPT:设置--vision_backbone 'SwinUNETR'和--text_encoder 'medcpt' ;
开始训练前的一些准备:
sh/中的 slurm 脚本来启动训练过程。以SAT-Pro为例: sbatch sh/train_sat_pro.sh
这还需要在此存储库之后构建测试数据。您可以参考 slurm 脚本sh/evaluate_sat_pro.sh来启动评估过程:
sbatch sh/evaluate_sat_pro.sh
如果您将此代码用于您的研究或项目,请引用:
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}