该项目包括对总体进行抽样的不同方法以及对不同方法的评估。我们纳入了许多可能导致基于样本的感染水平估计出现偏差的情况,包括无应答者、假阳性/阴性率、患者在感染期间的传播能力。基于EpiABM模型,该软件包还可以通过运行疾病传播模拟来输出最佳采样方法,以查看每种采样方法的预测误差。
EpiOS 尚未在 PyPI 上使用,但该模块可以在本地 pip 安装。应首先将该目录下载到本地计算机,然后可以使用以下命令进行安装:
pip install -e .我们还建议您安装EpiABM模型来生成感染模拟的数据。您可以首先将 pyEpiabm 下载到计算机上的任何位置,然后可以使用以下命令进行安装:
pip install -e path/to/pyEpiabm 可以通过上述docs徽章访问文档。
这是我们项目的 UML 类图: 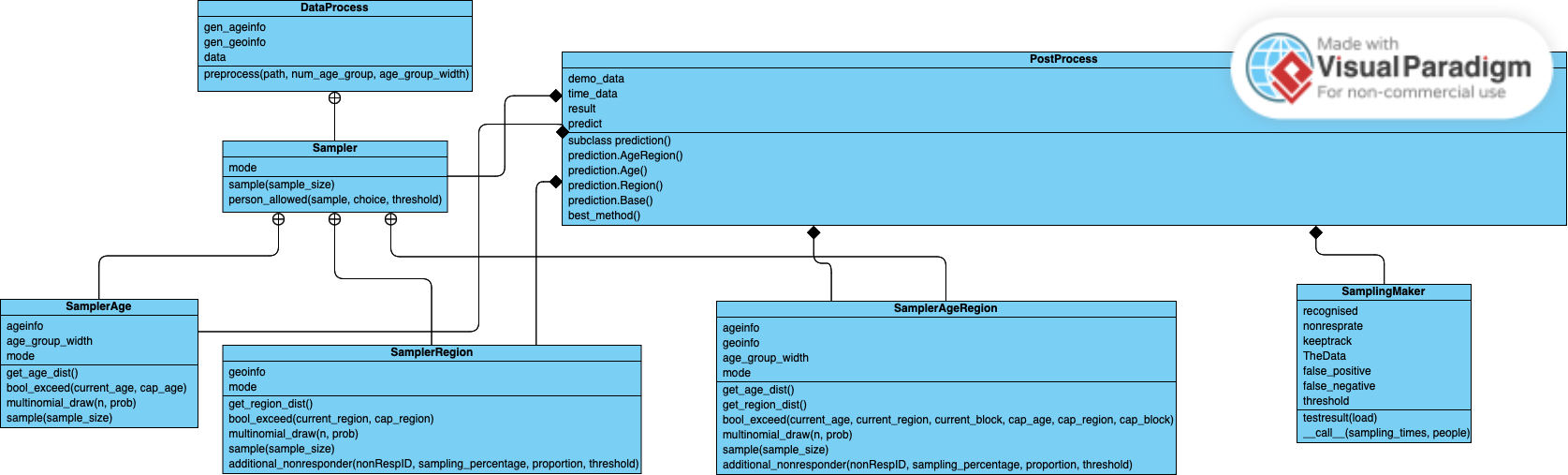
params.py文件包含该模型所需的所有参数。另外, input文件夹中的文件是数据预处理过程中生成的临时文件的示例。它将由采样器类使用。每个采样器类中的data_store_path参数是存储这些文件的路径。
PostProcess生成绘图首先,您需要定义一个新的PostProcess对象并输入 pyEpiabm 生成的人口统计数据demodata和感染数据timedata 。其次,您可以使用PostProcess.predict根据不同的采样方法进行预测。可以直接调用你想要使用的采样方法作为方法;然后指定采样的时间点和样本大小。这里,我们将使用AgeRegion作为采样方法, [0, 1, 2, 3, 4, 5]作为采样时间点, 3作为样本大小。最后,您可以通过指定参数non_responder和comparison来指定是否要考虑非响应者以及是否要将结果与真实数据进行比较。
对于代码示例,您可以看到以下内容:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)现在,您的图形将保存到给定的路径!
PostProcess选择最佳采样方法首先,您需要定义一个新的PostProcess对象并输入 pyEpiabm 生成的人口统计数据demodata和感染数据timedata 。其次,您可以使用PostProcess.best_method来比较不同采样方法的性能。您可以提供您想要比较的方法;然后指定采样间隔和样本大小。第三,您可以通过指定参数non_responder和comparison来指定是否要考虑非响应者以及是否要将结果与真实数据进行比较。此外,由于采样方法是随机的,因此您可以指定运行的迭代次数以获得平均性能。而且,可以开启parallel_computation来加速。最后,您可以打开hyperparameter_autotune自动查找超参数的最佳组合。
对于代码示例,您可以看到以下内容:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed