guar_genotypes
1.0.0
包含瓜尔豆 ( Cyamopsis tetragonoloba (L.) Taub.) 基因分型和 GWAS 项目的数据和代码的存储库。存储库内容的简要描述:
make_consensus_vcf.py脚本用于使用原始 GATK-HC、NGSEP 和 TASSEL 5 变体调用生成最终变体数据集, make_snp_stats.py脚本用于相同的输入以探索变体调用的质量统计数据。
callset_refinement_filtering.ipynb笔记本用于运行变体质量控制并构建常见变体的最终过滤数据集。
genetic_analysis.ipynb笔记本用于执行大部分遗传分析,包括 PCA、连锁不平衡分析和基于广义线性模型的关联分析。
使用prepare_farmcpu.py脚本来格式化FarmCPU 工具的输入数据(所得p 值的QQ 图显示在论文和下面)。
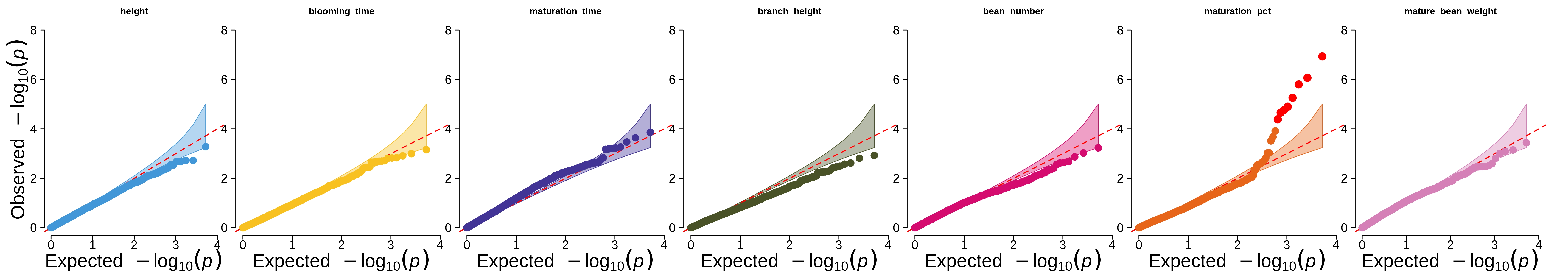
admix_file_create.py文件用于预处理 ADMIXTURE 分析的基因型(其中部分是使用genetic_analysis.ipynb中的 Hail 完成的)。
guar_stat.R脚本用于对最终数据进行统计分析,使用FarmCPU工具进行全基因组关联分析,并绘制主要数据
parse_vcf_fcpu.py脚本用于格式化最终的 SNP 基因型表,该表可作为此存储库中的genotypes.xlsx文件使用。