该软件包提供经过训练的 U-net 模型用于肺部分割。目前有四种型号可供选择:
U-net(R231):该模型是在涵盖广泛视觉变量的大型且多样化的数据集上进行训练的。该模型对各个切片进行分割,分别提取左右肺,包括气囊、肿瘤和积液。气管不会包含在肺分割中。 https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRCLobes):该模型是在 LTRC 数据集的子集上进行训练的。该模型对各个肺叶进行分割,但当存在密集的病变或每个切片上不可见裂隙时,其性能有限。
U-net(LTRCLobes_R231):这将运行 R231 和 LTRCLobes 模型并融合结果。 LTRCLobes 的假阴性将由 R231 预测填充并映射到邻居标签。 LTRCLobes 的误报将被删除。融合过程需要大量计算,并且根据数据和结果,每个卷可能需要长达几分钟的时间。
U-net(R231CovidWeb)
两种模型的应用实例。左: U-net(R231),将区分左肺和右肺,包括非常密集的区域,例如积液(第三行)、肿瘤或严重纤维化(第四行)。右: U-net(LTRLobes),将区分肺叶,但不包括非常密集的区域。 LTRCLobes_R231将融合 LTRCLobes 和 R231 结果。 R231CovidWeb使用额外的 COVID-19 数据进行训练。
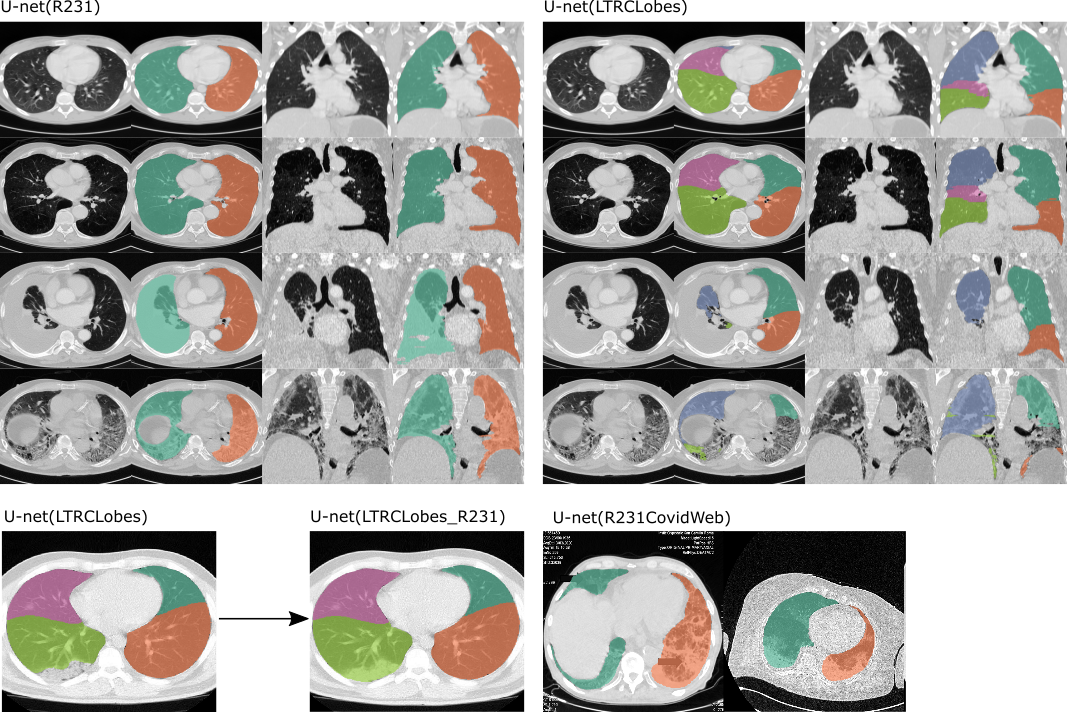
输出的语义:
两种标签模型(左-右):
1 = 右肺
2 = 左肺
五个标签模型(肺叶):
1 = 左上叶
2 = 左下叶
3 = 右上叶
4 = 右中叶
5 = 右下叶
有关肺部 CT 数据的更多令人兴奋的研究,请查看我们研究小组的网站:https://www.cir.meduniwien.ac.at/research/lung/
如果您在工作中使用此代码或经过训练的模型之一,请参阅:
Hofmanninger, J.、Prayer, F.、Pan, J. 等人。常规成像中的自动肺部分割主要是数据多样性问题,而不是方法问题。欧洲无线电指数 4, 50 (2020)。 https://doi.org/10.1186/s41747-020-00173-2
本文包含对所用数据集的详细描述、对 U-net(R231) 模型的全面评估以及与参考方法的比较。
pip install lungmask
或者
pip install git+https://github.com/JoHof/lungmask
在 Windows 上,根据您的设置,可能需要提前安装 torch:https://pytorch.org
仅 CPU 推理和 GPU 支持推理之间的运行时间差异很大。使用 GPU 处理一个卷只需几秒钟,仅使用 CPU 则需要几分钟。要使用 GPU,请确保您的手电筒安装支持 CUDA。如果出现 cuda 内存不足错误,请使用可选参数--batchsize 1将批大小减少到 1
lungmask INPUT OUTPUT
如果 INPUT 指向一个文件,则该文件将被处理。如果INPUT指向一个目录,则会在该目录中搜索DICOM系列。找到的最大体积(就体素数量而言)将用于计算肺掩模。 OUTPUT 是输出文件名。支持所有 ITK 格式。
选择型号:
默认使用 U-net(R231)。但是,您可以指定替代模型,例如 LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
对于其他选项,请输入:
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image 必须是 SimpleITK 对象。
加载替代模型,如下所示:
inferer = LMInferer(modelname="R231CovidWeb")
要将模型融合功能用于(例如 LTRCLobes_R231),请使用:
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
从版本 0.2.9 开始,支持 numpy 数组作为输入卷。此模式假设输入 numpy 数组的每个轴具有以下格式:
该模型仅适用于完整切片。要处理的切片必须显示完整的肺部,并且肺部必须被组织包围才能进行分割。然而,只要肺部被组织包围,该模型对于视野被裁剪的情况就相当稳定。
lungmask INPUT OUTPUT --modelname R231CovidWeb
常规 U-net(R231) 模型非常适合 COVID-19 CT 扫描。然而,来自网络的切片和病例报告的集合通常会被裁剪、注释或以常规图像格式编码,因此只能估计原始的亨斯菲尔德单位 (HU) 值。 U-net(R231CovidWeb) 模型的训练数据通过从常规成像格式映射回 HU 的 COVID-19 切片进行了增强。数据由 MedSeg (http://medicalsegmentation.com/covid19/) 收集和准备。虽然常规 U-net(R231) 对这些图像显示出非常好的结果,但在某些情况下,该模型可能会产生稍微改进的分割效果。请注意,使用网络图像时,您必须将图像映射回 HU。这篇博文描述了如何做到这一点。或者,您可以设置--noHU标签。 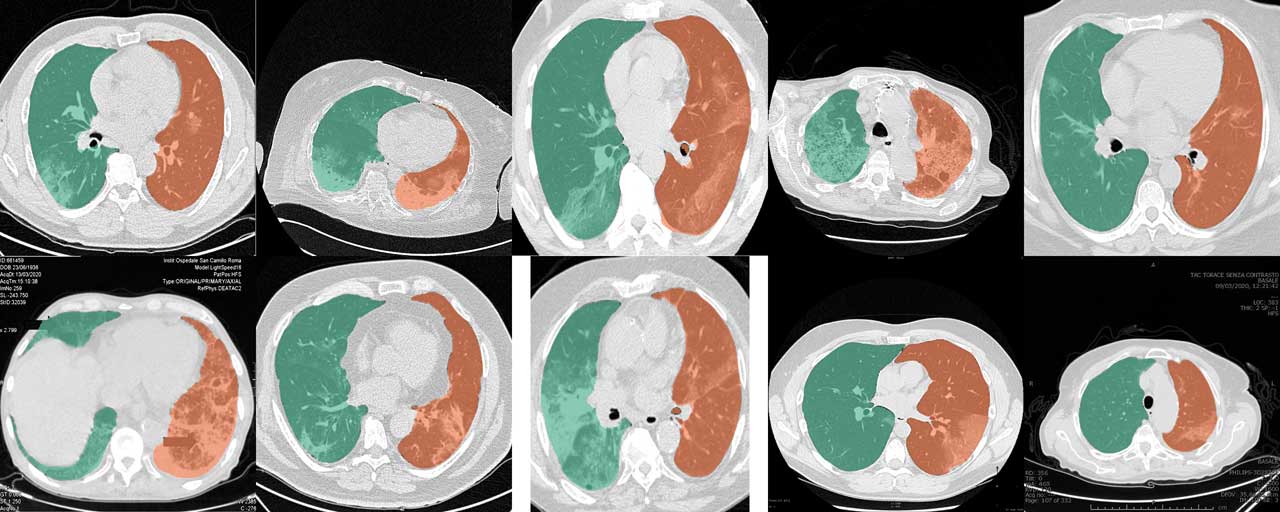
此功能仅在 0.2.5 和 0.2.14 之间的版本中可用。从版本 0.2.5 开始,支持这些图像。如果处理未以 HU 编码的图像,请使用--noHU标记。请记住,模型是根据 HU 中编码的正确 CT 扫描进行训练的。裁剪后的、带注释的、非常高和非常低强度的偏移图像的结果可能不是很可靠。当使用--noHU标签时,只能处理单个切片。