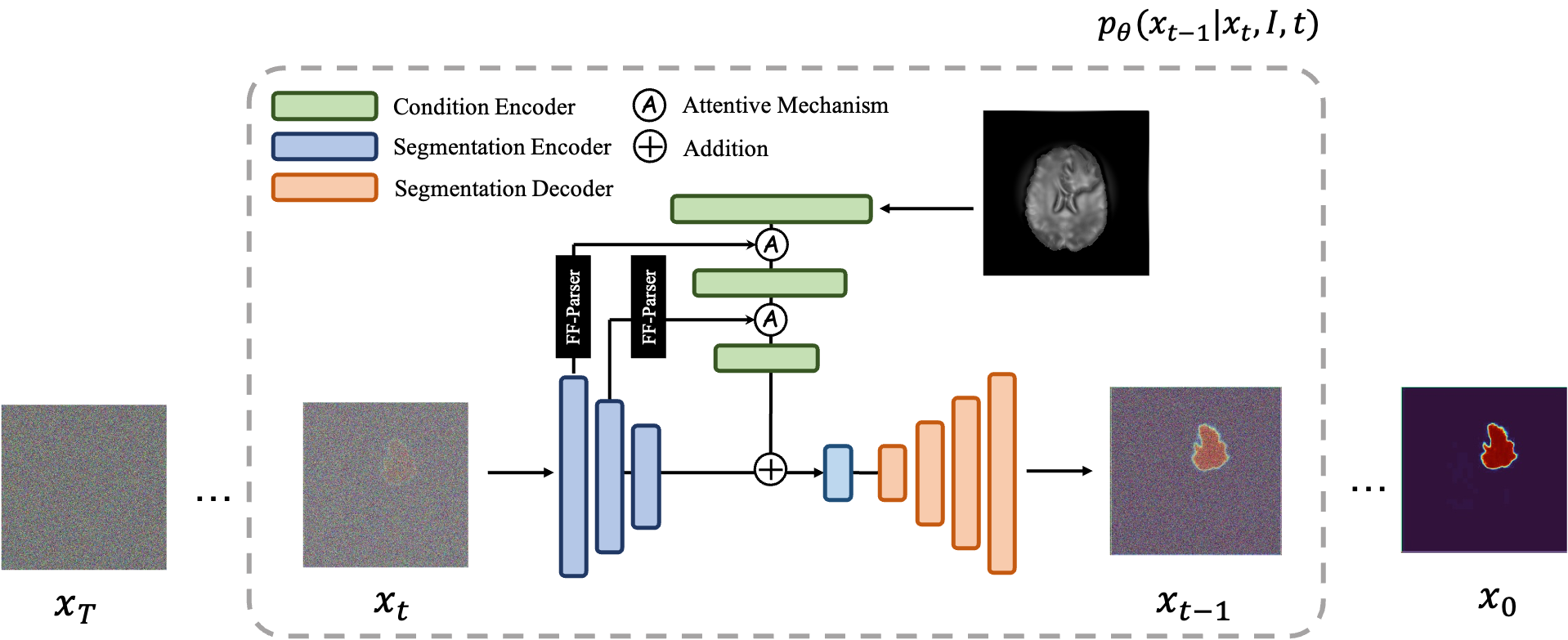
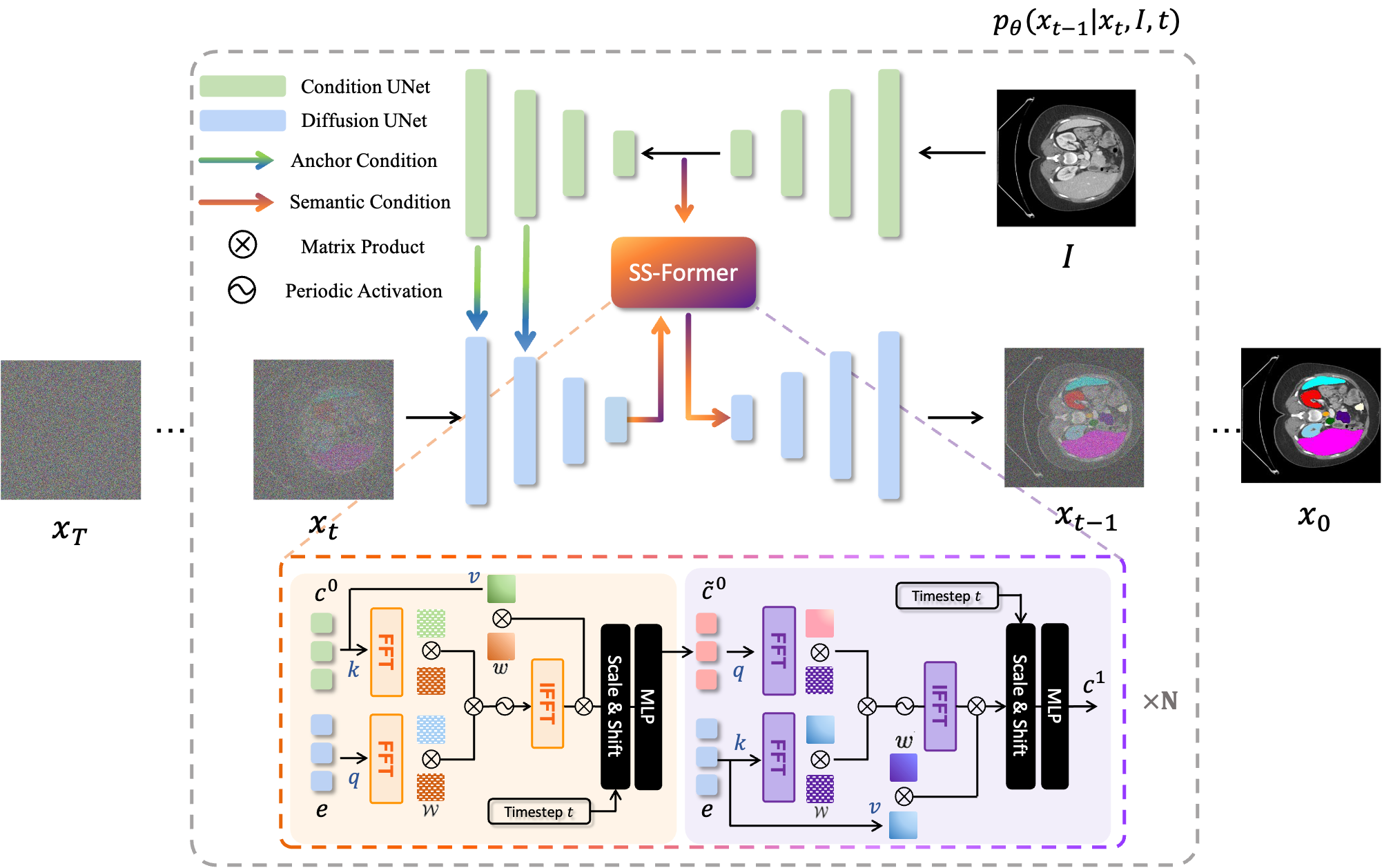
MedSegDiff 是一种基于扩散概率模型 (DPM) 的医学图像分割框架。该算法在我们的论文 MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model 和 MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer 中进行了详细阐述。
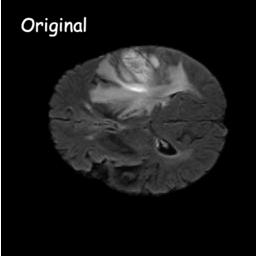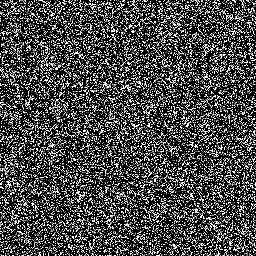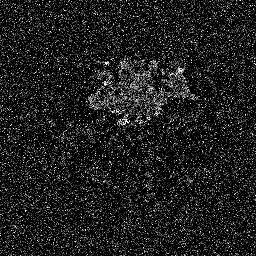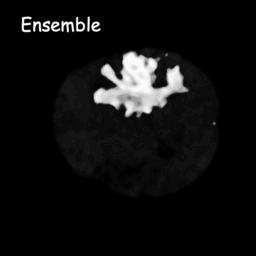 扩散模型的工作原理是通过连续添加高斯噪声来破坏训练数据,然后学习通过逆转该噪声过程来恢复数据。训练后,我们可以使用扩散模型通过学习的去噪过程简单地传递随机采样的噪声来生成数据。在这个项目中,我们将这个想法扩展到医学图像分割。我们利用原始图像作为条件,从随机噪声中生成多个分割图,然后对它们进行集成以获得最终结果。这种方法捕获了医学图像中的不确定性,并且在多个基准测试中优于以前的方法。
扩散模型的工作原理是通过连续添加高斯噪声来破坏训练数据,然后学习通过逆转该噪声过程来恢复数据。训练后,我们可以使用扩散模型通过学习的去噪过程简单地传递随机采样的噪声来生成数据。在这个项目中,我们将这个想法扩展到医学图像分割。我们利用原始图像作为条件,从随机噪声中生成多个分割图,然后对它们进行集成以获得最终结果。这种方法捕获了医学图像中的不确定性,并且在多个基准测试中优于以前的方法。
 |  |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver True享受其闪电般的采样速度(1000 步 20 步⭕️)。python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
对于训练,运行: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
对于采样,请运行: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
为了进行评估,运行python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
默认情况下,样本将保存在./results/
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
对于训练,运行: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
对于采样,运行: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
在其他数据集上运行 MedSegDiff 很简单。只需在./guided_diffusion/isicloader.py或./guided_diffusion/bratsloader.py之后编写另一个数据加载器文件。如果您遇到任何问题,欢迎打开问题。如果您能贡献您的数据集扩展,我们将不胜感激。与自然图像不同,医学图像根据不同的任务而有很大差异。扩大一种方法的泛化性需要每个人的努力。
为了训练一个精细的模型,即论文中的MedSegDiff-B,将模型超参数设置为:
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
扩散超参数为:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
为了加快采样速度:
--diffusion_steps 50 --dpm_solver True
在多个 GPU 上运行:
--multi-gpu 0,1,2 (for example)
训练超参数为:
--lr 5e-5 --batch_size 8
并在采样中设置--num_ensemble 5 。
训练中运行大约 100,000 个步骤将在大多数数据集上收敛。请注意,虽然后面的大多数步骤中损失不会减少,但结果的质量仍在提高。在其他 DPM 应用程序(例如图像生成)上也可以观察到这样的过程。希望懂的人能告诉我这是为什么吗?
我很快就会发布它在较小批量大小(适合在 24GB GPU 上运行)下的性能,以供比较。
释放其所有潜力的设置是 (MedSegDiff++):
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
然后使用批量大小--batch_size 64对其进行训练,并使用集合编号--num_ensemble 25对其进行采样。
欢迎为 MedSegDiff 做出贡献。任何可以提高性能或加速算法的技术都是值得赞赏的。我正在写 MedSegDiff V2,目标是 Nature 期刊/CVPR 之类的出版物。我很高兴将贡献者列为我的共同作者?
代码复制了很多来自 openai/improved-diffusion、WuJunde/ MrPrism、WuJunde/ DiagnosisFirst、LuChengTHU/dpm-solver、JuliaWolleb/Diffusion-based-Segmentation、hojonathanho/diffusion、guided-diffusion、bigmb/Unet-Segmentation-Pytorch-Nest -of-Unet,nnUnet, Lucidrains/vit-pytorch
请引用
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu