
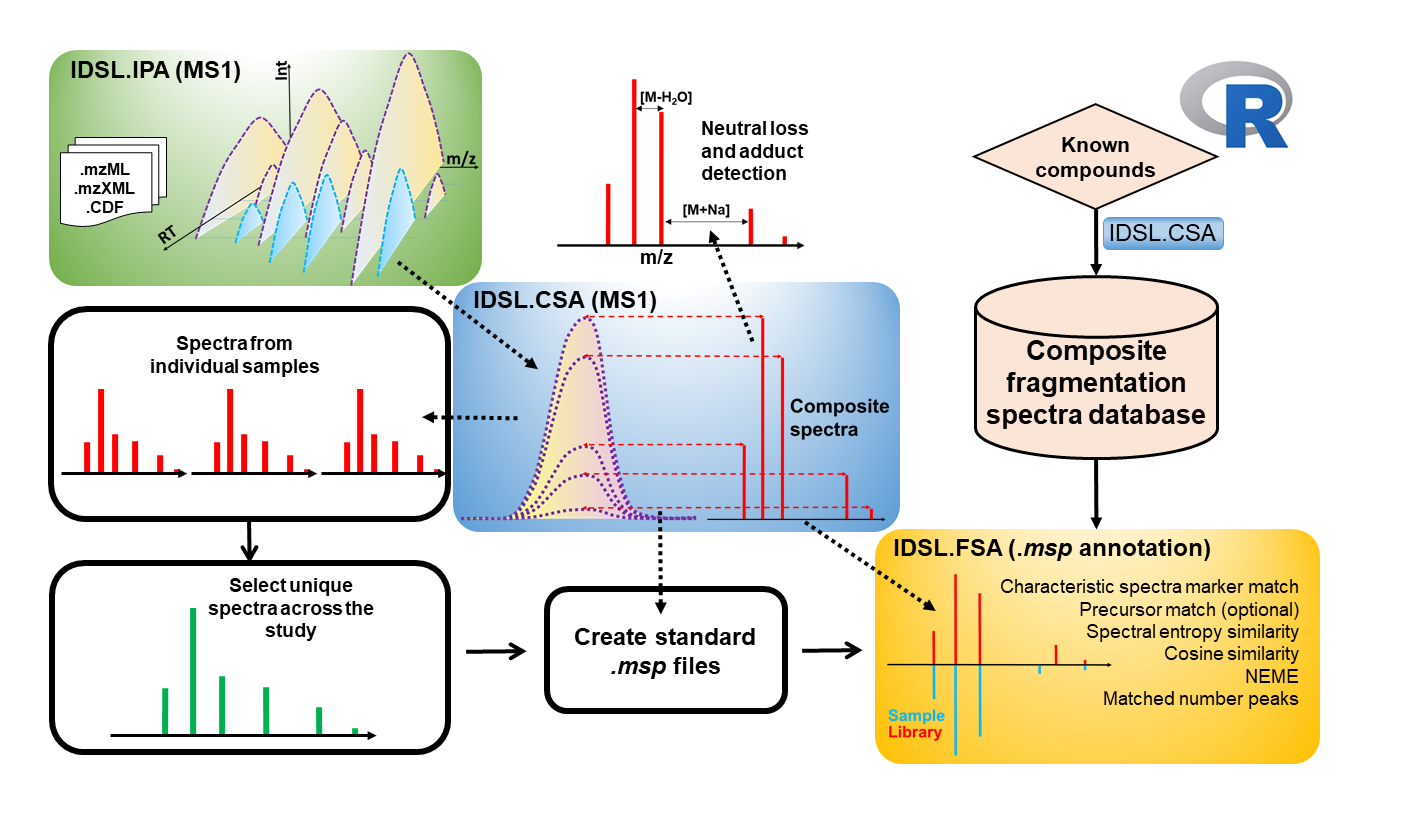
用于质谱数据分析的复合光谱分析(IDSL.CSA) R软件包已由代谢组学的综合数据科学实验室(IDSL.ME)开发了。该软件包可用于通过各种分析方法(例如仅MS1复合光谱反卷积分析( CSA ),数据依赖性获取( DDA )和各种不依赖数据依赖性习得( DIA )在内的各种分析方法获得的破碎光谱的反卷积,包括MS E ,全离子碎片(AIF)和Swath-MS分析。 IDSL.CSA软件包的目的是协助简化数据分析过程并改善代谢组学和Exposomics领域的整体化学结构注释。
Start选项卡中尝试Param0006 。 install.packages("IDSL.CSA")
在处理使用IDSL.CSA工作流程的质谱数据( MZXML , MZML , NETCDF )之前,应使用IDSL.IPA工作流程处理质谱数据,以获取峰( M/Z-RT )的色谱信息。当使用IDSL.IPA工作流生成单个和汇总对齐的峰列的色谱信息时,请下载IDSL.CSA参数电子表格,然后相应地选择参数,然后将此电子表格用作IDSL.CSA Workflow的输入:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
请按照以下步骤进行快速案例研究(n = 33)ST002263,该案例具有Hilic-Esi-POS/NEG模式中收集的Thermo Q Extive HF混合轨道数据。
使用针对IDSL.IPA描述的方法处理原始质谱数据和色谱信息
复合光谱分析需要39个参数分布在5个单独的部分中才能进行全尺度分析。对于本研究,使用IDSL.CSA参数电子表格中介绍的默认参数值。接下来,提供信息
2.1。在Start ”选项卡中为param0001选择“是”,以仅处理CSA工作流程。
2.2。 HRMS数据位置地址(MS1级HRMS数据)的CSA0005
2.3。 CSA0008,用于由IDSL生成的peaklists目录的地址
2.4。 CSA0009用于由IDSL生成的peak_alignment目录的地址
2.5。 CSA0011用于输出位置(.MSP文件和EICS)
2.6。您还可以根据计算能力使用CSA0004增加处理线程的数量
在r/rstudio控制台或终端中运行此命令:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
您可以在为CSA0011提供的地址解析结果。
4.1。 CSA_MSP包含.msp文件
4.2。 CSA_ADDUCT_ANNOTATION包括具有潜在加合物信息的峰列表
4.3。 peak_alignment_subset包括每个CSA群集中主要离子的对齐峰表的子集
4.4。 Aligned_spectra_table包括对齐表上CSA聚合的信息
[1] Fakouri Baygi,S.,Kumar,Y。Barupal,DK IDSL.CSA:无靶向代谢组学数据集化学注释的复合光谱分析。分析化学, 2023,95 (25) ,9480–9487。
[2] Fakouri Baygi,S.,Kumar,Y。Barupal,DK IDSL。 IPA表征了未靶向的LC/HRMS数据集中的有机化学空间。蛋白质组研究杂志, 2022,21 (6) ,1485-1494。