我们很高兴地宣布,磁盘上的索引大小减小了8次,并且在内存中的记忆中只有4次,因为仅移至一种类型的FM索引(2bit.64而不是2bit.64和8bit.32)和8x压缩后缀阵列。例如,对于人类基因组,磁盘上的索引大小从〜80GB降至〜10GB,并且记忆足迹降至〜40GB的〜10GB。由于降低,索引时间的降低大幅下降,几乎没有对读取映射的任何性能影响。由于索引结构的这种变化(在2020年10月10日的提交#4B59796中),您将需要重建索引。
在提交A591E22中的输出SAM文件中添加了MC标志。输出应匹配原始BWA-MEM版本0.7.17。
从提交E0AC59E开始,我们有一个git subsodule safestringlib。要获得它,请在已经克隆的存储库中使用克隆或使用“ Git subpodule Init”和“ Git Subsodule Update”时使用 - 恢复性(有关更多详细信息,请参见下文)。
#使用预编译的二进制文件(推荐)curl -l https://github.com/bwa-mem2/bwa-mem2/releases/download/download/v2.2.1/bwa-mem2-2.2.1_x64-linux.tar.tar.tar.tar.bz2 | TAR JXF- BWA-MEM2-2.2.1_x64-Linux/BWA-MEM2索引参考 BWA-MEM2-2.2.2.1_x64-linux/bwa-mem2 mem ref.fa read1.fq read2.fq> out.sam#compile#commile#compile commile(不推荐给一般用户)#获取sourcegit clone-recursive https https:// https:// github.com/bwa-mem2/bwa-mem2cd bwa-mem2#orgit克隆https://github.com/bwa-mem2/bwa-mem2/bwa-mem2cd bwa-mem2 git subsodule init git子模块更新#编译和runmake ./bwa-mem2
该工具BWA-MEM2是BWA中BWA-MEM算法的下一个版本。它产生的对齐方式与BWA相同,并且根据用例,数据集和运行机器的速度约为1.3-3.1倍。
原始BWA由Heng Li(@LH3)开发。 BWA-MEM2的性能增强主要是由Vasimuddin MD(@Yuk12)和Sanchit Misra(@Sanchit-Misra)从英特尔(Intel)进行的。 BWA-MEM2根据MIT许可证分发。
对于一般用户,建议使用发布页面中的预编译二进制文件。这些二进制文件与英特尔编译器一起编译,并且运行速度比GCC计算的二进制文件更快。预编译的二进制文件还间接支持CPU调度。 bwa-mem2二进制文件可以自动根据运行计算机上可用的SIMD指令集选择最有效的实现。使用以下命令行在CentOS7机器上生成了预编译的二进制文件:
使CXX = ICPC Multi
使用与原始BWA MEM工具完全相同。这是一个简短的概述。为可用命令运行./bwa-mem2。
#索引参考序列(需要28n GB内存,其中n是参考序列的大小)../ bwa-mem2索引[-p prefix] <in.fasta>其中 <in.fasta>是参考序列fasta文件的途径,并且 <prefix>是存储结果索引的文件名称的前缀。默认值为IN.FASTA。#映射#运行“ ./bwa-mem2 mem”获取所有选项。/bwa-mem2mem -t <Num_threads> <prefix> <prefix> <reads.fq/fq/fa>> out.sam 其中<prefix>是创建索引或参考Fasta文件的路径时指定的前缀,以防不提供前缀。
数据集:
参考基因组:human_g1k_v37.fasta
| 别名 | 数据集源 | 阅读数 | 阅读长度 |
|---|---|---|---|
| D1 | 广泛研究所 | 2 x 2.5m bp | 151bp |
| D2 | SRA:SRR7733443 | 2 x 2.5m bp | 151bp |
| D3 | SRA:SRR9932168 | 2 x 2.5m bp | 151bp |
| D4 | SRA:SRX6999918 | 2 x 2.5m bp | 151bp |
机器详细信息:
处理器:Intel(R)Xeon(R)8280 CPU @ 2.70GHz
OS:Centos Linux版本7.6.1810
内存:100GB
我们遵循以下步骤收集绩效结果:
A.数据下载步骤:
从https://trace.ncbi.nlm.nih.gov/traces/sra/sra/sra/sra.cgi?view=softwarewheader-global下载SRA Toolkit
tar xfzv sratoolkit.2.10.5 centos_linux64.tar.gz
下载D2:sratoolkit.2.10.5-centos_linux64/bin/fastq-dump -slit-files srr7733443
下载D3:sratoolkit.2.10.5-centos_linux64/bin/fastq-dump -slit-files srr9932168
下载D4:sratoolkit.2.10.5-centos_linux64/bin/fastq-dump -slit-files srx69999918
B.对准步骤:
git克隆https://github.com/bwa-mem2/bwa-mem2.git
CD BWA-MEM2
make CXX=icpc (使用Intel C/C ++编译器)
或make (使用GCC编译器)
./bwa-mem2索引<ref.fa>
./bwa-mem2 mem [-t <#threads>] <ref.fa> <in_1.fastq> [<in_2.fastq>]> <output.sam>
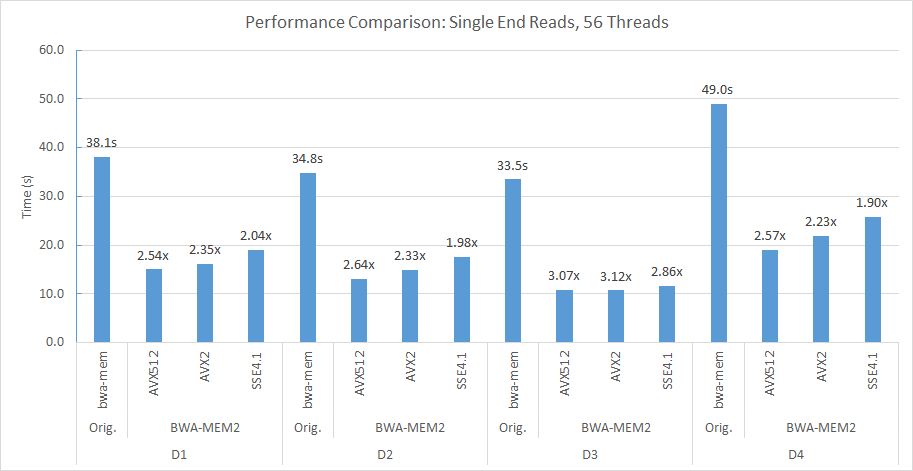
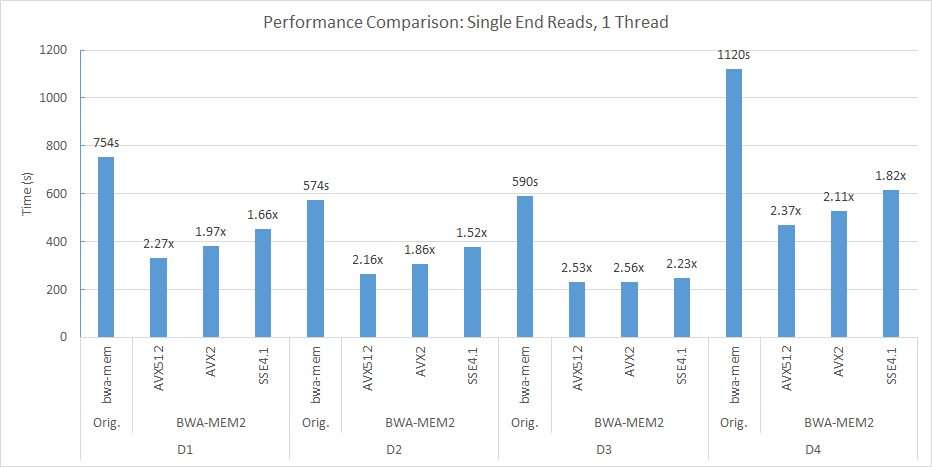
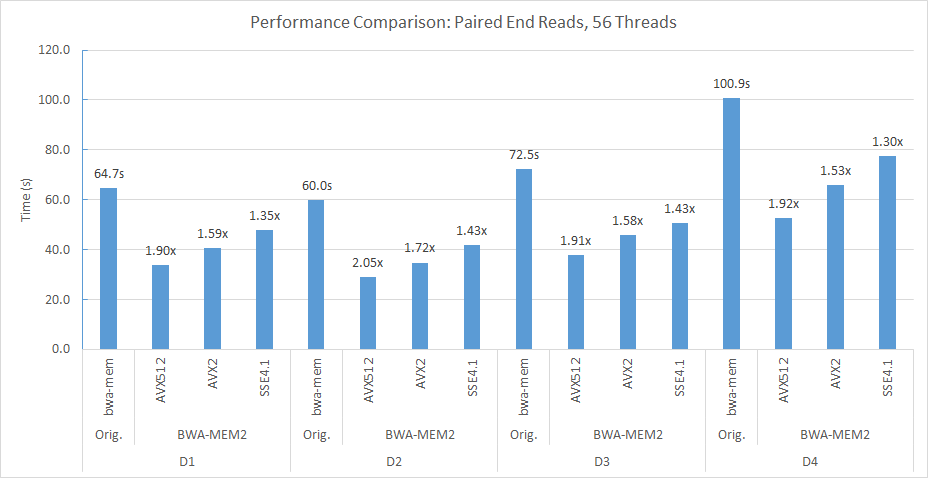
例如,在我们的双套接字(每个56个线程)和Double Numa计算节点中,我们使用以下命令行将D2与Human_G1K_V37.Fasta参考基因组相结合。
numactl -m 0 -C 0-27,56-83 ./bwa-mem2 index human_g1k_v37.fasta numactl -m 0 -C 0-27,56-83 ./bwa-mem2 mem -t 56 human_g1k_v37.fasta SRR7733443_1.fastq SRR7733443_2.fastq > d2_align.sam

BWA-MEM2-LISA是BWA-MEM2的加速版,我们将学习索引应用于播种阶段。 BWA-MEM2-LISA分支包含实现的源代码。以下是BWA-MEM2-LISA的特征:
与BWA-MEM2完全相同。
创建索引和读取映射的所有命令线与BWA-MEM2完全相同。
与BWA-MEM2相比,BWA-MEM2-LISA可加速播种阶段(BWA-MEM2中的主要瓶颈之一)。
对于人基因组,BWA-MEM2-LISA指数的记忆足迹约为120GB。
该代码存在于BWA-MEM2-LISA分支:https://github.com/bwa-mem2/bwa-mem2/bwa-mem2/tree/bwa-mem2-lisa
BWA-MEM2存储库的ERT分支包含基于BWA-MEM2的基于辐射树的加速度的代码库。 ERT代码构建在BWA-MEM2的顶部(这要归功于 @arun-sub的辛勤工作)。以下是基于ERT的BWA-MEM2工具的亮点:
与BWA-MEM完全相同(2)
该工具还有两个附加标志来启用ERT解决方案(用于创建索引和映射),否则它以Vanilla BWA-MEM2模式运行
它使用1个额外标志来创建ERT索引(与BWA-MEM2索引不同)和1个使用该ERT索引的附加标志(请参阅ERT Branch的读数)
与Vanilla BWA -MEM2相比,ERT解决方案更快10%-30%(在上述机器配置上测试) - 建议用户使用选项-K 1000000来查看Speedups
ERT索引的记忆脚打印〜60GB
该代码存在于ERT分支:https://github.com/bwa-mem2/bwa-mem2/tree/ert/ert
Vasimuddin MD,Sanchit Misra,Heng Li,Srinivas Aluru。 BWA-MEM的有效体系结构与多核心系统的加速度。 IEEE平行和分布式处理研讨会(IPDPS),2019。10.1109/ipdps.2019.00041