
Isling是在配对末端读取中检测病毒或向量积分的工具。请阅读我们的论文以获取完整的详细信息。
如果您安装了conda和snakemake ,请在本地使用(随附的)测试数据运行:
git clone https://github.com/aehrc/isling.git && cd isling
snakemake --configfile test/config/test.yml --cores <cores> --use-conda
如果您安装了snakemake和singularity ,则可以使用:
snakemake --configfile test/config/test.yml --cores <cores> --use-singularity
另外,如果您安装了Docker,则可以在MacOS上运行:
docker run --rm -it -v"$(pwd)"/out:/opt/isling/out szsctt/isling:latest snakemake --configfile test/config/test.yml --cores 1
这将使用容器内的配置文件和数据,结果将出现在当前工作目录中out的文件夹中。在Linux上,您需要将此命令作为root运行,在Windows上,您需要调整绑定安装语法( -v参数)。
输入数据(读取和主机和病毒引用)已指定为ni a config文件 - 对于您自己的数据,您需要修改示例配置文件( test/config/test.yml ),以指向您自己的数据。有关配置文件格式的更多信息,请参见configfile.md 。
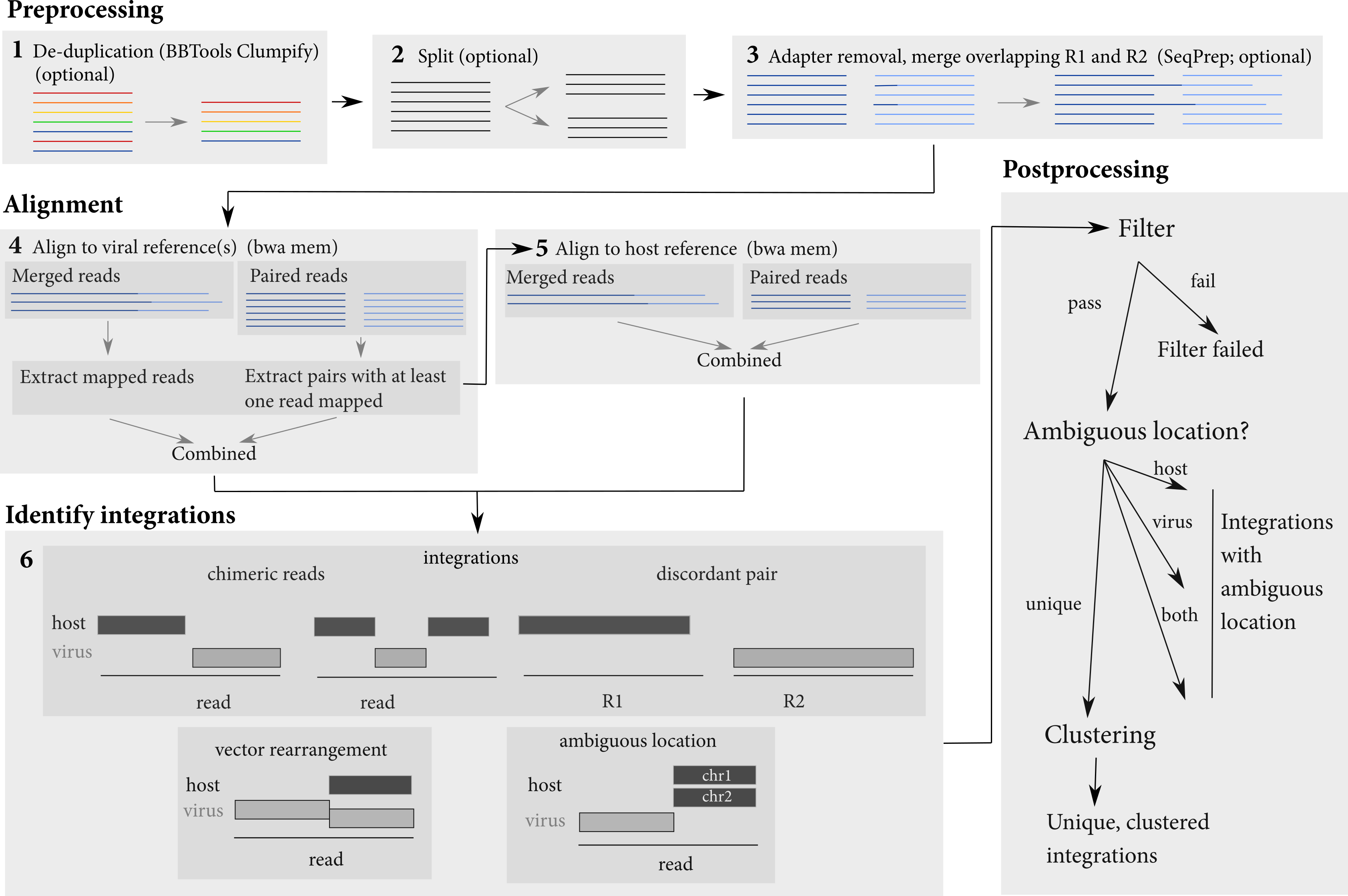
管道执行多个步骤以识别集成站点。它作为由FASTQ文件或BAM文件组成的输入数据集。它对读取(合并重叠读数,可选)进行了一些预处理,然后将它们对齐到宿主和病毒序列。首先将读取与病毒序列对齐,然后将对齐的读数提取并与宿主排列。这些比对用于识别病毒整合。
ISLING需要snakemake和singularity (推荐)或conda来提供依赖性。额外的, python版本3.5或更高版本以及pandas (如果使用conda安装snakemake ,则应自动安装这些版本。
Alternativley,使用包含Isling和所有依赖项的Docker版本。
所需的输入是配置文件,该文件指定主机和病毒/向量引用,并且需要读取。在配置文件中指定所有输入。 ISLING当前仅适用于配对读取。
有关此配置文件格式的说明,请参见文件configfile.md 。
ISLING输出集成站点在配置文件中指定的输出目录中以选项卡分隔格式。
在输出文件夹中,为配置文件中的每个数据集创建一个文件夹,对于每个数据集,可以在ints目录中找到集成。每个示例将有一组输出文件。
对于每个示例,都有许多输出文件,对于特定用例可能会很感兴趣。
<sample>.<host>.<virus>.integrations.txt :有关所有检测到的连接的信息,无论他们是否通过过滤器<sample>.<host>.<virus>.integrations.post.txt :有关通过所有过滤器的检测到的连接的信息<sample>.<host>.<virus>.integrations.post.unique.txt :有关通过所有过滤器的检测到的连接的信息,并且在主机和向量/病毒中都有明确的位置<sample>.<host>.<virus>.integrations.post.unique.merged.txt :在主机和病毒/载体中合并重叠连接或与相同坐标的重叠连接或连接的结果。合并仅使用在宿主和病毒/载体中都有明确位置的集成连接进行进行合并。<sample>.<host>.<virus>.integrations.post.host_ambig.txt :有关通过所有过滤器并具有明确的位置向量/病毒但在主机中具有含糊位置的检测到的连接的信息<sample>.<host>.<virus>.integrations.post.virus_ambig.txt :有关通过所有过滤器并具有明确位置主机但在矢量/病毒中模棱两可的位置主机但位置模棱两可的检测到的信息的信息<sample>.<host>.<virus>.integrations.post.both_ambig.txt :有关通过所有过滤器的检测到的连接的信息输出文件给出了已确定的集成及其属性的位置。根据其模棱两可的基础,指定了集成连接的坐标。也就是说,读物的宿主和病毒部分之间通常存在差距或重叠: 
由于在这种情况下不能唯一确定积分的位置,因此ISLING输出这些碱基的坐标和载体/病毒基因组作为整合的位置。
除了合并的群集输出文件外,所有文件都包含以下列:
gap或overlap (在同一读取中识别出的主机和病毒部分),如果没有模棱两可的基础,则连接很clean 。另外,如果在不一致的对中识别出Jucntion, OverlapType是discordanthv )或病毒/宿主( vh )的方向+或-bwa )bwa )OverlapType为gap则含糊的基础数chimeric读物中观察到的连接,是discordant一对还是一个short集成,在同一读取中都观察到两个连接bwa )bwa )'xxx'分开要重现Isling手稿中的数字,请参见benchmarking目录中的读数。