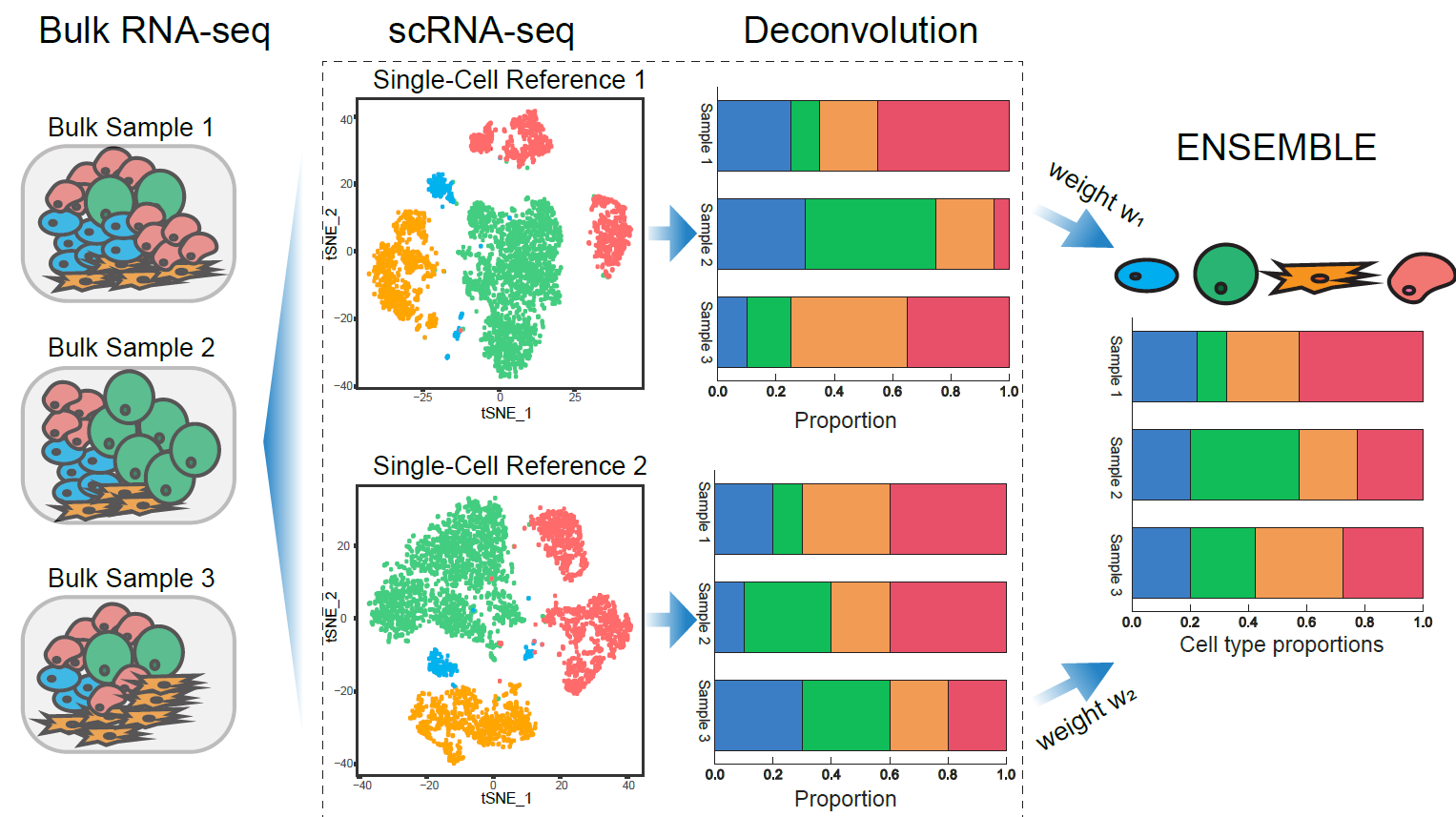
SCDC是用于散装RNA-Seq的反卷积方法,它利用来自多个SCRNA-SEQ参考数据集的细胞类型特定基因表达式。 SCDC采用了一种集成方法来整合来自不同实验室和不同时间中产生的不同SCRNA-SEQ数据集的反卷积结果,并隐式地解决了批处理效应的混杂。
Meichen Dong,Aatish Thennavan,Eugene Urrutia,Yun Li,Charles M Perou,Fei Zou,Yuchao Jiang,SCDC:由多个单单细胞RNA测序参考,BBZ166,HTTPS的BioInformatics,BioInformant,BioInformants in Botuers单细胞RNA测序参考,散装基因表达卷积。 org/10.1093/bib/bbz166
许可证:麻省理工学院
您可以使用以下方式从GitHub安装已发布的SCDC版本
if(!quired(“ devtools”)){
install.packages(“ devtools”)
} devtools :: install_github(“ meichendong/scdc”)有关“ XBIOC”的依赖项问题问题可以通过:
install.packages(“远程”)远程:: install_github(“ renozao/xbioc”)
请参阅“小插图”页面。
SCDC论文在生物信息学的简报上发表。
有关包裹的问题可以通过电子邮件发送至:[email protected]
当单元数据集中只有“一个主题/个人”时,请使用SCDC_qc_ONE() , SCDC_prop_ONE()函数。
可能影响反卷积结果的方面:
数据格式:批量和单细胞样品是否都是原始计数 /相同格式?我们希望数据格式保持一致和可比性。
基因过滤:您是否过滤降低表达的基因 /核糖体基因 /线粒体基因?这些基因可能会影响下游分析。
单元格的大小和库的大小因素:对于单个单元格,您认为所有基因计数(库大小)的总和可以反映其实际细胞大小吗?这是我们的假设之一:细胞类型之间的库大小的比率可以反映细胞类型之间的真实细胞大小的比率。如果没有,则可以在构造“基矩阵”时手动输入单元格因子。
类似的细胞类型:是否存在可能混淆分析的细胞类型?例如,具有非常相似的曲线 /标记基因的细胞类型。
缺少主要的细胞类型 /技术问题:您是否期望测序程序在散装和SC上会产生很大的影响,甚至技术是相同的?有时,单细胞参考数据可能会丢失某些单元类型的信息。例如,您的批量样品中有脂肪细胞,但是以某种方式您没有单细胞数据。
使用单个参考数据集进行反卷积:您是否尝试使用一个参考数据集来测试结果一般是否有意义?我看到你尝试了浓汤。您是否尝试过诸如Cibersortx之类的其他方法?如果其他“单一引用”反卷积方法的结果更有意义,那么您可以使用我们的合奏步骤直接输入这些方法。