最後更新:2024 年 9 月 30 日
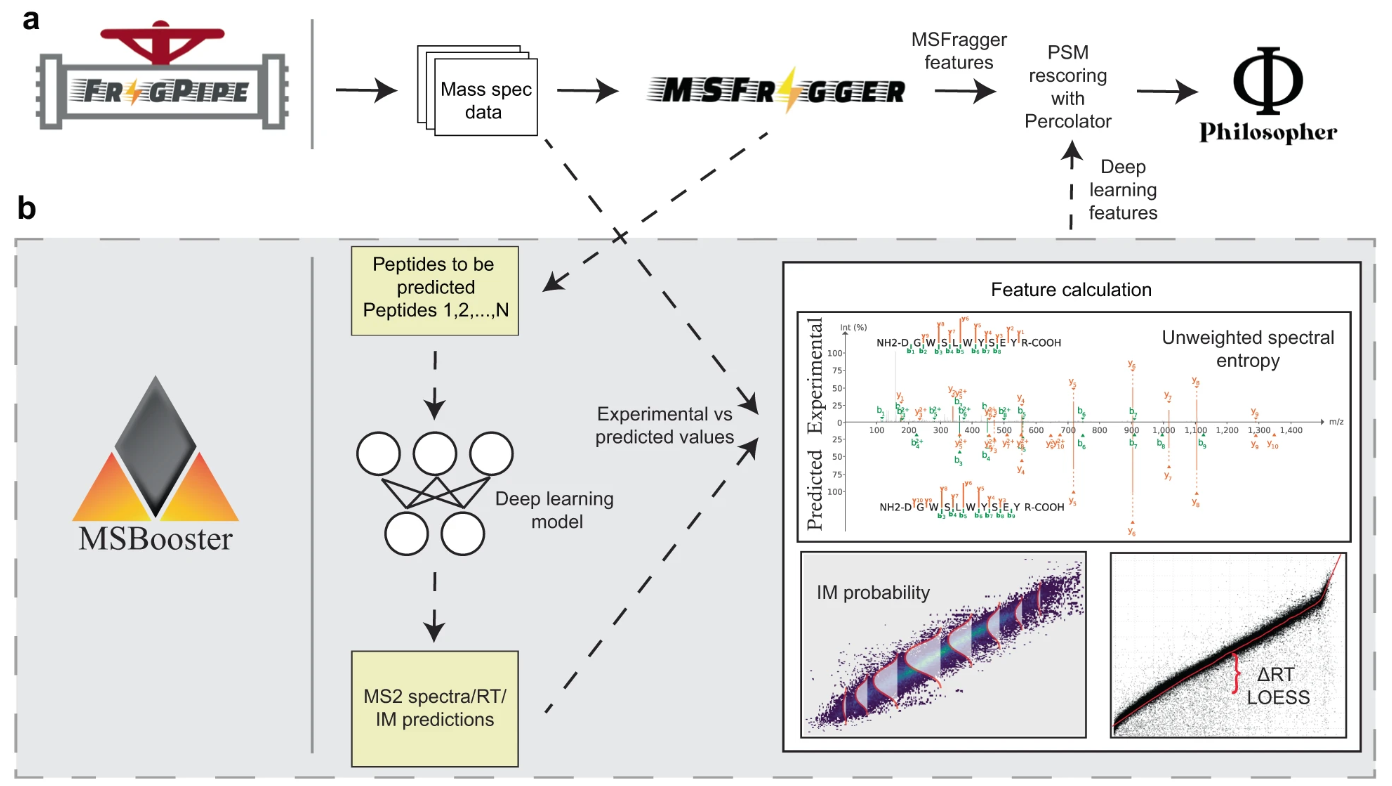
MSBooster 是一種工具,用於將譜庫預測納入自下而上串聯液相層析質譜蛋白質組學數據中的勝肽譜匹配 (PSM) 重新評分中。大致分為4步:
從搜尋結果中的 PSM 中提取勝肽,並格式化機器/深度學習 (ML/DL) 預測器的輸入文件
調用預測模型並保存輸出
特徵計算
在搜尋結果檔案中新增功能
MSBooster 與多種類型的資料庫搜尋相容,包括 HLA 免疫勝肽組學、DDA 和 DIA 以及單細胞蛋白質組學。它被合併到 FragPipe 中並包含在其許多工作流程中。 MSBooster 是在開發時考慮了其他 FragPipe 工具,例如 FragPipe-PDV。
MSBooster 可以處理多種輸入檔格式和模型:
| 質譜儀輸出 |
|---|
| .mzML |
| .mgf |
| PSM檔案 |
|---|
| 。 |
| .pepXML(正在進行中) |
| 預測模型 |
|---|
| DIA-NN |
| 科伊納模型 |
MSBooster可以在Windows和Linux系統中運作。如果使用FragPipe,除了安裝FragPipe外,不需要其他安裝步驟。 MSBooster 位於「驗證」標籤中。選擇啟用「預測 RT」的保留時間功能和「預測光譜」的 MS/MS 光譜功能。有關如何運行分析的信息,請參閱 FragPipe 文件。
如果使用獨立的 MSBooster 在命令列中執行,請從 Releases 下載最新的 jar 檔案。 MSBooster 也需要 DIA-NN 進行 MS/MS 和 RT 預測。請安裝 DIA-NN 並記下 DIA-NN 執行檔的路徑(例如 Windows 中的 DiaNN.exe,Linux 中的 diann-1.8.1.8)。
您可以使用類似於以下內容的命令來執行 MSBooster:
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
需要傳遞的最少參數是:
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
雖然您可以單獨傳遞這些參數,但在 paramsList 檔案的每一行上放置一個參數會更容易。請參閱 msbooster_params.txt 取得範本。
以下參數僅供一般使用。 Koina 特定參數位於 Koina 文件中
paramsList (String) :包含此運行參數的文字檔案的位置
fragger (String) :MSFragger 運行中的 fragger.params 檔案的檔案路徑。 MSBooster將讀入多個參數並根據它們調整內部參數,例如碎片質量誤差容限和質量偏移
outputDirectory (String) :輸出新檔案的位置
editedPin (String) :MSBooster 將根據提供的檔案命名新檔案。例如,A.pin 將有一個名為 A_edited.pin 的對應項。若要變更預設的“已編輯”,請在此處提供新字串
renamePin (int) :是否產生新的 pin 檔案或重寫舊的 pin 檔案。這裡預設是1,不會覆蓋。設定為 0 將覆蓋舊的 pin 文件
deletePreds (boolean) :成功運行後是否刪除儲存模型預測的檔案。預設情況下,設定為 false。如果您想刪除這些,請設定為 true
loadingPercent (int) :使用進度報告器報告任務進度的頻率。預設情況下,設定為 10,表示每 10% 列印一次更新。
numThreads (int) :要使用的執行緒數。預設為 0,即使用所有可用執行緒減 1
splitPredInputFile (int) :僅在 DIA-NN 預測因記憶體不足錯誤而失敗時使用 (137)。預設情況下,設定為 1,但您可以增加此值以指定 DIA-NN 輸入檔應分解為多少個較小的檔案。然後將按順序預測每個文件,減輕記憶體負擔
plotExtension (String) :繪圖應採用什麼檔案格式。
features (String) :要計算的特徵清單。區分大小寫,以逗號分隔,中間沒有空格。預設為“predRTrealUnits,unweightedSpectralEntropy,deltaRTLOESS”
spectraPredFile (String) :如果您要重複使用舊的光譜預測(例如來自 DIA-NN 或 Koina),您可以在此處指定檔案位置
RTPredFile (String) :與spectrumPredFile相同,但用於RT預測
IMPredFile (String) :與 SpectrumPredFile 相同,但用於 IM 預測
spectraModel (String) :使用哪個光譜預測模型
rtModel (String) :與spectrumModel相同,但用於RT
imModel (String) :與 SpectrumModel 相同,但用於 IM
useSpectra (boolean) :是否使用基於光譜預測的特徵。預設為 true
useRT (boolean) :是否使用基於 RT 預測的特性。預設為 true
useIM (boolean) :是否使用基於 IM 預測的功能。預設為 false
ppmTolerance (float) :片段錯誤 ppm 容差(預設 20ppm)
matchWithDaltons (boolean) : 是否符合預測和觀察到的片段(以道爾頓為單位)(預設為 false)
DaTolerance (float) :在預測峰周圍有多少道爾頓來尋找實驗峰(預設為 0.05)
useTopFragments (boolean) :是否將光譜預測過濾到 N 個最高強度峰值(預設為 true)
topFragments (int) :最多應使用多少個預測片段進行特徵計算(預設為 20)。僅當 useTopFragments 為 true 時才應用
removeRankPeaks (boolean) :預設為 true,一旦匹配,就會從實驗光譜中過濾掉片段。如果為 false,則實驗片段可以透過同一掃描中的多個 PSM 進行匹配
useBasePeak (boolean) :是否應將下限應用於 MS2 預測,以僅使用強度較高的片段(預設為 true)
percentBasePeak (float) :相似性計算中包含強度為基峰強度某一百分比的片段的百分比。僅當 useBasePeak 為 true 時才套用(預設 1)
loessEscoreCutoff (float) :用於收集 PSM 以進行 RT/IM 校準的第一遍的期望值截止值。預設值為 10^-3.5,或約 0.000316
rtLoessRegressionSize (int) :用於 RT LOESS 校準的 PSM 最大數量(預設為 5000)
imLoessRegressionSize (int) :與 rtLoessRegressionSize 相同,但用於 IM(預設 1000)
minLoessRegressionSize (int) :嘗試 LOESS RT/IM 校準所需的最小 PSM 數量(預設 100)。如果可用的 PSM 數量少於此數量,則使用線性迴歸
minLinearRegressionSize (int) :嘗試線性迴歸 RT/IM 校準所需的最小 PSM 數量(預設 10)。如果可用的 PSM 數量少於此數量,則不會嘗試校準
loessBandwidth (String) :嘗試進行 RT/IM LOESS 校準的頻寬清單(預設為 0.01,0.05,0.1,0.2)。必須以逗號分隔,中間不能有空格
regressionSplits (int) :用於 RT/IM LOESS 校準的交叉驗證數量(預設 5)
massesForLoessCalibration (String) :質量偏移的品質應適合其自己的校準曲線。列表以逗號分隔,中間沒有空格。質量數應寫入與 PIN 檔案中相同的位數
loessScatterOpacity (float) : LOESS 校準圖中散佈圖的不透明度,從 0 到 1(預設為 0.35)
.pin 檔案具有新功能。預設情況下,將產生以“_edited.pin”結尾的新 pin 檔案。使用的預設特徵是“unweighted_spectral_entropy”、“delta_RT_loess”和“pred_RT_real_units”。如果啟用離子淌度功能,還將包括“delta_IM_loess”和“ion_mobility”
SpectrumRT.tsv 和 SpectrumRT_full.tsv:DIA-NN 預測模型的輸入文件
SpectrumRT.predicted.bin:包含 DIA-NN 預測的二進位文件,MSBooster 將使用該文件進行特徵計算。如果使用 FragPipe-PDV,這些檔案用於產生實驗和預測光譜的鏡像圖
MSBooster 產生多個圖表,可用於進一步檢查資料與模型預測的比較情況。
MSBooster_plots 資料夾:
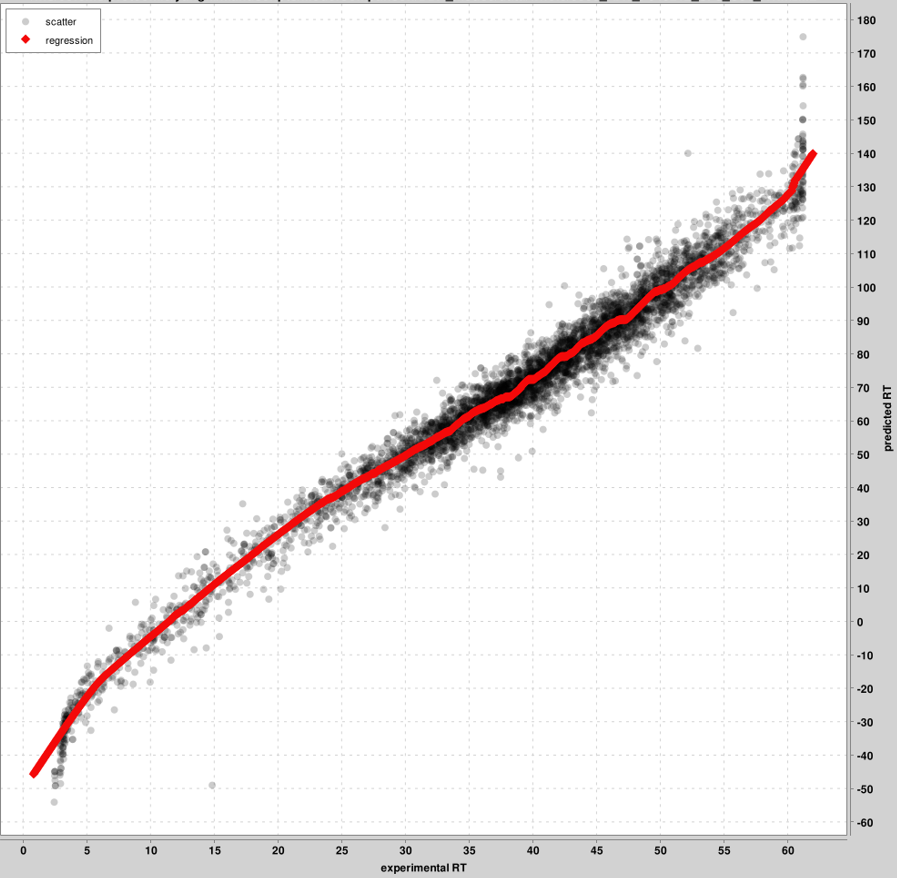
RT_calibration_curves:最多 5000 個 PSM 將用於實驗和預測 RT 尺度之間的校準。圖中顯示了這些頂級 PSM,而不是所有 PSM。每個 pin 檔案將產生一張圖表
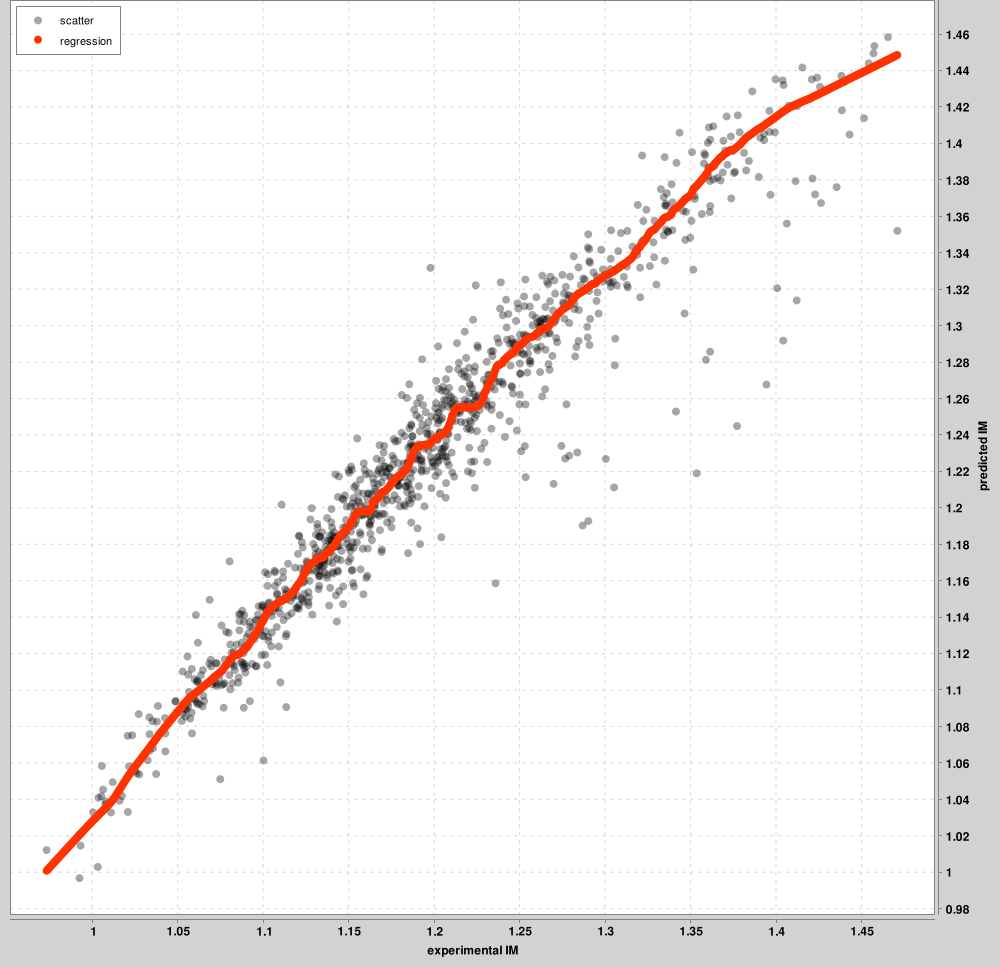
IM_calibration_curves:最多前 1000 個 PSM 將用於實驗和預測 IM 尺度之間的校準。圖中顯示了這些頂級 PSM,而不是所有 PSM。將為每個充電狀態學習一條單獨的曲線。下圖是電荷2前驅體的範例
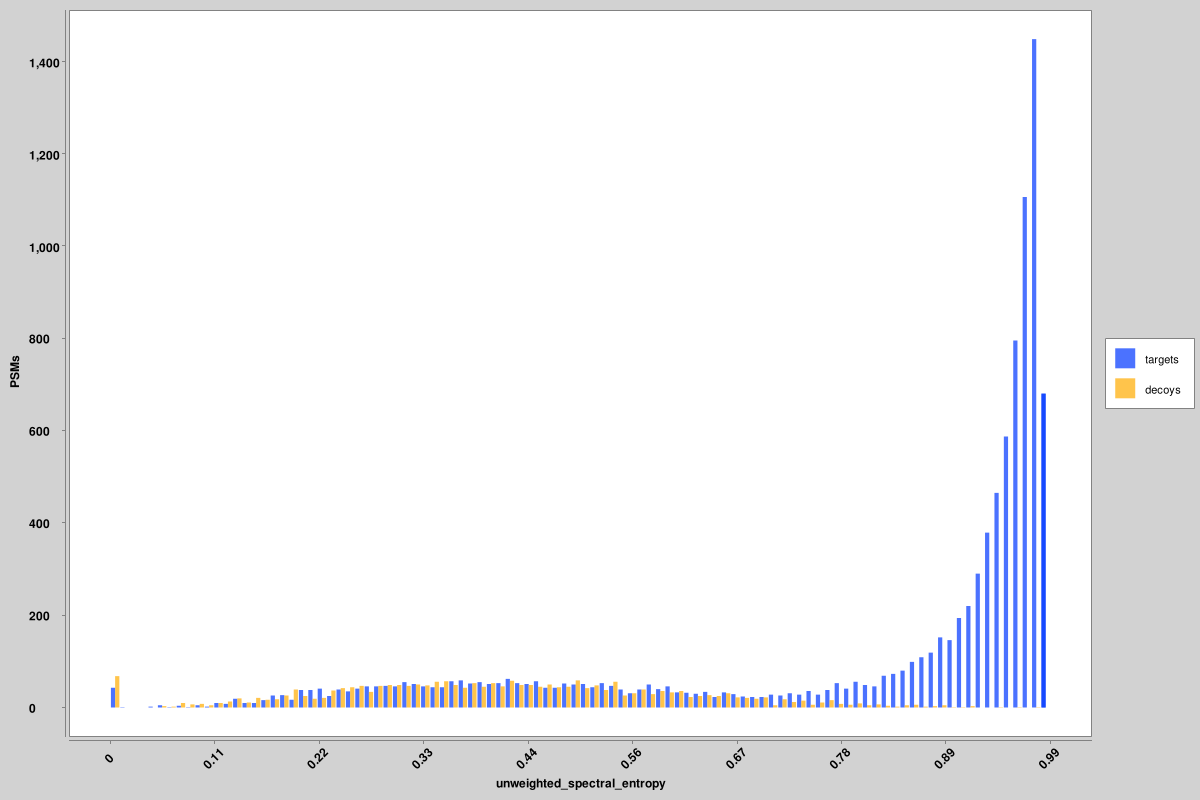
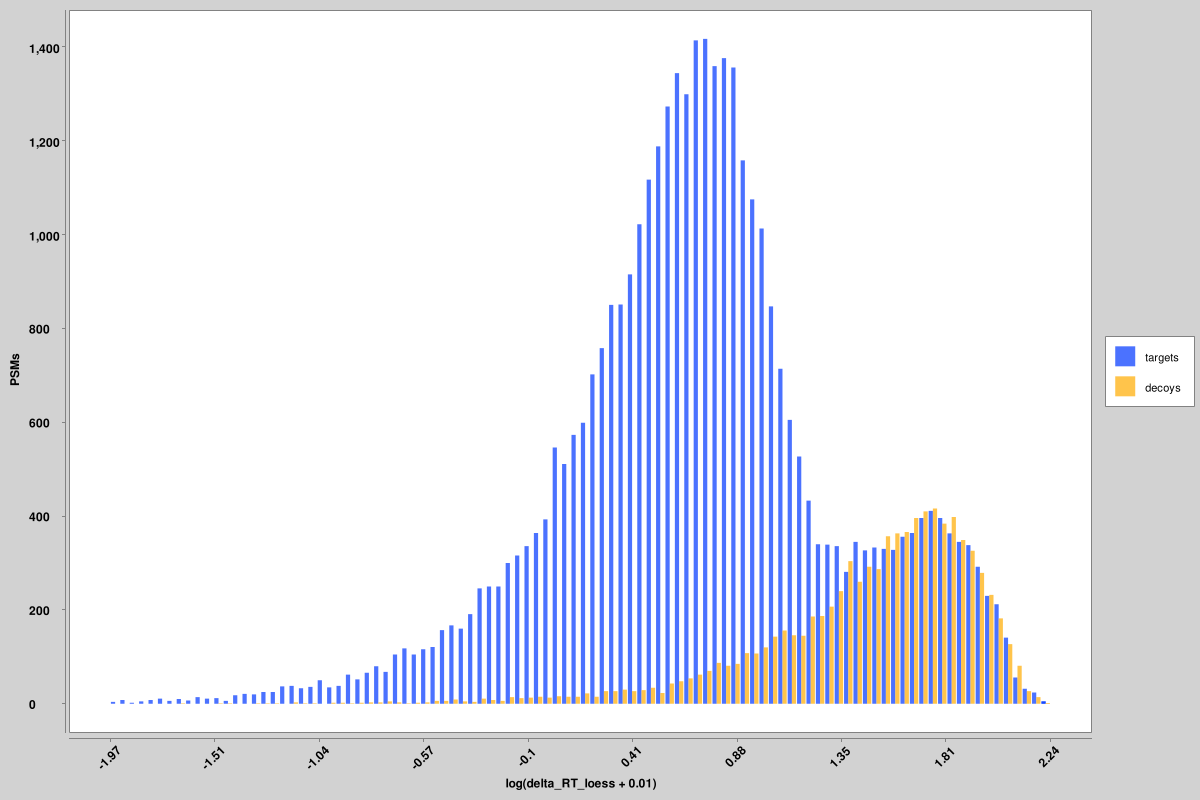
Score_histograms:每個 pin 檔案的所有目標和誘餌 PSM 的疊加直方圖。這裡以對數刻度繪製了一些特徵,以便更好地視覺化真值和誤報的雙峰分佈,但原始值是 pin 檔案中使用的值,而不是對數刻度版本。這裡顯示的是未加權譜熵和 delta RT 分數的直方圖,但所有特徵都會產生類似的直方圖
使用 Koina 的勝肽預測模型進行 MSBooster 特徵產生:https://fragpipe.nesvilab.org/docs/tutorial_koina.html
透過 MGF 檔案讀取任何模型的預測
有關所有允許的功能以及如何使用圖形輸出對其進行品質控制的文檔
使用 MSBooster 時請引用以下內容:https://www.nature.com/articles/s41467-023-40129-9