這是「一個模型來統治一切:透過文字提示實現醫學影像的通用分割」的官方儲存庫
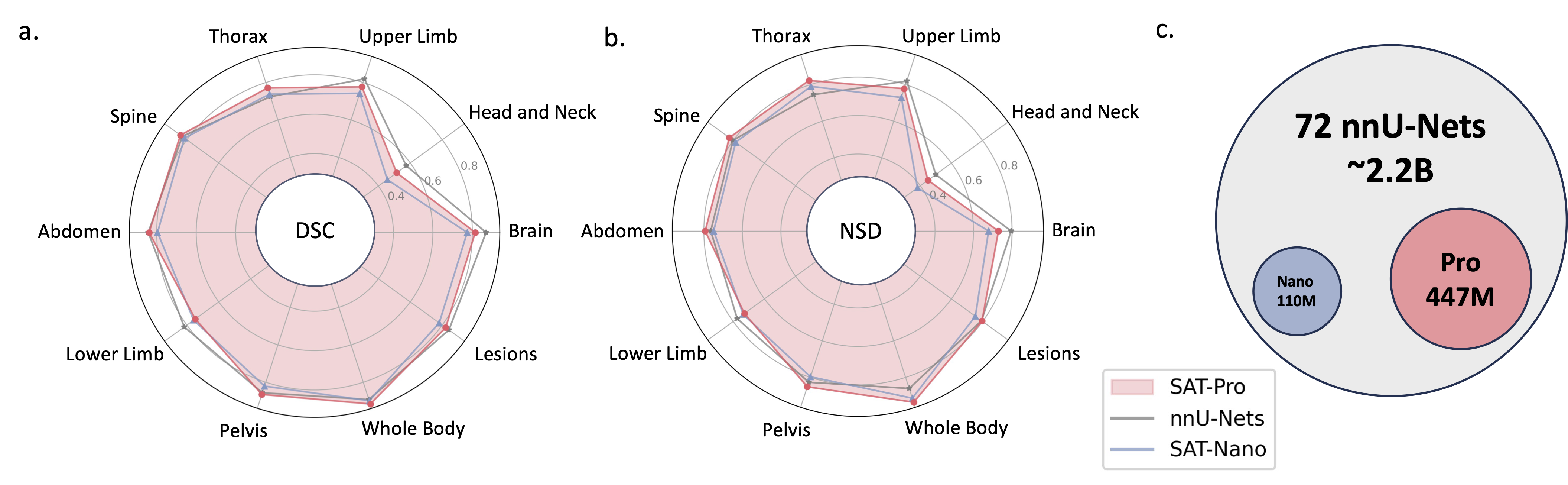
它是一個建立在前所未有的資料收集(72 個公共3D 醫學分割資料集)之上的知識增強型通用分割模型,可以根據文本(解剖學)提示,對來自3 種不同模式(MR、CT、PET )和8 個人體區域的497 個類別進行分割術語)。
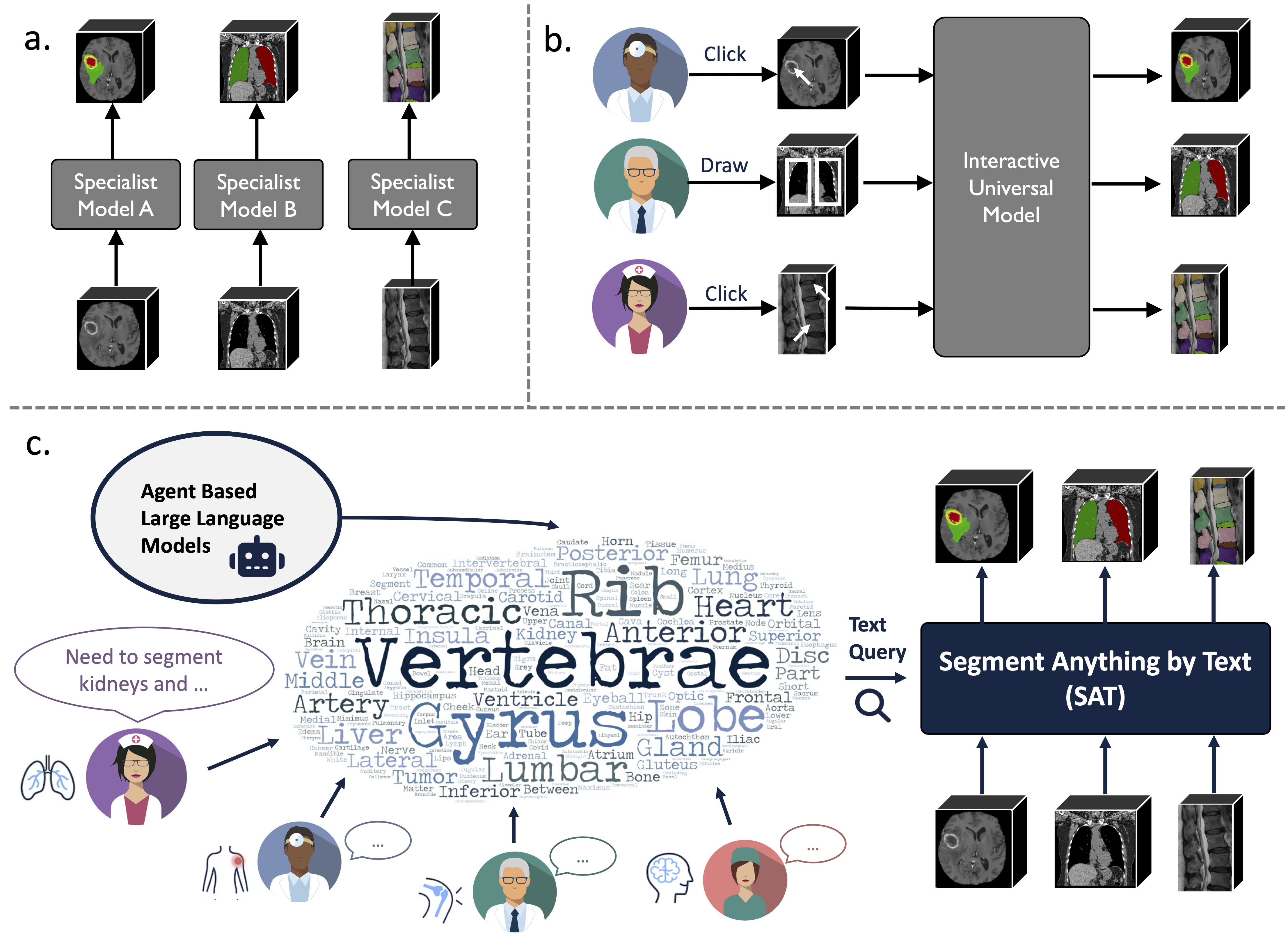
它比訓練和部署一系列專業模式更強大、更有效率。在我們的網站或報紙上查找更多資訊。
2024.08?基於SAT和大語言模型,我們建構了一個全面的、大規模的、區域引導的3D胸部CT判讀資料集。它包含 196 個類別的器官級分割和多粒度報告,其中每個句子都基於相應的分割。在“hushingface”上檢查一下。
2024.06?我們已經發布了建構SAT-DS 的程式碼,它是72 個公共分割資料集的集合,包含超過22K 3D 影像、302K 分割遮罩和來自3 種不同模式(MRI、CT、PET)和8 個人區域的497 個類別。我們還提供 42/72 資料集的快捷下載鏈接,這些資料集經過我們的預處理和打包,以方便您下載和提取後立即使用。檢查此存儲庫以獲取詳細資訊。
2024.05?我們訓練了新版本的 SAT,模型尺寸更大( SAT-Pro ),資料集更多( 72 ),現在支援497 個類別!我們也更新了 SAT-Nano,並基於不同的視覺主幹(U-Mamba 和 SwinUNETR)和文字編碼器(MedCPT 和 BERT-Base)發布了 SAT-Nano 的一些變體。有關此更新的更多詳細信息,請參閱我們的新論文。
U-Net的實作依賴於一個定製版本的dynamic-network-architectures,安裝它:
cd model
pip install -e dynamic-network-architectures-main
其他一些關鍵要求:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
如果您想要 SAT-Nano 的 U-Mamba 變體,您還需要安裝mamba_ssm
S1。依照requirements.txt建置環境。
S2。從 Huggingface 下載 SAT 和文字編碼器的檢查點。
S3。準備 jsonl 檔案中的資料。檢查data/inference_demo/demo.jsonl中的示範。
每個要分割的樣本都需要image (圖像路徑)、 labe (分割目標名稱)、 dataset (樣本所屬的資料集)和modality (ct、mri 或 pet)。 SAT 支援的模式和類別可以在本文的表 12 中找到。
orientation_code (方向)預設為RAS ,適合大多數軸面影像。對於矢狀面中的影像(例如脊椎檢查),將其設定為ASR 。輸入影像應具有H,W,D形狀。 demoprocessed_data中可以找到兩個處理成功的影像,請確保正確完成歸一化以確保 SAT 的效能。
S4。用 SAT-Pro 開始推理?
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d是輸入影像區塊的批次大小,需要根據 GPU 記憶體進行調整(請參閱下表); --max_queries建議設定大於推理資料集中的類,除非你的 GPU 記憶體非常有限;
| 模型 | 批量大小_3d | 顯存 |
|---|---|---|
| 衛星專業版 | 1 | 〜34GB |
| 衛星專業版 | 2 | 〜62GB |
| SAT-奈米 | 1 | 〜24GB |
| SAT-奈米 | 2 | 〜36GB |
S5。檢查--rcd_dir的輸出。結果按資料集組織。對於每種情況,都會找到輸入影像、聚合分割結果和包含每個類別分割的資料夾。所有輸出都儲存為 nifiti 檔案。您可以使用 ITK-SNAP 將它們視覺化。
如果您想使用在 72 個資料集上訓練的 SAT-Nano,只需將--vision_backbone修改為“UNET”,並相應地更改--checkpoint和--text_encoder_checkpoint即可。
對於其他 SAT-Nano 變體(在 49 個資料集上訓練):
UNET-我們的:設定--vision_backbone 'UNET'和--text_encoder 'ours' ;
UNET-CPT:設定--vision_backbone 'UNET'和--text_encoder 'medcpt' ;
UNET-BB:設定--vision_backbone 'UNET'和--text_encoder 'basebert' ;
UMamba-CPT:設定--vision_backbone 'UMamba'和--text_encoder 'medcpt' ;
SwinUNETR-CPT:設定--vision_backbone 'SwinUNETR'和--text_encoder 'medcpt' ;
開始訓練前的一些準備:
sh/中的 slurm 腳本來啟動訓練過程。以SAT-Pro為例: sbatch sh/train_sat_pro.sh
這還需要在此存儲庫之後建立測試資料。您可以參考 slurm 腳本sh/evaluate_sat_pro.sh來啟動評估過程:
sbatch sh/evaluate_sat_pro.sh
如果您將此程式碼用於您的研究或項目,請引用:
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}