該項目包括對總體進行抽樣的不同方法以及對不同方法的評估。我們納入了許多可能導致基於樣本的感染水平估計偏差的情況,包括無應答者、假陽性/陰性率、患者在感染期間的傳播能力。基於EpiABM模型,該軟體包還可以透過執行疾病傳播模擬來輸出最佳採樣方法,以查看每種採樣方法的預測誤差。
EpiOS 尚未在 PyPI 上使用,但該模組可以在本地 pip 安裝。應先將該目錄下載到本機計算機,然後可以使用以下命令進行安裝:
pip install -e .我們也建議您安裝EpiABM模型來產生感染模擬的數據。您可以先將 pyEpiabm 下載到電腦上的任何位置,然後可以使用以下命令進行安裝:
pip install -e path/to/pyEpiabm 可以透過上述docs徽章存取文件。
這是我們專案的 UML 類別圖: 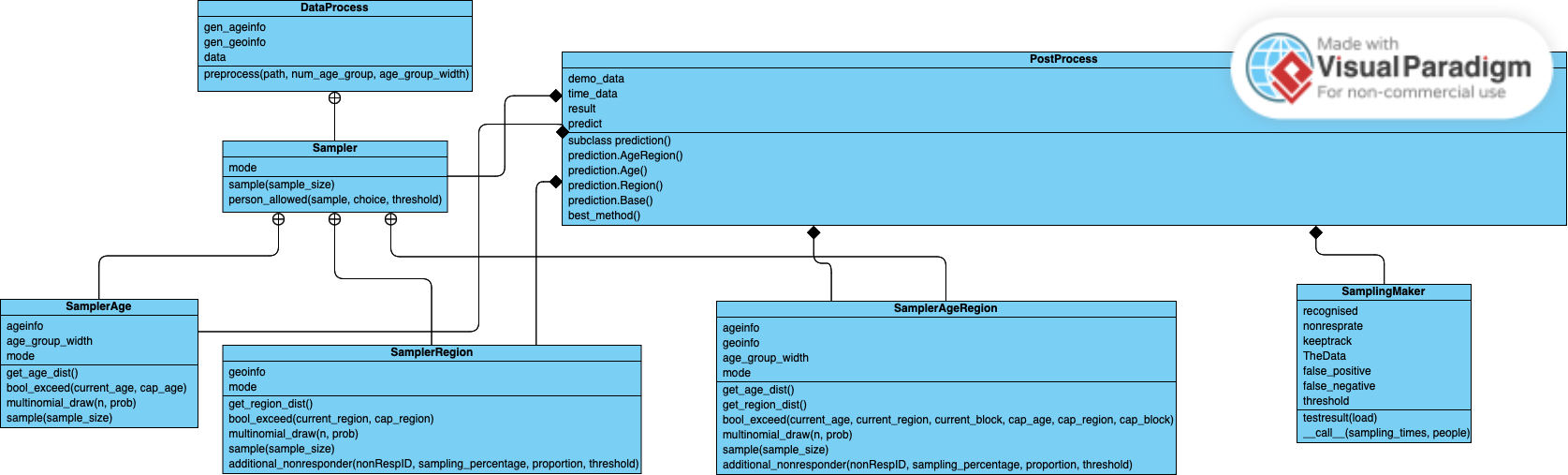
params.py檔案包含該模型所需的所有參數。另外, input資料夾中的檔案是資料預處理過程中產生的臨時檔案的範例。它將由採樣器類別使用。每個採樣器類別中的data_store_path參數是儲存這些檔案的路徑。
PostProcess產生繪圖首先,您需要定義一個新的PostProcess物件並輸入 pyEpiabm 產生的人口統計資料demodata和感染資料timedata 。其次,您可以使用PostProcess.predict根據不同的取樣方法進行預測。可以直接呼叫你想要使用的採樣方法作為方法;然後指定採樣的時間點和樣本大小。這裡,我們將使用AgeRegion作為採樣方法, [0, 1, 2, 3, 4, 5]作為採樣時間點, 3為樣本大小。最後,您可以透過指定參數non_responder和comparison來指定是否要考慮非回應者以及是否要將結果與真實資料進行比較。
對於程式碼範例,您可以看到以下內容:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)現在,您的圖形將保存到給定的路徑!
PostProcess選擇最佳採樣方法首先,您需要定義一個新的PostProcess物件並輸入 pyEpiabm 產生的人口統計資料demodata和感染資料timedata 。其次,您可以使用PostProcess.best_method來比較不同取樣方法的效能。您可以提供您想要比較的方法;然後指定採樣間隔和樣本大小。第三,您可以透過指定參數non_responder和comparison來指定是否要考慮非回應者以及是否要將結果與真實資料進行比較。此外,由於採樣方法是隨機的,因此您可以指定運行的迭代次數以獲得平均效能。而且,可以開啟parallel_computation來加速。最後,您可以開啟hyperparameter_autotune自動尋找超參數的最佳組合。
對於程式碼範例,您可以看到以下內容:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed