該倉庫包含:
sepal需要python3 ,最好是 3.5 以上的版本。若要下載並安裝,請開啟終端並將其變更為您想要將sepal下載到的目錄,然後執行以下操作:
git clone https://github.com/almaan/sepal.git
cd sepal
chmod +x setup.py
./setup.py install
根據您的使用者權限,您可能需要新增--user作為setup.py的參數。運行該設定將為您提供計算擴散時間所需的最少安裝。但是,如果您希望能夠使用分析模組,您還需要安裝建議的軟體包。為此,只需(在同一目錄中)運行:
pip install -e " .[full] "同樣,可能需要包含--user 。另外,如果這是您設定python-pip介面的方式,您可能必須使用pip3 。如果您使用conda或虛擬環境,請按照他們的建議安裝軟體包。
這應該安裝命令列介面 (CLI) 和標準套件。若要測試並查看安裝是否成功,您可以嘗試執行以下命令:
sepal -h
它應該會列印與萼片相關的幫助訊息。如果到目前為止一切順利,您可以繼續查看範例部分以查看sepal實際應用!
建議透過命令列介面使用 sepal。計算擴散時間的模擬以及隨後的結果分析或檢查都可以透過鍵入sepal並隨後run或analyze來輕鬆執行。 analyze模組有不同的選項,用於視覺化結果( inspect )、將檔案分類到模式家族( family )或對已識別的家族進行功能富集分析( fea )。有關可用命令的完整列表,請執行sepal module -h ,其中 module 是run和analyze之一。下面,我們說明如何使用萼片來找出具有空間模式的轉錄譜。
我們將建立一個資料夾來保存我們的結果,該資料夾也將作為我們的工作目錄。從儲存庫的主目錄中,執行以下操作:
cd res
mkdir example
cd exampleMOB 樣本將用於舉例說明我們的分析。我們首先計算每個轉錄譜的擴散時間:
sepal run -c ../../data/real/mob.tsv.gz -mo 10 -mc 5 -o . -ar 1下面是一個範例(附加顯示了幫助命令),展示了它的外觀

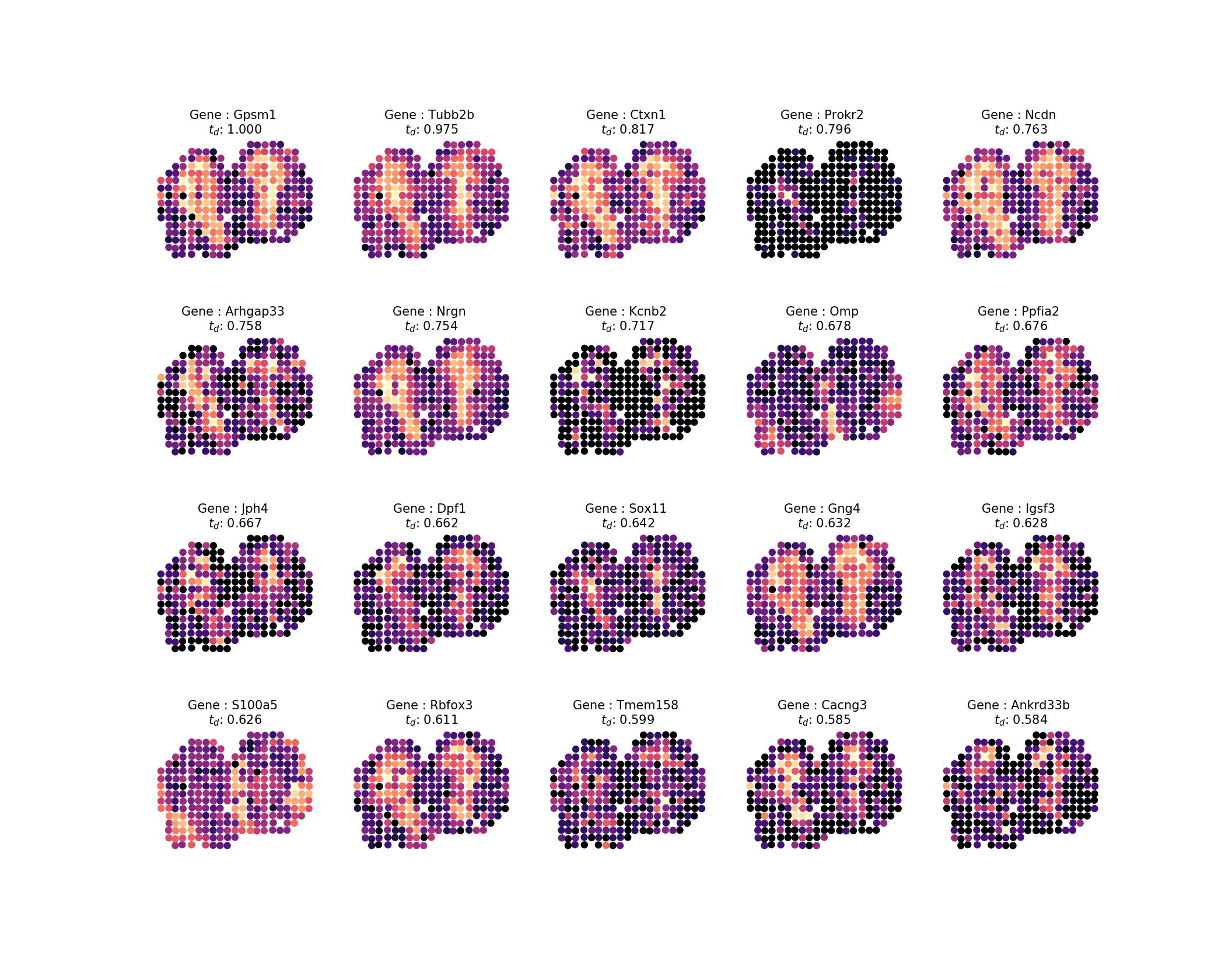
計算出擴散時間後,我們想要檢查結果,就像在研究中一樣,我們將查看前 20 個設定檔。我們可以透過執行以下命令輕鬆地根據結果生成圖像:
sepal analyze -c ../../data/real/mob.tsv.gz
-r 20200409173043610345-top-diffusion-times.tsv
-ar 1k -o . inspect -ng 20 -nc 5這看起來像這樣:

輸出將如下圖所示:
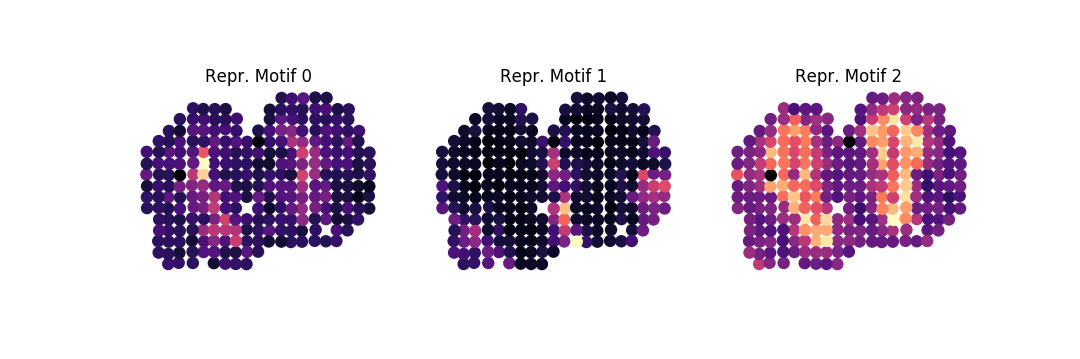
然後,要將排名前 100 的基因分類到一組模式族中,其中模式中 85% 的變異數應由特徵模式來解釋,請執行以下操作:
sepal analyze -c ../../data/real/mob.tsv.gz
-r 20200409173043610345-top-diffusion-times.tsv
-ar 1k -o . family -ng 100 -nbg 100 -eps 0.85 --plot -nc 3由此,我們得到每個家族以下三個代表性的主題:
我們可以透過執行以下命令對我們的家庭進行富集分析:
sepal analyze -c ../../data/real/mob.tsv.gz
-r 20200409173043610345-top-diffusion-times.tsv
-ar 1k -o . fea -fl mob.tsv-family-index.tsv -or " mmusculus "例如,我們看到 Family 2 豐富了與神經元功能、生成和調節相關的幾個過程:
| 家庭 | 本國的 | 姓名 | p_值 | 來源 | 交叉點大小 | |
|---|---|---|---|---|---|---|
| 2 | 2 | GO:0007399 | 神經系統發育 | 0.00035977 | 去:BP | 26 |
| 3 | 2 | 號碼:0050773 | 樹突發育的調控 | 0.000835883 | 去:BP | 8 |
| 4 | 2 | GO:0048167 | 突觸可塑性的調節 | 0.00196494 | 去:BP | 8 |
| 5 | 2 | 號碼:0016358 | 枝晶發育 | 0.00217167 | 去:BP | 9 |
| 6 | 2 | 號碼:0048813 | 樹突形態發生 | 0.00741589 | 去:BP | 7 |
| 7 | 2 | 號碼:0048814 | 樹突形態發生的調控 | 0.00800399 | 去:BP | 6 |
| 8 | 2 | 號碼:0048666 | 神經元發育 | 0.0114088 | 去:BP | 16 |
| 9 | 2 | 號碼:0099004 | 鈣調蛋白依賴性激酶訊號通路 | 0.0159572 | 去:BP | 3 |
| 10 | 2 | GO:0050804 | 化學突觸傳遞的調節 | 0.0341913 | 去:BP | 10 |
| 11 | 2 | GO:0099177 | 跨突觸訊號傳導的調節 | 0.0347783 | 去:BP | 10 |
當然,這種分析絕對不是詳盡無遺的。而是一個簡單的範例來展示如何操作sepal的 CLI。
雖然sepal被設計為獨立工具,但我們也將其建構成標準 python 包,可以從中匯入函數並在整合工作流程中使用。為了說明如何做到這一點,我們提供了一個範例,重現黑色素瘤分析。稍後可能會添加更多範例。
sepal的輸入必須採用n_locations x n_genes格式,但是如果您的資料以相反的方式建構( n_genes x n_locations ),則只需在執行模擬或分析時提供--transpose標誌即可的。
我們目前支援.csv 、 .tsv和.h5ad格式。對於後者,您的文件應根據此格式建置。我們預計scanpy團隊將在不久的將來發布一個版本,其中提出了空間資料的標準化格式,但在那之前我們將使用上述標準。
我們使用的所有真實數據都是公開的,可以透過以下連結找到:
合成數據是透過以下方式產生的:
synthetic/img2cnt.pysynthetic/turing.pysynthetic/ablation.py 研究中提出的所有結果都可以在res資料夾中找到,包括真實資料和合成資料。對於每個樣本,我們都相應地建立了結果:
res/sample-name/X-diffusion-times.tsv :所有排序基因的擴散時間analysis/ :包含二次分析的輸出