
(c) 2017 年蒂莫西·貝克爾和李萬平
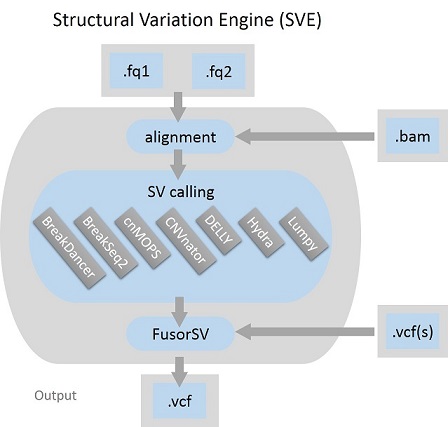
SVE 是一個基於 Python 腳本的執行引擎,用於結構變異 (SV) 檢測,可用於任何層級的資料輸入、原始 FASTQ、對齊的 BAM 或變異調用格式 (VCF),並產生統一的 VCF 作為其輸出。根據設計,SVE 預設由對齊、重新對齊和最先進的 SV 呼叫演算法集合組成。它們是 BreakDancer、BreakSeq、cnMOPS、CNVnator、DELLY、Hydra 和 LUMPY。 FusorSV 還嵌入了一種資料探勘方法,用於評估效能並合併來自 SV 呼叫演算法集合的呼叫集。
請設定ROOT環境。
export ROOTSYS=/ROOT_Build_Path
export LD_LIBRARY_PATH=$LD_LIBRARY_PATH:$ROOTSYS/lib
git clone --recursive https://github.com/TheJacksonLaboratory/SVE.git
cd SVE
make請檢查python2.7頭檔並修改Makefile中的「CFLAGS_FUSOR_SV」。頭檔可能位於“/usr/include/python2.7”上,並使用“CFLAGS_FUSOR_SV=-I /usr/include/python2.7”代替。
make FusorSV
或者,您可以透過 setup.py 安裝 FusorSV
cd SVE/scripts/FusorSV/
python setup.py build_ext --inplace
tar -zxvf data.tar.gz
或者,提供 Dockerfile 和 Docker 映像。請注意,根據您的電腦設置,docker 使用可能需要 sudo。
cd SVE
docker build .
從儲存庫中提取 docker 映像。
docker pull wanpinglee/sve
SVE 建構於 /tools/SVE 之上。透過查看幫助
/tools/SVE/bin/sve
FASTQ 中的短讀將根據給定的 FASTA 進行映射,並產生排序的 BAM。
bin/sve align [options] -r <FASTA> <FASTQ1 [FASTQ2]>
如果讀取以 BAM 格式給出,則重新對齊將根據 FASTA 重新映射讀取並產生排序的 BAM。我們使用 SpeedSeq 來完成重新對齊。
bin/sve realign -r <FASTA> <BAM>
有七種 SV 調用演算法可用於 SV 呼叫。將產生 VCF。
bin/sve call -r <FASTA> -g <hg19|hg38|others> -a <breakdancer|breakseq|cnvnator|hydra|delly|lumpy|cnmops> <BAM [BAM ...]>
呼叫後,每個樣本可能有多個 VCF,具體取決於使用的呼叫者數量。請將樣本的 VCF 收集到資料夾中。
vcfs 應使用 SVE ID 來指示呼叫者。
| SVE ID | 呼叫者 |
|---|---|
| 4 | 霹靂舞 (v1.4.5) |
| 9 | cn.MOPS (v1.20) |
| 10 | CNVnator (v0.3.3) |
| 11 | 黛莉 (v2) |
| 14* | 基因組STRiP |
| 17 號 | 九頭蛇 |
| 18 | 塊狀 |
| 35 | BreakSeq (v2.2) |
| 0 | 真相(可選) |
注意*:由於許可證問題,GenomeSTRiP 未嵌入 SVE 中。然而,FusorSV 預設模型能夠處理 GenomeSTRiP VCF。
範例輸入 vcf 檔案可以如下組織。請注意,vcfFiles 是 FusorSV 的 -i 的參數。
python scripts/FusorSV/FusorSV.py -f scripts/FusorSV/data/models/default.pickle -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
根據S0.vcf,將產生一個新模型,並且VCF將被新模型合併。
python scripts/FusorSV/FusorSV.py -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
該專案根據 GPL-3.0 許可證獲得許可。詳細資訊請參閱許可證。