歡迎來到 InstaDeep Github 儲存庫,其中有:
我們很高興能夠開源這些作品,並向社群提供這九種基因組語言模型和 2 個分割模型的程式碼和預訓練權重。 nucleotide transformer計畫的模型是與 Nvidia 和 TUM 合作開發的,並且這些模型在 Cambridge-1 上的 DGX A100 節點上進行訓練。 Agro nucleotide transformer專案的模型是與 Google 合作開發的,並在 TPU-v4 加速器上進行了訓練。
總的來說,我們的工作提供了與語言基礎模型的預訓練和應用相關的新穎見解,以及使用它們作為骨幹編碼器的模型的訓練,以及基因組學在該領域的應用的充足機會。
在此存儲庫中,您將找到以下內容:
與其他方法相比,我們的模型不僅整合了來自單一參考基因組的訊息,還利用了來自 3,200 多個不同人類基因組的 DNA 序列,以及來自廣泛物種(包括模型和非模型生物)的 850 個基因組。透過穩健和廣泛的評估,我們表明,與現有方法相比,這些大型模型提供了極其準確的分子表型預測。
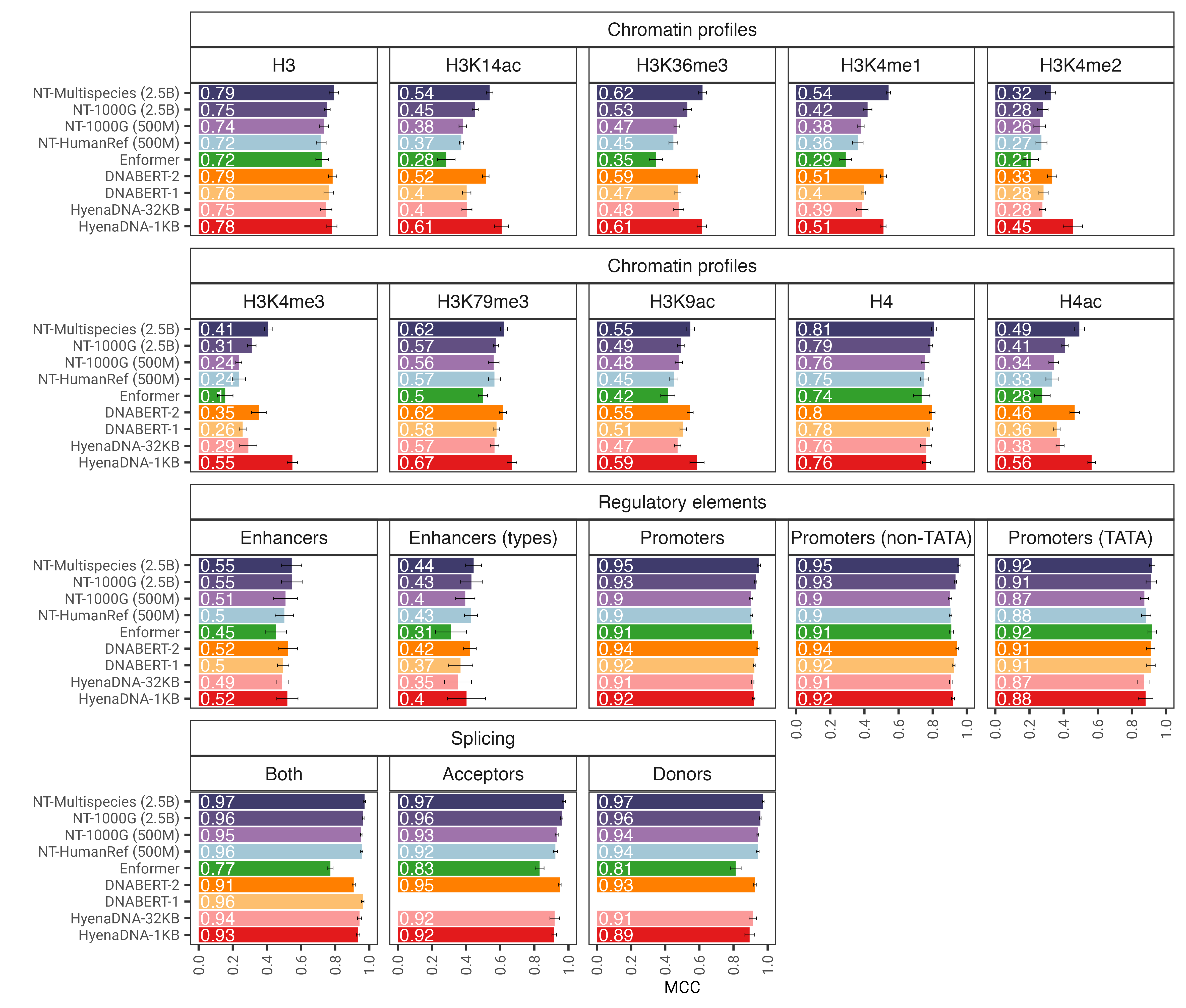
圖 1:經過微調後, nucleotide transformer模型可以準確預測各種基因組學任務。我們展示了微調變壓器模型的下游任務的性能結果。誤差線代表 2 個源自 10 倍交叉驗證的 SD。
在這項工作中,我們提出了一種新穎的基礎大語言模型,該模型在 48 個植物物種的參考基因組上進行訓練,主要關注作物物種。我們評估了 AgroNT 在調控特徵、RNA 處理和基因表現等多個預測任務中的表現,並表明 AgroNT 可以獲得最先進的性能。
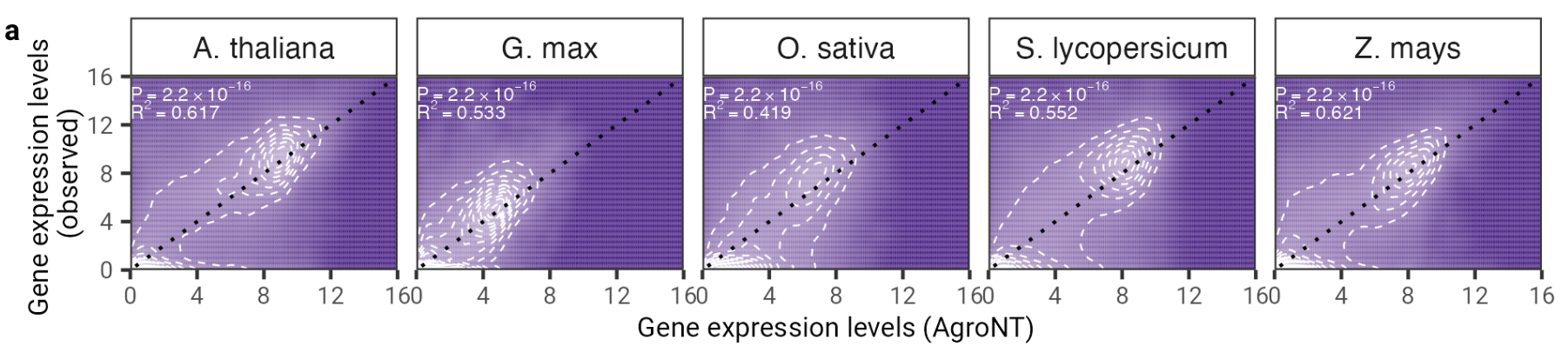
圖 2:AgroNT 提供不同植物物種的基因表現預測。所有組織中保留基因的基因表現預測與觀察到的基因表現量相關。顯示了線性模型的確定係數 (R 2 ) 以及預測值和觀測值之間的相關 P 值。
要使用程式碼和預訓練模型,只需:
pip install . 。然後,您只需幾行程式碼即可下載並使用我們的九個模型中的任何一個進行推理:
import haiku as hk
import jax
import jax . numpy as jnp
from nucleotide_transformer . pretrained import get_pretrained_model
# Get pretrained model
parameters , forward_fn , tokenizer , config = get_pretrained_model (
model_name = "500M_human_ref" ,
embeddings_layers_to_save = ( 20 ,),
max_positions = 32 ,
)
forward_fn = hk . transform ( forward_fn )
# Get data and tokenize it
sequences = [ "ATTCCGATTCCGATTCCG" , "ATTTCTCTCTCTCTCTGAGATCGATCGATCGAT" ]
tokens_ids = [ b [ 1 ] for b in tokenizer . batch_tokenize ( sequences )]
tokens_str = [ b [ 0 ] for b in tokenizer . batch_tokenize ( sequences )]
tokens = jnp . asarray ( tokens_ids , dtype = jnp . int32 )
# Initialize random key
random_key = jax . random . PRNGKey ( 0 )
# Infer
outs = forward_fn . apply ( parameters , random_key , tokens )
# Get embeddings at layer 20
print ( outs [ "embeddings_20" ]. shape )支援的型號名稱有:
您還可以運行我們的模型並在 google colab 中找到更多範例程式碼
感謝 Jax,程式碼可以在 GPU 和 TPU 上運行!
我們的第二個版本的nucleotide transformer v2 模型包括一系列被證明更有效的架構變化:我們不使用學習的位置嵌入,而是使用在每個注意層使用的旋轉嵌入和具有無偏差的快速啟動的門控線性單元。這些改進的模型還接受多達 2,048 個標記的序列,從而產生 12kbp 的更長上下文視窗。受Chinchilla 縮放定律的啟發,與v1 模型(300B 代幣)相比,我們還在多物種資料集上訓練NT-v2 模型,訓練持續時間更長(50M 和100M 模型使用300B 代幣;250M 和500M模型使用1T 令牌)適用於所有四種型號)。
轉換器層的索引為 1,這表示使用參數model_name="500M_human_ref"和embeddings_layers_to_save=(1, 20,)呼叫get_pretrained_model將導致在第一個和第 20 個轉換器層之後提取嵌入。對於使用 Roberta LM head 的 Transformer,通常的做法是在 LM head 的第一層範數之後而不是在最後一個 Transformer 區塊之後提取最終嵌入。因此,如果使用下列參數呼叫get_pretrained_model embeddings_layers_to_save=(24,) ,則嵌入不會在最終 Transformer 層之後提取,而是在 LM 頭的第一層範數之後提取。
SegmentNT 模型利用nucleotide transformer (NT) 變換器,我們從中刪除了語言模型頭並替換為一維U-Net 分段頭,以單核苷酸分辨率預測序列中幾種類型的基因組元素的位置。我們在高達 30kb 的輸入序列中針對 14 種不同類別的人類基因組元素提出了兩種不同的模型變體。這些包括基因(蛋白質編碼基因、lncRNA、5'UTR、3'UTR、外顯子、內含子、剪接受體和供體位點)和調節(polyA 訊號、組織不變和組織特異性啟動子和增強子,以及CTCF 結合站點)元素。受益於 NT 的預訓練權重,SegmentNT 實現了優於最先進的 U-Net 分割架構的性能,並展示了高達 50kbp 的零樣本泛化能力。
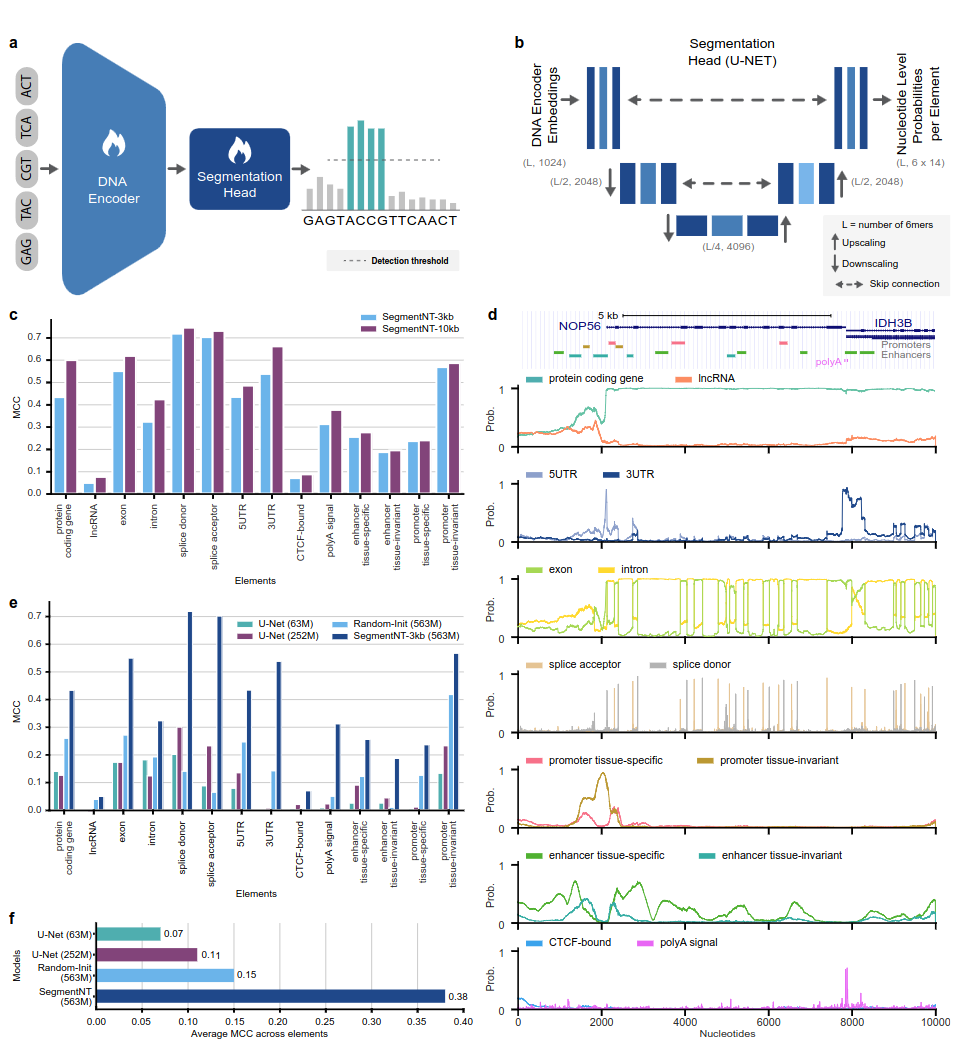
圖 1:SegmentNT 以核苷酸解析度定位基因組元件。
要使用程式碼和預訓練模型,只需:
pip install . 。然後,您只需幾行程式碼即可下載並使用我們的任何模型推斷序列:
rescaling factor設定為訓練期間使用的因子。如果您需要推斷 30kbp 到 50kbp 之間的序列,請確保在get_pretrained_segment_nt_model函數中傳遞rescaling_factor參數,其值為rescaling_factor = max_num_nucleotides / max_num_tokens_nt其中num_dna_tokens_inference基的序列)對)和max_num_tokens_nt是骨幹核苷酸轉換器訓練的最大標記數,即2048 。
?筆記本examples/inference_segment_nt.ipynb展示如何推斷 50kb 序列並繪製機率以重現論文的圖 3。
? SegmentNT 模型不處理輸入序列中的任何“N”,因為每個核苷酸都需要標記為 6 聚體,而使用包含一個或多個“N”鹼基對的序列時則不能出現這種情況。
import haiku as hk
import jax
import jax . numpy as jnp
from nucleotide_transformer . pretrained import get_pretrained_segment_nt_model
# Initialize CPU as default JAX device. This makes the code robust to memory leakage on
# the devices.
jax . config . update ( "jax_platform_name" , "cpu" )
backend = "cpu"
devices = jax . devices ( backend )
num_devices = len ( devices )
print ( f"Devices found: { devices } " )
# The number of DNA tokens (excluding the CLS token prepended) needs to be dividible by
# 2 to the power of the number of downsampling block, i.e 4.
max_num_nucleotides = 8
assert max_num_nucleotides % 4 == 0 , (
"The number of DNA tokens (excluding the CLS token prepended) needs to be dividible by"
"2 to the power of the number of downsampling block, i.e 4." )
parameters , forward_fn , tokenizer , config = get_pretrained_segment_nt_model (
model_name = "segment_nt" ,
embeddings_layers_to_save = ( 29 ,),
attention_maps_to_save = (( 1 , 4 ), ( 7 , 10 )),
max_positions = max_num_nucleotides + 1 ,
)
forward_fn = hk . transform ( forward_fn )
apply_fn = jax . pmap ( forward_fn . apply , devices = devices , donate_argnums = ( 0 ,))
# Get data and tokenize it
sequences = [ "ATTCCGATTCCGATTCCAACGGATTATTCCGATTAACCGATTCCAATT" , "ATTTCTCTCTCTCTCTGAGATCGATGATTTCTCTCTCATCGAACTATG" ]
tokens_ids = [ b [ 1 ] for b in tokenizer . batch_tokenize ( sequences )]
tokens_str = [ b [ 0 ] for b in tokenizer . batch_tokenize ( sequences )]
tokens = jnp . asarray ( tokens_ids , dtype = jnp . int32 )
random_key = jax . random . PRNGKey ( seed = 0 )
keys = jax . device_put_replicated ( random_key , devices = devices )
parameters = jax . device_put_replicated ( parameters , devices = devices )
tokens = jax . device_put_replicated ( tokens , devices = devices )
# Infer on the sequence
outs = apply_fn ( parameters , keys , tokens )
# Obtain the logits over the genomic features
logits = outs [ "logits" ]
# Transform them in probabilities
probabilities = jnp . asarray ( jax . nn . softmax ( logits , axis = - 1 ))[..., - 1 ]
print ( f"Probabilities shape: { probabilities . shape } " )
print ( f"Features inferred: { config . features } " )
# Get probabilities associated with intron
idx_intron = config . features . index ( "intron" )
probabilities_intron = probabilities [..., idx_intron ]
print ( f"Intron probabilities shape: { probabilities_intron . shape } " )支援的型號名稱有:
感謝 Jax,程式碼可以在 GPU 和 TPU 上運行!
這些模型在長度最多 1000 個標記的序列上進行訓練,包括自動添加到序列開頭的 <CLS> 標記。分詞器透過將字母「A」、「C」、「G」和「T」分組為 6 聚體,開始從左到右分詞。 「N」字母被選擇不分組在k-mers 內,因此每當分詞器遇到「N」時,或者如果序列中的核苷酸數量不是6 的倍數,它將對核苷酸進行分詞而不分組他們。舉例如下:
dna_sequence_1 = "ACGTGTACGTGCACGGACGACTAGTCAGCA"
tokenized_dna_sequence_1 = [ < CLS > , < ACGTGT > , < ACGTGC > , < ACGGAC > , < GACTAG > , < TCAGCA > ]
dna_sequence_2 = "ACGTGTACNTGCACGGANCGACTAGTCTGA"
tokenized_dna_sequence_2 = [ < CLS > , < ACGTGT > , < A > , < C > , < N > , < TGCACG > , < G > , < A > , < N > , < CGACTA > , < GTCTGA > ]因此,如果內部沒有“N”,所有 v1 和 v2 轉化子可以分別採用最多 5994 個和 12282 個核苷酸的序列。
此儲存庫中提供的模型集合可在 Instadeep 的擁抱空間中找到: nucleotide transformer空間和農業nucleotide transformer空間!
我們感謝 Maša Roller 以及 Rostlab 的成員,特別是 Tobias Olenyi、Ivan Koludarov 和 Burkhard Rost 的建設性討論,幫助確定了有趣的研究方向。此外,我們向所有將實驗數據存入公共資料庫的人、維護這些資料庫的人以及免費提供分析和預測方法的人表示感謝。我們也感謝 Jax 開發團隊。
如果您發現此儲存庫對您的工作有用,請在我們的相關論文中添加相關引用:
nucleotide transformer論文:
@article { dalla2023nucleotide ,
title = { The nucleotide transformer : Building and Evaluating Robust Foundation Models for Human Genomics } ,
author = { Dalla-Torre, Hugo and Gonzalez, Liam and Mendoza Revilla, Javier and Lopez Carranza, Nicolas and Henryk Grywaczewski, Adam and Oteri, Francesco and Dallago, Christian and Trop, Evan and Sirelkhatim, Hassan and Richard, Guillaume and others } ,
journal = { bioRxiv } ,
pages = { 2023--01 } ,
year = { 2023 } ,
publisher = { Cold Spring Harbor Laboratory }
}農業nucleotide transformer紙:
@article { mendoza2024foundational ,
title = { A foundational large language model for edible plant genomes } ,
author = { Mendoza-Revilla, Javier and Trop, Evan and Gonzalez, Liam and Roller, Ma{v{s}}a and Dalla-Torre, Hugo and de Almeida, Bernardo P and Richard, Guillaume and Caton, Jonathan and Lopez Carranza, Nicolas and Skwark, Marcin and others } ,
journal = { Communications Biology } ,
volume = { 7 } ,
number = { 1 } ,
pages = { 835 } ,
year = { 2024 } ,
publisher = { Nature Publishing Group UK London }
}段NT紙
@article { de2024segmentnt ,
title = { SegmentNT: annotating the genome at single-nucleotide resolution with DNA foundation models } ,
author = { de Almeida, Bernardo P and Dalla-Torre, Hugo and Richard, Guillaume and Blum, Christopher and Hexemer, Lorenz and Gelard, Maxence and Pandey, Priyanka and Laurent, Stefan and Laterre, Alexandre and Lang, Maren and others } ,
journal = { bioRxiv } ,
pages = { 2024--03 } ,
year = { 2024 } ,
publisher = { Cold Spring Harbor Laboratory }
}如果您對程式碼和模型有任何疑問或回饋,請隨時與我們聯繫。
感謝您對我們工作的興趣!