guar_genotypes
1.0.0
包含瓜爾豆 ( Cyamopsis tetragonoloba (L.) Taub.) 基因分型和 GWAS 計畫的資料和程式碼的儲存庫。儲存庫內容的簡要描述:
make_consensus_vcf.py腳本用於使用原始 GATK-HC、NGSEP 和 TASSEL 5 變體呼叫產生最終變體資料集, make_snp_stats.py腳本用於相同的輸入以探索變體呼叫的品質統計資料。
callset_refinement_filtering.ipynb筆記本用於運行變體品質控制並建立常見變體的最終過濾資料集。
genetic_analysis.ipynb筆記本用於執行大部分遺傳分析,包括 PCA、連鎖不平衡分析和基於廣義線性模型的關聯分析。
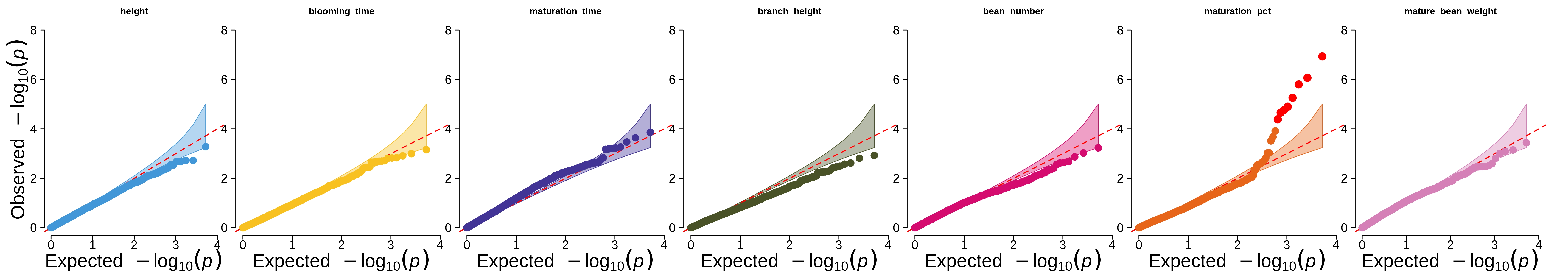
使用prepare_farmcpu.py腳本來格式化FarmCPU 工具的輸入資料(所得p 值的QQ 圖顯示在論文和下方)。
admix_file_create.py檔案用於預處理 ADMIXTURE 分析的基因型(其中部分是使用genetic_analysis.ipynb中的 Hail 完成的)。
guar_stat.R腳本用於對最終數據進行統計分析,使用FarmCPU工具進行全基因組關聯分析,並繪製主要數據
parse_vcf_fcpu.py腳本用於格式化最終的 SNP 基因型表,該表可作為此儲存庫中的genotypes.xlsx檔案使用。