MedSegDiff 是一種基於擴散機率模型 (DPM) 的醫學影像分割框架。演算法在我們的論文 MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model 和 MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer 中進行了詳細闡述。
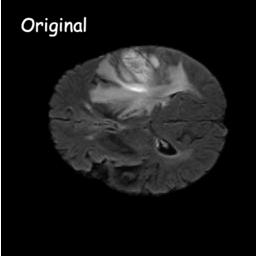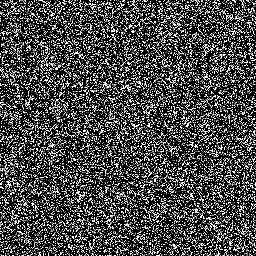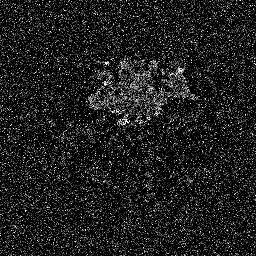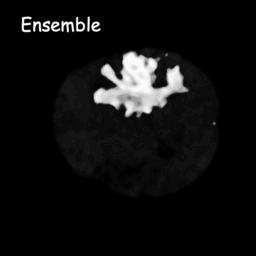 擴散模型的工作原理是透過連續添加高斯雜訊來破壞訓練數據,然後學習透過逆轉該雜訊過程來恢復資料。訓練後,我們可以使用擴散模型透過學習的去噪過程簡單地傳遞隨機取樣的雜訊來產生資料。我們利用原始影像作為條件,從隨機雜訊中產生多個分割圖,然後對它們進行整合以獲得最終結果。這種方法捕捉了醫學影像中的不確定性,並且在多個基準測試中優於先前的方法。
擴散模型的工作原理是透過連續添加高斯雜訊來破壞訓練數據,然後學習透過逆轉該雜訊過程來恢復資料。訓練後,我們可以使用擴散模型透過學習的去噪過程簡單地傳遞隨機取樣的雜訊來產生資料。我們利用原始影像作為條件,從隨機雜訊中產生多個分割圖,然後對它們進行整合以獲得最終結果。這種方法捕捉了醫學影像中的不確定性,並且在多個基準測試中優於先前的方法。
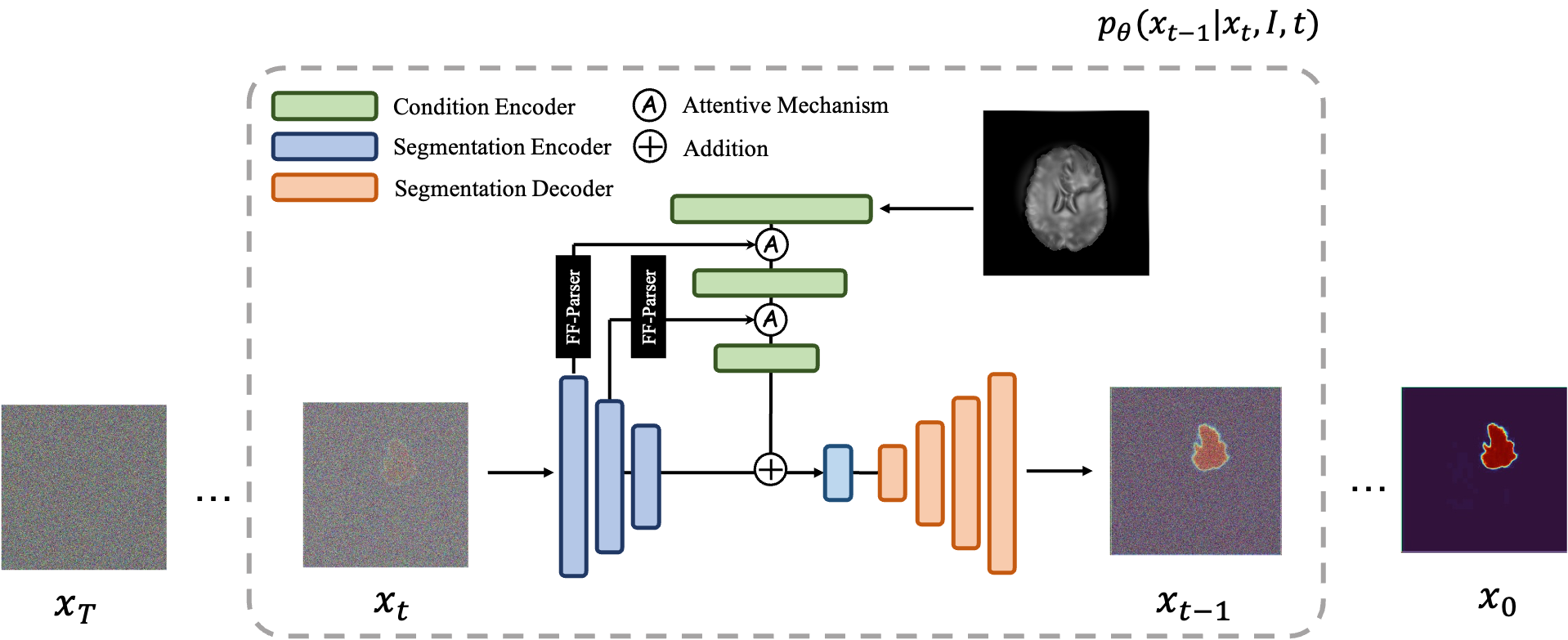 | 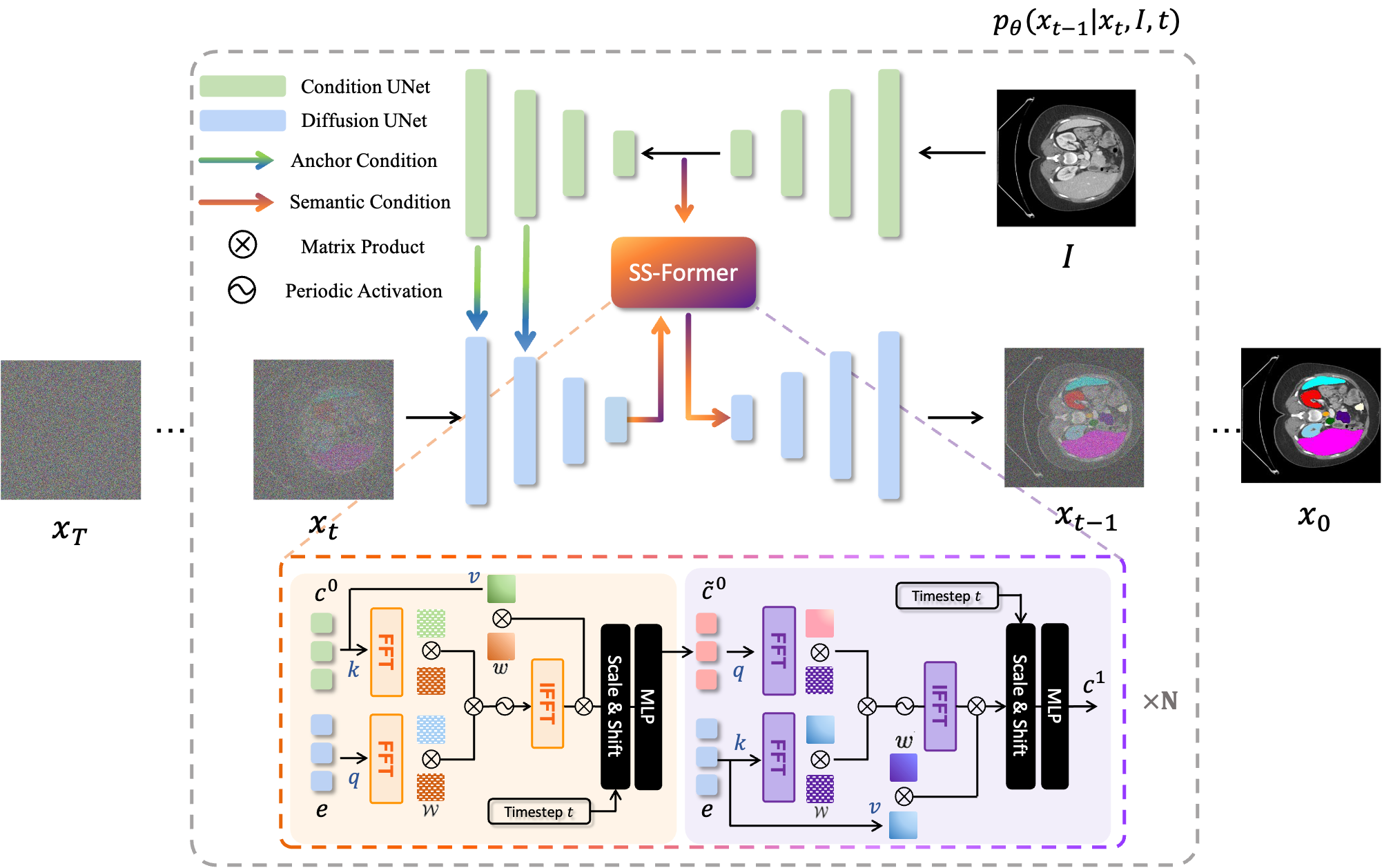 |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver True享受其閃電般的採樣速度(1000 步 20 步⭕️)。python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
對於訓練,運行: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
對於採樣,請執行: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
為了進行評估,請執行python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
預設情況下,樣本將保存在./results/
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
對於訓練,運行: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
對於採樣,運行: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
……
在其他資料集上執行 MedSegDiff 很簡單。只需在./guided_diffusion/isicloader.py或./guided_diffusion/bratsloader.py之後編寫另一個資料載入器檔案。如果您遇到任何問題,歡迎開啟問題。如果您能貢獻您的資料集擴展,我們將不勝感激。與自然影像不同,醫學影像根據不同的任務而有很大差異。擴大一種方法的泛化性需要每個人的努力。
為了訓練一個精細的模型,即論文中的MedSegDiff-B,將模型超參數設定為:
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
擴散超參數為:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
為了加快採樣速度:
--diffusion_steps 50 --dpm_solver True
在多個 GPU 上運行:
--multi-gpu 0,1,2 (for example)
訓練超參數為:
--lr 5e-5 --batch_size 8
並在採樣中設定--num_ensemble 5 。
訓練中運行大約 100,000 個步驟將在大多數資料集上收斂。請注意,雖然後面的大多數步驟中損失不會減少,但結果的品質仍在提高。在其他 DPM 應用程式(例如影像生成)上也可以觀察到這樣的過程。希望懂的人能告訴我這是為什麼嗎?
我很快就會發布它在較小批量大小(適合在 24GB GPU 上運行)下的性能,以供比較。
釋放其所有潛力的設定是 (MedSegDiff++):
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
然後使用批次大小--batch_size 64進行訓練,並使用集合編號--num_ensemble 25對其進行採樣。
歡迎為 MedSegDiff 做出貢獻。任何可以提高效能或加速演算法的技術都是值得讚賞的。我正在寫 MedSegDiff V2,目標是 Nature 期刊/CVPR 之類的出版物。我很高興將貢獻者列為我的共同作者?
程式碼複製了許多來自 openai/improved-diffusion、WuJunde/ MrPrism、WuJunde/ DiagnosisFirst、LuChengTHU/dpm-solver、JuliaWolleb/Diffusion-based-Segmentation、hojonathanho/diffusion、guided-diffus-gem. -of-Unet、nnUnet、lucidrains/vit-pytorch
請引用
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu