
(ج) 2017 تيموثي بيكر ووان بينج لي
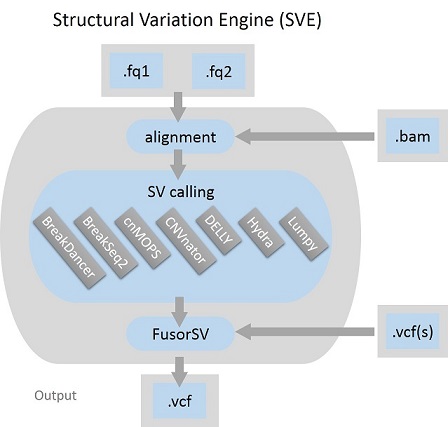
SVE هو محرك تنفيذ يستند إلى برنامج Python لاكتشاف التباين الهيكلي (SV) ويمكن استخدامه لأي مستويات من مدخلات البيانات، أو FASTQs الخام، أو BAMs المحاذاة، أو تنسيق الاتصال المتغير (VCFs)، ويقوم بإنشاء VCF موحد كمخرجاته. حسب التصميم، يتكون SVE من المحاذاة وإعادة التنظيم ومجموعة من خوارزميات استدعاء SV المتطورة بشكل افتراضي. وهم BreakDancer، وBreakSeq، وcnMOPS، وCNVnator، وDELLY، وHydra، وLUMPY. تم تضمين FusorSV أيضًا وهو أسلوب لاستخراج البيانات لتقييم الأداء ودمج مجموعات الاتصال من مجموعة من خوارزميات استدعاء SV.
يرجى ضبط بيئة الجذر.
export ROOTSYS=/ROOT_Build_Path
export LD_LIBRARY_PATH=$LD_LIBRARY_PATH:$ROOTSYS/lib
git clone --recursive https://github.com/TheJacksonLaboratory/SVE.git
cd SVE
makeالرجاء التحقق من ملفات رأس python2.7 وتعديل "CFLAGS_FUSOR_SV" في Makefile. قد تكون ملفات الرأس موجودة على "/usr/include/python2.7" واستخدم "CFLAGS_FUSOR_SV=-I /usr/include/python2.7" بدلاً من ذلك.
make FusorSV
أو يمكنك تثبيت FusorSV عن طريق setup.py
cd SVE/scripts/FusorSV/
python setup.py build_ext --inplace
tar -zxvf data.tar.gz
وبدلاً من ذلك، يتم توفير ملف Dockerfile وصورة Docker. يرجى ملاحظة أن Sudo قد يكون مطلوبًا لاستخدامات عامل الإرساء وفقًا لإعدادات جهازك.
cd SVE
docker build .
اسحب صورة عامل الإرساء من المستودع.
docker pull wanpinglee/sve
تم بناء SVE على /tools/SVE. التحقق من المساعدة من خلال
/tools/SVE/bin/sve
سيتم تعيين القراءات القصيرة في FASTQ مقابل FASTA المحدد وسيتم إنشاء BAM مرتبة.
bin/sve align [options] -r <FASTA> <FASTQ1 [FASTQ2]>
إذا تم تقديم القراءات بواسطة تنسيق BAM، فستقوم إعادة المحاذاة بإعادة تعيين القراءات مقابل FASTA وإنشاء BAM تم فرزها. نحن نستخدم SpeedSeq لإنجاز إعادة التنظيم.
bin/sve realign -r <FASTA> <BAM>
هناك سبع خوارزميات اتصال SV يمكن استخدامها للاتصال SV. سيتم إنشاء VCF.
bin/sve call -r <FASTA> -g <hg19|hg38|others> -a <breakdancer|breakseq|cnvnator|hydra|delly|lumpy|cnmops> <BAM [BAM ...]>
بعد الاتصال، قد تحتوي كل عينة على VCFs متعددة اعتمادًا على عدد المتصلين الذين تم استخدامهم. يرجى جمع VCFs لعينة في مجلد.
يجب أن يستخدم vcfs معرفات SVE للإشارة إلى المتصلين.
| معرف SVE | المتصل |
|---|---|
| 4 | بريك دانسر (v1.4.5) |
| 9 | cn.MOPS (الإصدار 1.20) |
| 10 | سي إن فيناتور (الإصدار 0.3.3) |
| 11 | ديلي (الإصدار الثاني) |
| 14* | جينوم ستريب |
| 17 | هيدرا |
| 18 | محفر |
| 35 | بريك سيك (الإصدار 2.2) |
| 0 | الحقيقة (اختياري) |
ملاحظة*: بسبب مشكلة الترخيص، لم يتم تضمين GenomeSTRiP في SVE. ومع ذلك، فإن النموذج الافتراضي FusorSV قادر على التعامل مع GenomeSTRiP VCF.
يمكن تنظيم ملفات vcf المدخلة على النحو التالي. يرجى ملاحظة أن vcfFiles هي الوسيطة لـ -i لـ FusorSV.
python scripts/FusorSV/FusorSV.py -f scripts/FusorSV/data/models/default.pickle -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
وفقًا لـ S0.vcf، سيتم إنشاء نموذج جديد وسيتم دمج VCFs بواسطة النموذج الجديد.
python scripts/FusorSV/FusorSV.py -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
المشروع مرخص بموجب ترخيص GPL-3.0. يرجى الاطلاع على الترخيص للحصول على التفاصيل.