spfy : منصة للتنبؤ بالأنواع الفرعية من تسلسل الجينوم الكامل للإشريكية القولونية، وإنشاء بيانات بيانية للتحليلات المقارنة على مستوى السكان.
نُشرت باسم: Le,KK, Whiteside,MD, Hopkins,JE, Gannon,VPJ, Laing,CR spfy : قاعدة بيانات رسم بياني متكاملة للتنبؤ في الوقت الفعلي بالأنماط الظاهرية البكتيرية والتحليلات المقارنة النهائية. قاعدة البيانات (2018) المجلد. 2018: معرف المقالة bay086؛ دوى:10.1093/قاعدة البيانات/bay086
مباشر: https://lfz.corefacility.ca/superphy/spfy/
git clone --recursive https://github.com/superphy/spfy.gitcd spfy /docker-compose upنوع EC:
عموم بريديك:
صورة عامل ميناء لكوندا:
مقارنة المجموعات السكانية المختلفة:
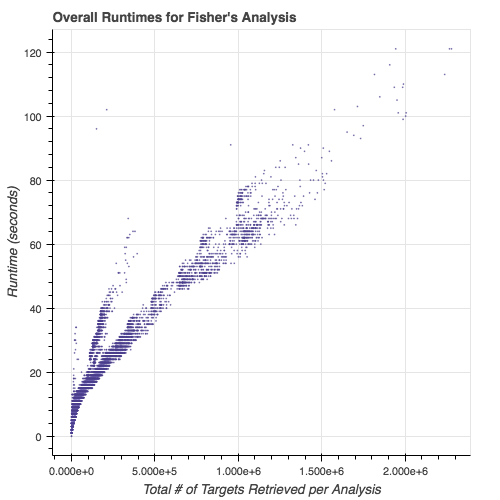
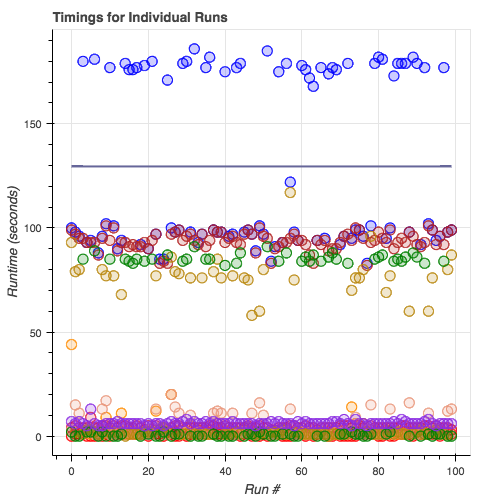
أوقات تشغيل وحدات الكتابة الفرعية:
cd app/python -m modules/savvy -i tests/ecoli/GCA_001894495.1_ASM189449v1_genomic.fna حيث تكون الوسيطة بعد -i هي ملف الجينوم (FASTA) الخاص بك. 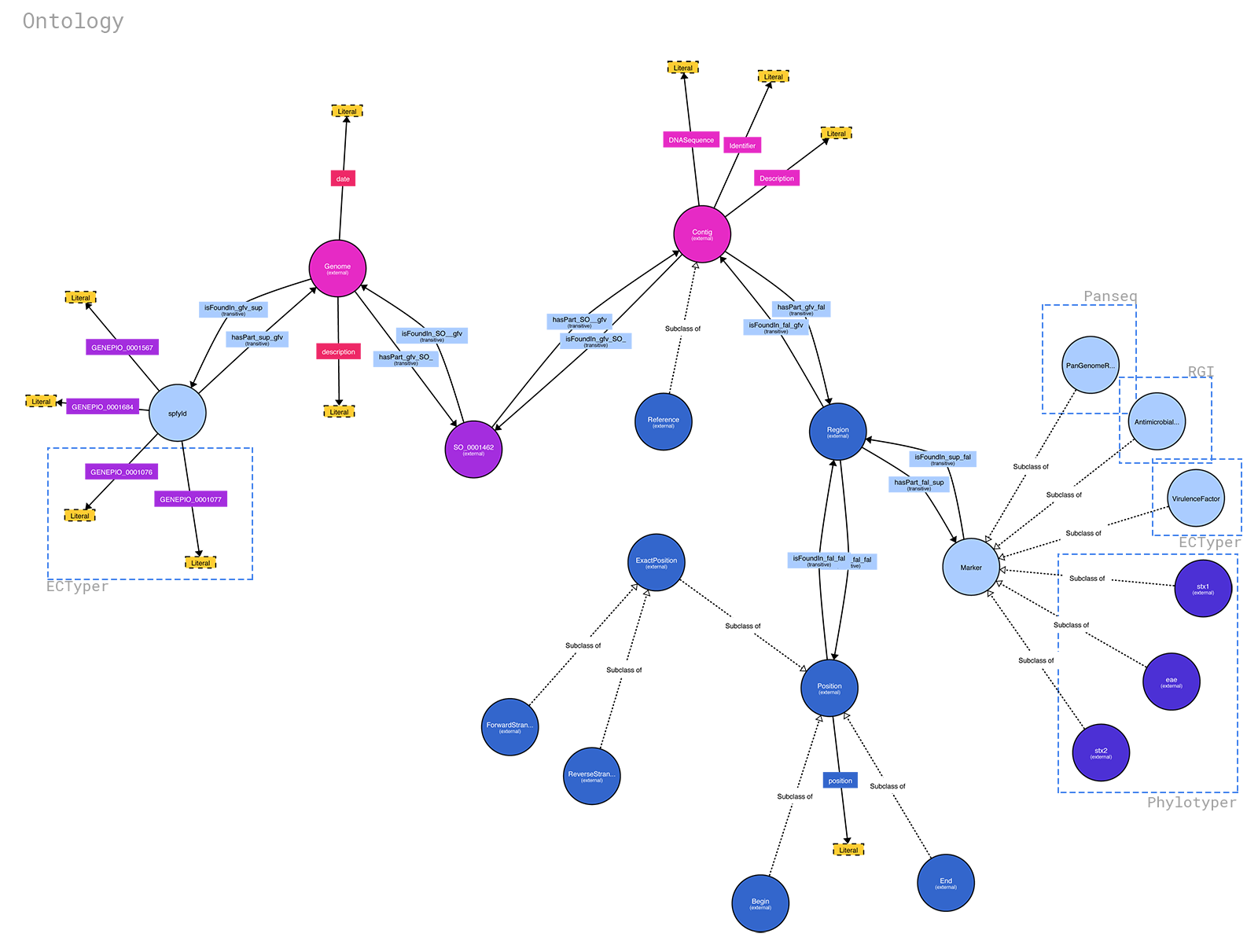
علم الوجود الخاص بـ spfy متاح على: https://raw.githubusercontent.com/superphy/backend/master/app/scripts/spfy_ontology.ttl تم إنشاؤه باستخدام https://raw.githubusercontent.com/superphy/backend/master /app/scripts/generate_ontology.py مع الوظائف المشتركة من كود الواجهة الخلفية لـ spfy . إذا كنت ترغب في تشغيله، فافعل: cd app/ python -m scripts/generate_ontology والذي سيضع علم الوجود في app/
يمكنك إنشاء رسم تخطيطي جميل من ملف .ttl باستخدام http://www.visualdataweb.de/webvowl/
ملحوظة
الإعداد حاليًا لملفات .fna فقط
يمكنك تجاوز موقع الويب الأمامي والاستمرار في إدراج مهام الكتابة الفرعية عن طريق:
/datastore في الحاويات.على سبيل المثال، إذا احتفظت بملفاتك في
/home/bob/ecoli-genomes/، فيمكنك تحرير ملفdocker-compose.ymlواستبداله:volumes : - /datastoreمع:
volumes : - /home/bob/ecoli-genomes:/datastore
docker-compose down docker-compose up -d
docker exec -it backend_webserver_1 sh python -m scripts/sideload exit
لاحظ أنه قد يتم إنشاء المخلفات في مجلد الجينوم الخاص بك.

| قفص الاتهام إيه الصورة ه | ميناء ق | الاسم س | وصف |
|---|---|---|---|
| النهاية الخلفية- rq | 80/طن سي بي، 443/تي سي بي | النهاية الخلفية_wor ker_1 | عمال قائمة الانتظار الرئيسية |
| النهاية الخلفية- rq-b laze grap h | 80/طن سي بي، 443/تي سي بي | النهاية الخلفية_وور كير-بلاز إجرا ph-i ds_ 1 | تم استخدام هذه النسبة الجينية spfy ID لقاعدة بيانات blaz egra ph |
| النهاية الخلفية | 0.0. 0.0: 8000 ->80 /تكب، 443/تكب | back end_web -ngi nx-u wsgi _1 | النهاية الخلفية للفلاش التي تقوم بالبحث عن المهام |
| سوب rphy /bla zegr aph: 2.1. 4- في فري نسين ز | 0.0. 0.0: 8080 ->80 80/t cp | النهاية الخلفية_bla zegr aph_1 | قاعدة بيانات Blaz egra ph |
| ريدي ق:3. 2 | 6379 / تكب | النهاية الخلفية_الأحمر_1 | قاعدة بيانات ريدي |
| رد فعل تاب | 0.0. 0.0: 8090 ->50 00/طن سي بي | النهاية الخلفية_rea ctap p_1 | fron t-en d إلى spfy |
الصورة superphy/backend-rq:2.0.0 قابلة للتطوير : يمكنك إنشاء العديد من المثيلات التي تحتاجها/تمتلك قوة المعالجة لها. الصورة مسؤولة عن الاستماع إلى قائمة الانتظار multiples (12 عاملاً) والتي تتولى معظم المهام، بما في ذلك مكالمات RGI . كما أنه يستمع إلى قائمة الانتظار singles (عامل واحد) التي تقوم بتشغيل ECTyper . يتم ذلك لأن RGI هو الجزء الأبطأ في المعادلة. إدارة العمال في التعامل معها في supervisor .
صورة superphy/backend-rq-blazegraph:2.0.0 غير قابلة للتطوير: فهي مسؤولة عن الاستعلام عن قاعدة بيانات Blazegraph للإدخالات المكررة وتعيين معرفات spfy بترتيب تسلسلي . يتم الاحتفاظ بوظائفها في أدنى حد ممكن لتحسين الأداء (نظرًا لأن إنشاء المعرف هو عنق الزجاجة الوحيد في خطوط الأنابيب المتوازية)؛ تتم المقارنات عن طريق تجزئة sha1 للملفات المقدمة ويتم حجز معرفاتها غير المكررة عن طريق ربط معرف spfy الذي تم إنشاؤه بتجزئة الملف. إدارة العمال في التعامل معها في supervisor .
يستخدم superphy/backend:2.0.0 الذي يدير نقاط نهاية Flask supervisor لإدارة العمليات الداخلية: nginx و uWsgi .
database['blazegraph_url'] في /app/config.py تم توثيق الخطوات المطلوبة لإضافة وحدات جديدة في دليل المطور.