
ISLING هي أداة للكشف عن تكامل الفيروسي أو المتجهات في القراءات المقترنة. يرجى قراءة ورقتنا للحصول على التفاصيل الكاملة.
إذا قمت بتثبيت conda و snakemake ، لتشغيلها مع بيانات الاختبار (المضمنة) محليًا:
git clone https://github.com/aehrc/isling.git && cd isling
snakemake --configfile test/config/test.yml --cores <cores> --use-conda
إذا كنت قد تم تثبيت snakemake و singularity ، فيمكنك الاستخدام بدلاً من ذلك:
snakemake --configfile test/config/test.yml --cores <cores> --use-singularity
بدلاً من ذلك ، إذا قمت بتثبيت Docker ، على MacOS ، يمكنك تشغيل:
docker run --rm -it -v"$(pwd)"/out:/opt/isling/out szsctt/isling:latest snakemake --configfile test/config/test.yml --cores 1
سيستخدم هذا ملف التكوين والبيانات داخل الحاوية ، وستظهر النتائج في مجلد تم out في دليل العمل الحالي الخاص بك. على Linux ، ستحتاج إلى تشغيل هذا الأمر كجذر ، وعلى Windows ستحتاج إلى ضبط بناء جملة Mount ( -v ).
يتم تحديد بيانات الإدخال (القراءات والمضيف والمراجع الفيروسية) ni ملف التكوين - بالنسبة لبياناتك الخاصة ، ستحتاج إلى تعديل ملف التكوين ( test/config/test.yml ) للإشارة إلى بياناتك الخاصة. راجع configfile.md لمزيد من المعلومات حول تنسيق ملف التكوين.
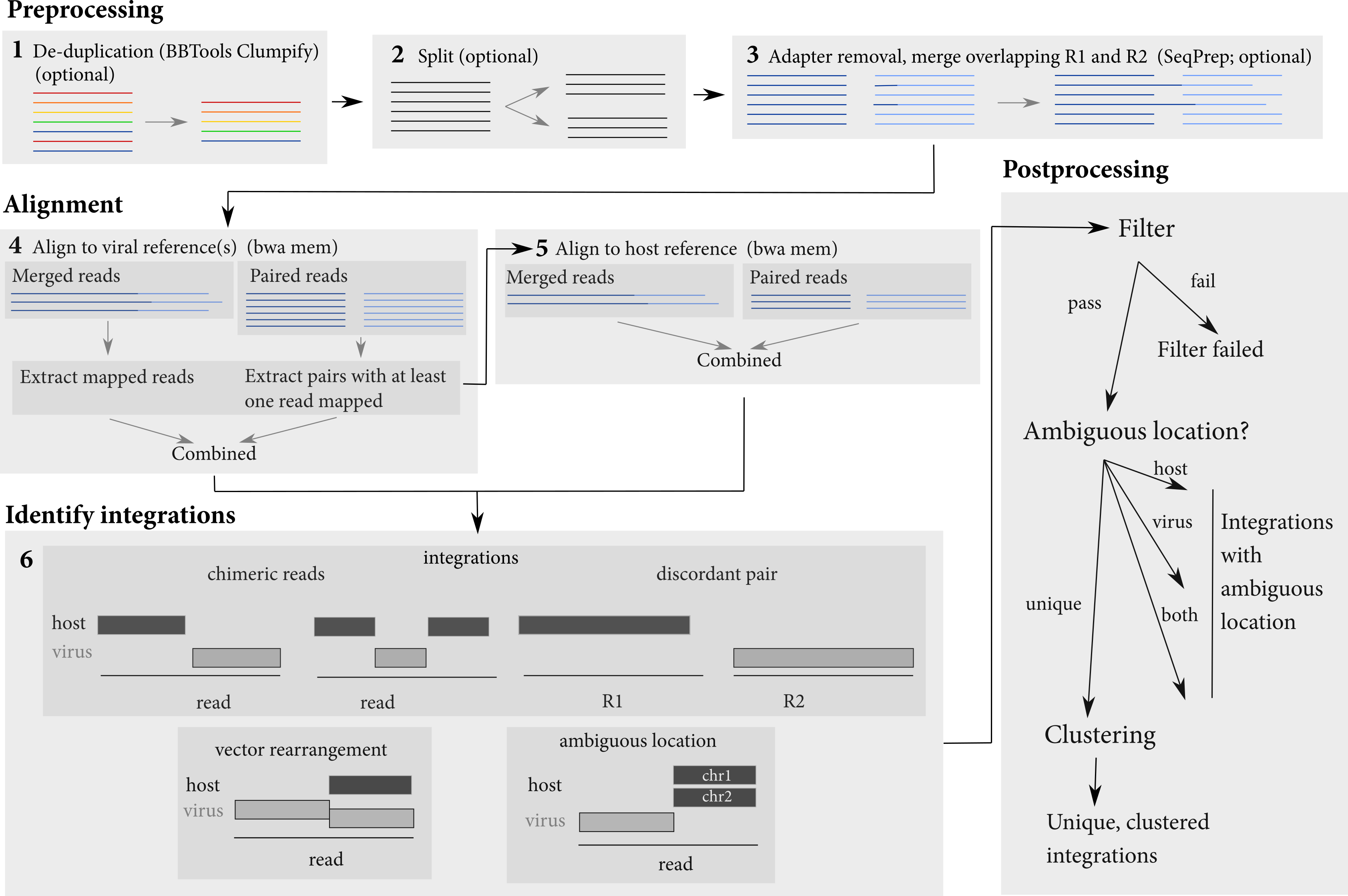
يقوم خط الأنابيب بإجراء عدة خطوات من أجل تحديد مواقع التكامل. يستغرق الأمر كمجموعات بيانات إدخال تتكون من ملفات FastQ أو ملفات BAM. يقوم ببعض المعالجة المسبقة للقراءات (دمج القراءات المتداخلة ، اختياري) ثم يحاذه مع كل من المضيف والتسلسل الفيروسي. يتم محاذاة القراءات أولاً مع التسلسل الفيروسي ، ثم يتم استخراج القراءات المحاذاة وتوافقها مع المضيف. يتم استخدام هذه المحاذاة لتحديد التكامل الفيروسي.
يتطلب ISLING snakemake وإما singularity (الموصى به) أو conda لتزويد التبعيات. الإضافة ، الإصدار python 3.5 أو أعلى pandas مطلوبة (يجب تثبيت هذه تلقائيًا في حالة تثبيت snakemake مع conda .
Alternativley ، استخدم إصدار Docker الذي يحتوي على Isling وجميع التبعيات.
المدخلات المطلوبة هي ملف التكوين ، الذي يحدد المراجع المضيف والفيروس/المتجهات ، والقراءات مطلوبة. حدد جميع المدخلات في ملف التكوين. يعمل Isling حاليًا فقط للقراءات المقترنة.
راجع ملف configfile.md للحصول على وصف لتنسيق ملف التكوين هذا.
مواقع تكامل مخرجات ISLING بتنسيق مفصل لعلامات التبويب في دليل الإخراج المحدد في ملف التكوين.
ضمن مجلد الإخراج ، يتم إنشاء مجلد واحد لكل مجموعة بيانات في ملف التكوين ، ولكل مجموعة بيانات ، يمكن العثور على عمليات تكامل في دليل ints . سيكون هناك مجموعة واحدة من ملفات الإخراج لكل عينة.
لكل عينة ، هناك عدد من ملفات الإخراج ، والتي قد تكون ذات أهمية بالنسبة لحالات الاستخدام المعينة.
<sample>.<host>.<virus>.integrations.txt : معلومات حول جميع الوصلات المكتشفة ، بغض النظر عما إذا كانت قد اجتازت المرشحات أم لا<sample>.<host>.<virus>.integrations.post.txt : معلومات حول الوصلات المكتشفة التي مرت بجميع المرشحات<sample>.<host>.<virus>.integrations.post.unique.txt : معلومات حول الوصلات المكتشفة التي مرت بجميع المرشحات ولها موقع لا لبس فيه في كل من المضيف والفيروس/فيروس<sample>.<host>.<virus>.integrations.post.unique.merged.txt : نتيجة دمج الوصلات المتداخلة أو الوصلات مع نفس الإحداثيات في كل من المضيف والفيروس/المتجه. يتم الاندماج مع تقاطعات التكامل التي لها موقع لا لبس فيه في كل من المضيف والفيروس/المتجه.<sample>.<host>.<virus>.integrations.post.host_ambig.txt : معلومات حول الوصلات المكتشفة التي اجتاز جميع المرشحات ولديها متجه/فيروس لا لبس فيه ولكن موقع غامض في المضيف<sample>.<host>.<virus>.integrations.post.virus_ambig.txt : معلومات حول الوصلات المكتشفة التي مرت بجميع المرشحات ولديها مضيف موقع لا لبس فيه ولكن موقع غامض في Vector/Virus<sample>.<host>.<virus>.integrations.post.both_ambig.txt : معلومات حول الوصلات المكتشفة التي اجتاز جميع المرشحات ولها موقع غامض في كل من المضيف والمتجه/الفيروس تعطي ملفات الإخراج موقع التكامل المحدد ، وخصائصها. يتم تحديد إحداثيات تقاطعات التكامل من حيث قواعدها الغامضة. أي غالبًا ما تكون هناك فجوة أو تداخل بين الأجزاء المضيفة والأجزاء الفيروسية للقراءة: 
نظرًا لأن موقع التكامل لا يمكن تحديده بشكل فريد في هذه الحالة ، فإن إخراج الإحداثيات لهذه القواعد في جينوم المضيف والفيروس/الفيروس كموقع للتكامل.
باستثناء ملف إخراج الكتلة المدمج ، تحتوي جميع الملفات على هذه الأعمدة:
gap أو overlap لقراءة أكثر وضوحًا (المضيف والأجزاء الفيروسية المحددة في نفس القراءة) ، يكون الوصل clean إذا لم يكن له قواعد غامضة. بدلاً من ذلك ، إذا تم تحديد jucntion في زوج متناقض ، فإن OverlapType discordanthv ) أو فيروس/مضيف ( vh )+ أو -bwa ) من محاذاة المضيفbwa ) من المحاذاة الفيروسيةOverlapType gapchimeric ، زوج discordant أم أنه تكامل short لوحظ فيه كلا الوصلات في نفس القراءةbwa )bwa )'xxx' لإعادة إنتاج الأشكال في مخطوطة ISLING ، راجع ReadMe في دليل benchmarking .