Organisation: University of New South Wales (UNSW) AI4Science & GreenDynamics AI
Darwin ist ein Open-Source-Projekt, das sich dem Vortraining und der Feinabstimmung des LLaMA-Modells anhand wissenschaftlicher Literatur und Datensätze widmet. Darwin wurde speziell für den wissenschaftlichen Bereich mit Schwerpunkt auf Materialwissenschaften, Chemie und Physik entwickelt und integriert strukturiertes und unstrukturiertes wissenschaftliches Wissen, um die Wirksamkeit von Sprachmodellen in der wissenschaftlichen Forschung zu verbessern.
Nutzungs- und Lizenzhinweise : Darwin ist lizenziert und nur für Forschungszwecke bestimmt. Der Datensatz ist unter CC BY NC 4.0 lizenziert und erlaubt eine nichtkommerzielle Nutzung. Mit diesem Datensatz trainierte Modelle sollten nicht außerhalb von Forschungszwecken verwendet werden. Der Gewichtsunterschied steht ebenfalls unter der CC BY NC 4.0-Lizenz
[20.11.2024]
Wichtigste Erfolge
Einblicke in die Modellleistung
Datenstrategien und Erkenntnisse
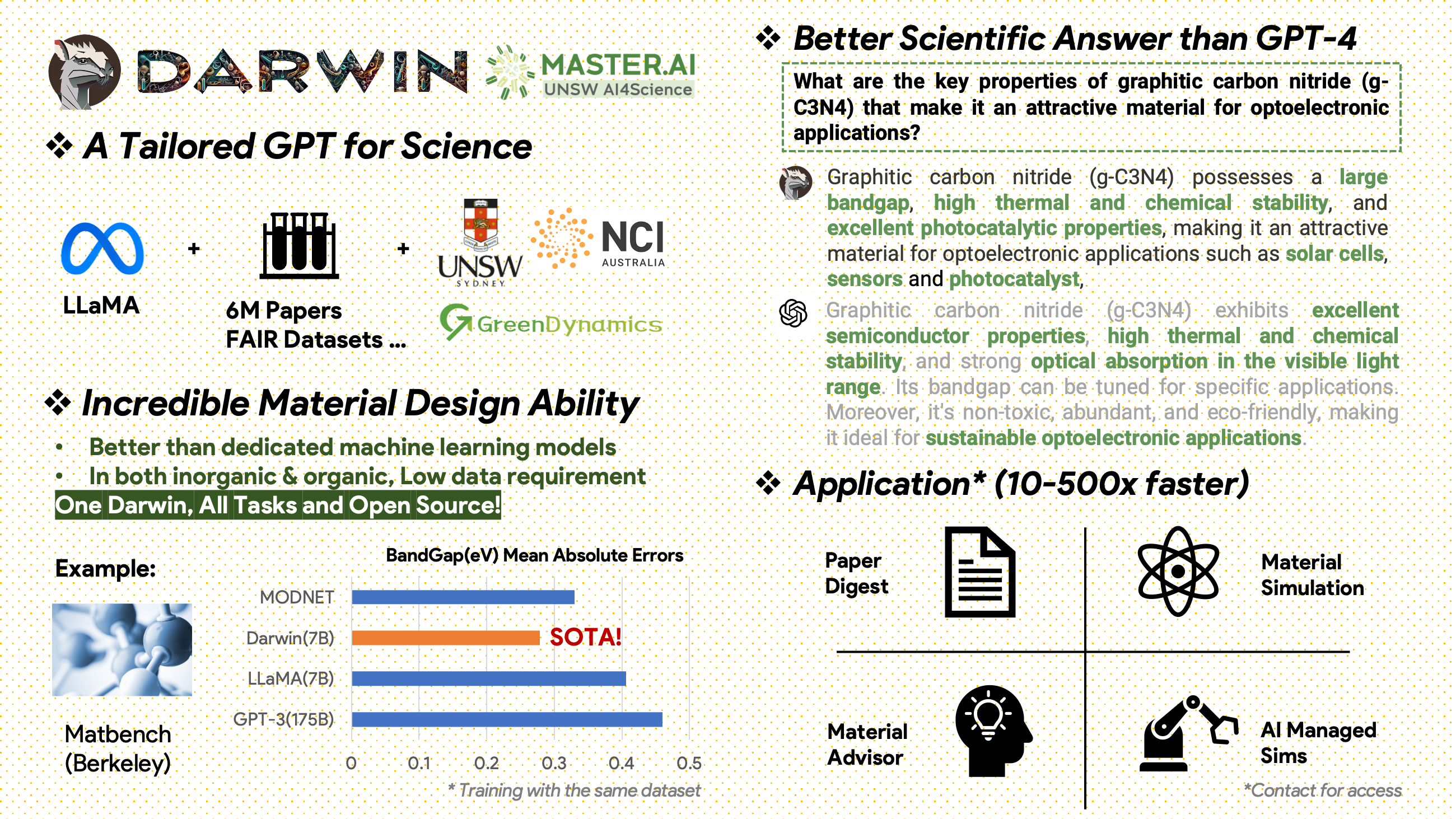
[15.02.2024] SOTA in MatBench von Material Projects: DARWIN ist das SOTA-Modell für experimentelle Bandlückenvorhersageaufgaben und metallische Klassifizierungsaufgaben, besser als fein abgestimmte GPT3.5- und dedizierte ML-Modelle. https://matbench.materialsproject.org/Leaderboards%20Per-Task/matbench_v0.1_matbench_expt_gap/
☆ [15.09.2023]Google Colab-Version verfügbar: Probieren Sie unseren DARWIN mit Google Colab aus: inference.ipynb
Darwin basiert auf dem 7B LLaMA-Modell und wird anhand von über 100.000 Datenpunkten zur Befehlsfolge trainiert, die vom Darwin Scientific Instruction Generator (SIG) aus verschiedenen wissenschaftlichen FAIR-Datensätzen und einem Literaturkorpus generiert wurden. Indem er sich auf die sachliche Richtigkeit der Modellantworten konzentriert, stellt Darwin einen bedeutenden Schritt hin zur Nutzung großer Sprachmodelle (LLMs) für wissenschaftliche Entdeckungen dar. Vorläufige menschliche Bewertungen deuten darauf hin, dass Darwin 7B GPT-4 bei wissenschaftlichen Fragen und Antworten übertrifft und GPT-3 bei der Lösung chemischer Probleme (wie gptChem) feinabgestimmt hat.
Wir entwickeln Darwin aktiv für komplexere Experimente im wissenschaftlichen Bereich und integrieren Darwin auch in LangChain, um komplexere wissenschaftliche Aufgaben zu lösen (z. B. einen privaten Forschungsassistenten für Personalcomputer).
Bitte beachten Sie, dass sich Darwin noch in der Entwicklung befindet und viele Einschränkungen behoben werden müssen. Am wichtigsten ist, dass wir Darwin noch für maximale Sicherheit optimieren müssen. Wir ermutigen Benutzer, bedenkliches Verhalten zu melden, um die Sicherheit und ethischen Überlegungen des Modells zu verbessern.
DEMO-LINK
Installieren Sie zunächst die Anforderungen:
pip install -r requirements.txtLaden Sie die Checkpoints der Darwin-7B-Gewichte von onedrive herunter. Sobald Sie das Modell heruntergeladen haben, können Sie unsere Demo ausprobieren:
python inference.py < your path to darwin-7b >Bitte beachten Sie, dass für die Inferenz mindestens 10 GB GPU-Speicher für Darwin 7B erforderlich sind.
Um unseren Darwin-7b mit verschiedenen Datensätzen weiter zu verfeinern, finden Sie unten einen Befehl, der auf einer Maschine mit 4 A100 80G-GPUs funktioniert.
torchrun --nproc_per_node=8 --master_port=1212 train.py
--model_name_or_path < your path to darwin-7b >
--data_path < your path to dataset >
--bf16 True
--output_dir < your output dir >
--num_train_epochs 3
--per_device_train_batch_size 1
--per_device_eval_batch_size 1
--gradient_accumulation_steps 1
--evaluation_strategy " no "
--save_strategy " steps "
--save_steps 500
--save_total_limit 1
--learning_rate 2e-5
--weight_decay 0.
--warmup_ratio 0.03
--lr_scheduler_type " cosine "
--logging_steps 1
--fsdp " full_shard auto_wrap "
--fsdp_transformer_layer_cls_to_wrap ' LlamaDecoderLayer '
--tf32 FalseUnsere Daten stammen aus zwei Hauptquellen:
Nach dem Jahr 2000 wurde ein Rohliteraturkorpus mit 6,0 Millionen Artikeln zu Materialwissenschaften, Chemie und Physik veröffentlicht. Zu den Herausgebern gehören ACS, RSC, Springer Nature, Wiley und Elsevier. Wir danken ihnen für ihre Unterstützung.
FAIR-Datensätze – Wir haben Daten aus 16 FAIR-Datensätzen gesammelt.
Wir haben Darwin-SIG entwickelt, um wissenschaftliche Anweisungen zu generieren. Es kann sich lange Texte aus vollständigen Literaturtexten merken (durchschnittlich etwa 5.000 Wörter) und Frage-und-Antwort-Daten (Q&A) basierend auf wissenschaftlichen Literaturschlüsselwörtern generieren (von der Web of Science-API).
Hinweis: Sie könnten zur Generierung auch GPT3.5 oder GPT-4 verwenden, diese Optionen könnten jedoch kostspielig sein.
Bitte beachten Sie, dass wir den Trainingsdatensatz aufgrund von Vereinbarungen mit den Herausgebern nicht weitergeben können.
Dieses Projekt ist eine Gemeinschaftsarbeit von:
UNSW & GreenDynamics: Tong Xie, Shaozhou Wang
UNSW: Imran Razzak, Cody Huang
USYD & DARE Center: Clara Grazian
GreenDynamics: Yuwei Wan,Yixuan Liu
Bram Hoex und Wenjie Zhang von UNSW Engineering haben alle beraten.
Wenn Sie die Daten oder den Code aus diesem Repository in Ihrer Arbeit verwenden, zitieren Sie diese bitte entsprechend.
DAWRIN Foundational Large Language Model und Semi-Self-Instruct Fine Tuning
@misc{xie2023darwin,
title={DARWIN Series: Domain Specific Large Language Models for Natural Science},
author={Tong Xie and Yuwei Wan and Wei Huang and Zhenyu Yin and Yixuan Liu and Shaozhou Wang and Qingyuan Linghu and Chunyu Kit and Clara Grazian and Wenjie Zhang and Imran Razzak and Bram Hoex},
year={2023},
eprint={2308.13565},
archivePrefix={arXiv},
primaryClass={cs.CL}
}
Feinabstimmung von GPT-3 und LLaMA für Material Discovery (Einzelaufgabentraining)
@article{xie2023large,
title={Large Language Models as Master Key: Unlocking the Secrets of Materials Science},
author={Xie, Tong and Wan, Yuwei and Zhou, Yufei and Huang, Wei and Liu, Yixuan and Linghu, Qingyuan and Wang, Shaozhou and Kit, Chunyu and Grazian, Clara and Zhang, Wenjie and others},
journal={Available at SSRN 4534137},
year={2023}
}
Dieses Projekt hat sich auf die folgenden Open-Source-Projekte bezogen:
Besonderer Dank geht an NCI Australia für die HPC-Unterstützung.
Wir erweitern kontinuierlich das Entwicklungsteam von Darwin. Begleiten Sie uns auf dieser spannenden Reise, die wissenschaftliche Forschung mit KI voranzutreiben!
Für Doktoranden- oder PostDoc-Stellen wenden Sie sich bitte an [email protected] oder [email protected], um weitere Informationen zu erhalten.
Für andere Positionen besuchen Sie bitte www.greendynamics.com.au