Bitte beachten Sie, dass dieses Repository nur für Archivzwecke dient. Die eigenständigen ANCOM-Funktionen in diesem Repository werden nicht mehr gepflegt. Benutzern wird dringend empfohlen, die Funktionen ancom oder ancombc in unserem ANCOMBC R-Paket zu verwenden. Für Fehlerberichte gehen Sie bitte zum ANCOMBC-Repository.
Der aktuelle Code implementiert ANCOM in Querschnitts- und Längsschnittdatensätzen und ermöglicht gleichzeitig die Verwendung von Kovariaten. Damit der R-Code ausgeführt werden kann, müssen die folgenden Bibliotheken enthalten sein:
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )Wir haben die Methodik von ANCOM-II als Vorverarbeitungsschritt übernommen, um mit verschiedenen Arten von Nullstellen umzugehen, bevor wir eine Differentialhäufigkeitsanalyse durchführen.
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : Datenrahmen oder Matrix, die die beobachtete OTU/SV-Tabelle mit Taxa in Zeilen ( rownames ) und Proben in Spalten ( colnames ) darstellt. Beachten Sie, dass dies die Tabelle mit der absoluten Häufigkeit ist. Wandeln Sie sie nicht in eine Tabelle mit relativer Häufigkeit um (in der die Spaltensummen gleich 1 sind).meta_data : Datenrahmen oder Matrix aller interessierenden Variablen und Kovariaten.sample_var : Zeichen. Der Name der Spalte, in der Proben-IDs gespeichert sind.group_var : Charakter. Der Name des Gruppenindikators. group_var wird zum Erkennen struktureller Nullen und Ausreißer benötigt. Die Definitionen verschiedener Nullpunkte (Strukturnullpunkt, Ausreißernullpunkt und Stichprobennullpunkt) finden Sie in ANCOM-II.out_cut Numerischer Bruch zwischen 0 und 1. Für jedes Taxon werden Beobachtungen mit einem Anteil der Mischungsverteilung kleiner als out_cut als Ausreißer-Nullen erkannt; während Beobachtungen mit einem Anteil der Mischungsverteilung größer als 1 - out_cut als Ausreißerwerte erkannt werden.zero_cut : Numerischer Bruch zwischen 0 und 1. Taxa mit einem Anteil an Nullen größer als zero_cut werden nicht in die Analyse einbezogen.lib_cut : Numerisch. Proben mit einer Bibliotheksgröße kleiner als lib_cut werden nicht in die Analyse einbezogen.neg_lb : Logisch. TRUE gibt an, dass ein Taxon anhand seiner asymptotischen Untergrenze in der entsprechenden Versuchsgruppe als strukturelle Null klassifiziert würde. Genauer gesagt bedeutet neg_lb = TRUE , dass Sie beide in Abschnitt 3.2 von ANCOM-II genannten Kriterien verwenden, um strukturelle Nullen zu erkennen. Andernfalls verwendet neg_lb = FALSE nur die Gleichung 1 in Abschnitt 3.2 von ANCOM-II zur Deklaration struktureller Nullen. feature_table : Ein Datenrahmen einer vorverarbeiteten OTU-Tabelle.meta_data : Ein Datenrahmen vorverarbeiteter Metadaten.structure_zeros : Eine Matrix besteht aus 0 und 1, wobei 1 angibt, dass das Taxon in der entsprechenden Gruppe als strukturelle Null identifiziert wird.ANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : Datenrahmen, der die OTU/SV-Tabelle mit Taxa in Zeilen ( rownames ) und Beispielen in Spalten ( colnames ) darstellt. Es kann der Ausgabewert von feature_table_pre_process sein. Beachten Sie, dass dies die Tabelle mit der absoluten Häufigkeit ist. Wandeln Sie sie nicht in eine Tabelle mit relativer Häufigkeit um (in der die Spaltensummen gleich 1 sind).meta_data : Datenrahmen von Variablen. Kann der Ausgabewert von feature_table_pre_process sein.struc_zero : Eine Matrix besteht aus 0 und 1, wobei 1 angibt, dass das Taxon in der entsprechenden Gruppe als strukturelle Null identifiziert wird. Kann der Ausgabewert von feature_table_pre_process sein.main_var : Charakter. Der Name der wichtigsten interessierenden Variablen. ANCOM v2.1 unterstützt derzeit kategoriale main_var .p_adjust_method : Zeichen. Angeben der Methode zum Anpassen von p-Werten für mehrere Vergleiche. Der Standardwert ist „BH“ (Benjamini-Hochberg-Verfahren).alpha : Signifikanzniveau. Der Standardwert ist 0,05.adj_formula : Zeichenfolge, die die Formel für die Anpassung darstellt (siehe Beispiel).rand_formula : Zeichenfolge, die die Formel für Zufallseffekte in lme darstellt. Einzelheiten finden Sie ?lme .lme_control : Eine Liste, die Kontrollwerte für die LME-Anpassung angibt. Einzelheiten finden Sie unter ?lmeControl . 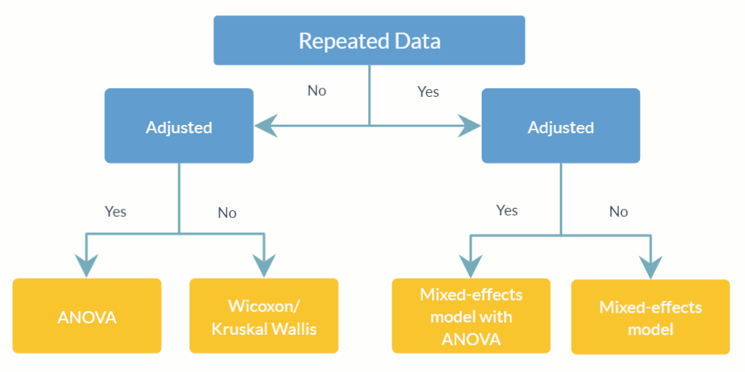
out : Ein Datenrahmen mit der W Statistik für jedes Taxa und nachfolgende Spalten, die logische Indikatoren dafür sind, ob eine OTU oder ein Taxon unter einer Reihe von Cutoffs (0,9, 0,8, 0,7 und 0,6) unterschiedlich häufig vorkommt. detected_0.7 wird häufig verwendet. Sie können jedoch detected_0.8 oder detected_0.9 wählen, wenn Sie bei Ihren Ergebnissen konservativer vorgehen möchten (kleinere FDR), oder detected_0.6 verwenden, wenn Sie mehr Entdeckungen untersuchen möchten (größere Leistung).
fig .: Ein ggplot Objekt des Vulkanplots.
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Hier möchten wir wissen, ob es über verschiedene delivery hinweg einige strukturelle Nullstellen gibt library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var . Hier möchten wir wissen, ob es über verschiedene delivery hinweg einige strukturelle Nullstellen gibt library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Hier möchten wir wissen, ob es über verschiedene delivery hinweg einige strukturelle Nullstellen gibt library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )