Este paquete proporciona modelos U-net entrenados para la segmentación pulmonar. Por ahora, hay cuatro modelos disponibles:
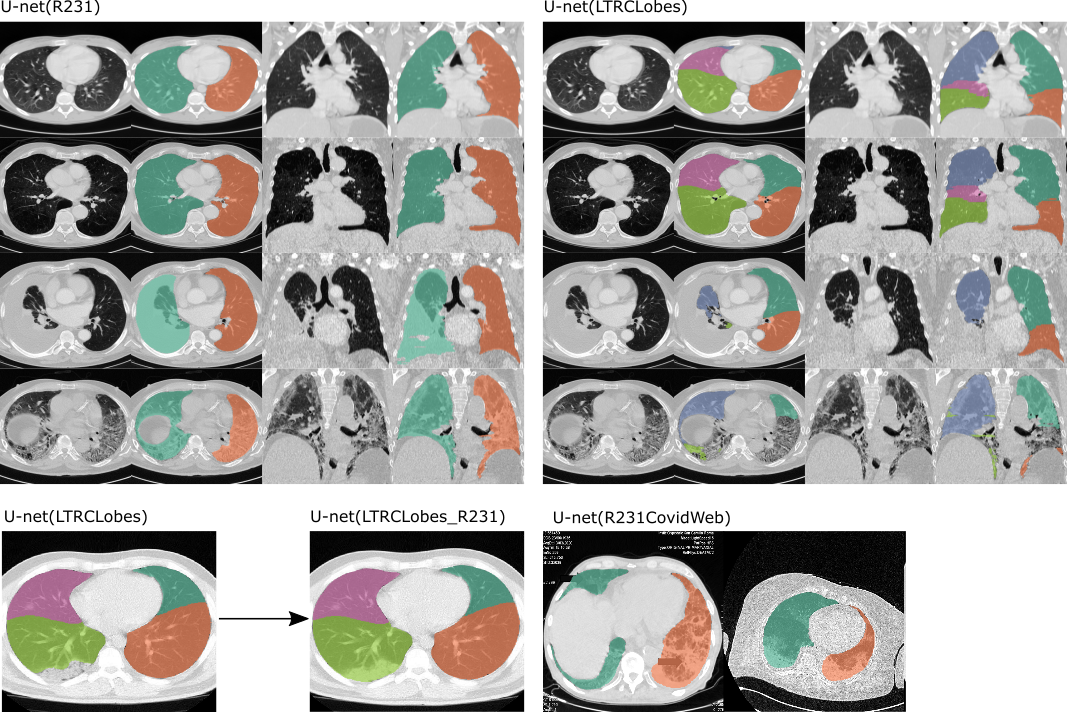
U-net (R231): este modelo se entrenó en un conjunto de datos grande y diverso que cubre una amplia gama de variabilidad visual. El modelo realiza la segmentación en cortes individuales, extrae el pulmón derecho-izquierdo por separado, incluye bolsas de aire, tumores y derrames. La tráquea no se incluirá en la segmentación pulmonar. https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRCLobes): este modelo se entrenó en un subconjunto del conjunto de datos LTRC. El modelo realiza la segmentación de lóbulos pulmonares individuales, pero produce un rendimiento limitado cuando hay patologías densas o cuando las fisuras no son visibles en cada corte.
U-net(LTRCLobes_R231): esto ejecutará el modelo R231 y LTRCLobes y fusionará los resultados. Los falsos negativos de LTRCLobes se completarán con predicciones R231 y se asignarán a una etiqueta vecina. Se eliminarán los falsos positivos de LTRCLobes. El proceso de fusión requiere una gran cantidad de cálculos y, según los datos y los resultados, puede tardar hasta varios minutos por volumen.
U-net(R231CovidWeb)
Ejemplos de los dos modelos aplicados . Izquierda: U-net (R231), distinguirá entre pulmón izquierdo y derecho e incluirá áreas muy densas como derrames (tercera fila), tumores o fibrosis grave (cuarta fila). Derecha: U-net(LTRLobes), distinguirá entre lóbulos pulmonares pero no incluirá áreas muy densas. LTRCLobes_R231 fusionará los resultados de LTRCLobes y R231. R231CovidWeb está entrenado con datos adicionales de COVID-19.
Semántica de salida :
Dos modelos de etiquetas (Izquierda-Derecha):
1 = pulmón derecho
2 = pulmón izquierdo
Cinco modelos de etiquetas (lóbulos pulmonares):
1 = Lóbulo superior izquierdo
2 = Lóbulo inferior izquierdo
3 = Lóbulo superior derecho
4 = Lóbulo medio derecho
5 = Lóbulo inferior derecho
Para obtener más investigaciones interesantes sobre datos de TC de pulmón, consulte el sitio web de nuestro grupo de investigación: https://www.cir.meduniwien.ac.at/research/lung/
Si utiliza este código o uno de los modelos entrenados en su trabajo, consulte:
Hofmanninger, J., Prayer, F., Pan, J. et al. La segmentación pulmonar automática en imágenes de rutina es principalmente un problema de diversidad de datos, no un problema de metodología. Eur Radiol Exp 4, 50 (2020). https://doi.org/10.1186/s41747-020-00173-2
Este artículo contiene una descripción detallada del conjunto de datos utilizado, una evaluación exhaustiva del modelo U-net(R231) y una comparación con los métodos de referencia.
pip install lungmask
o
pip install git+https://github.com/JoHof/lungmask
En Windows, dependiendo de su configuración, puede que sea necesario instalar Torch de antemano: https://pytorch.org
El tiempo de ejecución entre la inferencia compatible solo con CPU y GPU varía mucho. Usando la GPU, procesar un volumen toma solo unos segundos, usando solo la CPU tomará varios minutos. Para utilizar la GPU, asegúrese de que la instalación de su antorcha sea compatible con CUDA. En caso de errores de cuda sin memoria, reduzca el tamaño del lote a 1 con el argumento opcional --batchsize 1
lungmask INPUT OUTPUT
Si INPUT apunta a un archivo, el archivo será procesado. Si INPUT apunta a un directorio, se buscará la serie DICOM en el directorio. El mayor volumen encontrado (en términos de número de vóxeles) se utilizará para calcular la máscara pulmonar. SALIDA es el nombre del archivo de salida. Todos los formatos ITK son compatibles.
Elige un modelo:
La red U (R231) se utilizará de forma predeterminada. Sin embargo, puede especificar un modelo alternativo como LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
Para opciones adicionales escriba:
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image tiene que ser un objeto SimpleITK.
Cargue un modelo alternativo así:
inferer = LMInferer(modelname="R231CovidWeb")
Para utilizar la capacidad de fusión de modelos (p. ej., LTRCLobes_R231), utilice:
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
A partir de la versión 0.2.9, se admiten numerosas matrices como volúmenes de entrada. Este modo supone que la matriz numpy de entrada tiene el siguiente formato para cada eje:
El modelo funciona únicamente con cortes completos. El corte a procesar debe mostrar el pulmón completo y el pulmón debe estar rodeado de tejido para poder segmentarse. Sin embargo, el modelo es bastante estable en casos con un campo de visión recortado siempre que el pulmón esté rodeado de tejido.
lungmask INPUT OUTPUT --modelname R231CovidWeb
El modelo U-net(R231) normal funciona muy bien para tomografías computarizadas de COVID-19. Sin embargo, las colecciones de cortes e informes de casos de la web a menudo se recortan, anotan o codifican en formatos de imagen regulares para que los valores originales de la unidad Hounsfield (HU) solo puedan estimarse. Los datos de entrenamiento del modelo U-net (R231CovidWeb) se aumentaron con cortes de COVID-19 que se asignaron desde formatos de imágenes regulares a HU. Los datos fueron recopilados y preparados por MedSeg (http://medicalsegmentation.com/covid19/). Si bien el U-net normal (R231) mostró muy buenos resultados para estas imágenes, puede haber casos en los que este modelo produzca segmentaciones ligeramente mejoradas. Tenga en cuenta que debe asignar imágenes a HU cuando utilice imágenes de la web. Esta publicación de blog describe cómo puede hacerlo. Alternativamente, puede configurar la etiqueta --noHU . 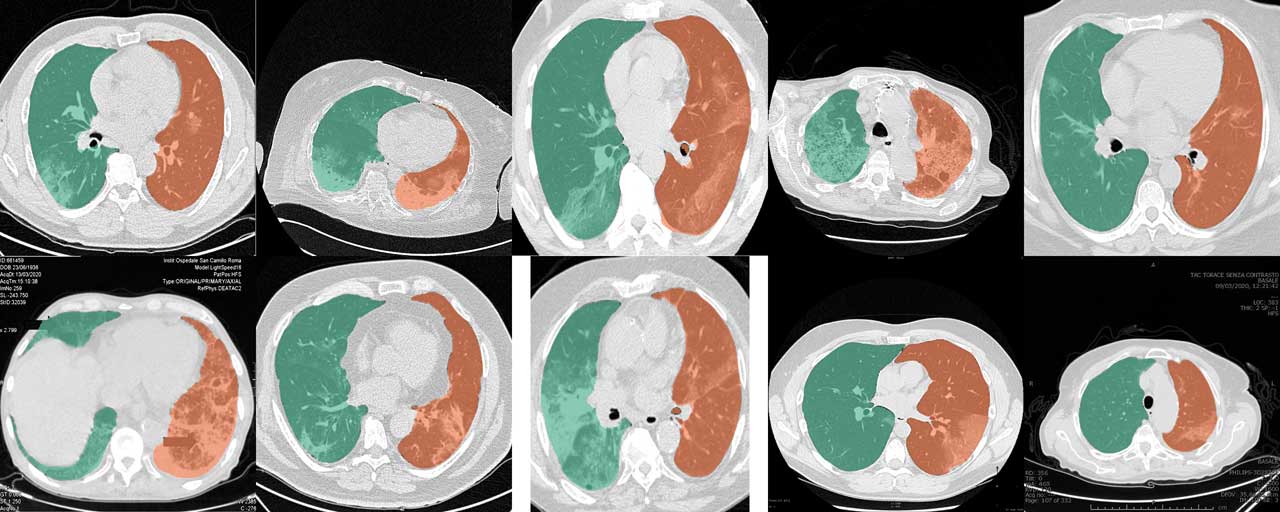
Esta característica sólo está disponible en las versiones entre 0.2.5 y 0.2.14. A partir de la versión 0.2.5 estas imágenes son compatibles. Utilice la etiqueta --noHU si procesa imágenes que no están codificadas en HU. Tenga en cuenta que los modelos fueron entrenados en tomografías computarizadas adecuadas codificadas en HU. Los resultados de imágenes recortadas, anotadas y desplazadas de muy alta y muy baja intensidad pueden no ser muy confiables. Cuando se utiliza la etiqueta --noHU , solo se pueden procesar sectores individuales.