La mise en œuvre de DiffAbXL a été comparée dans l'article : Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs.
| Rang | Modèles | Absci HER2 | Nature | Cible AZ-2 | Avenue ? | ||
|---|---|---|---|---|---|---|---|
| Tir zéro | Contrôle SPR | HEL | ELLE2 | ||||
| 1 | DiffAbXL-A-DN | 0,43 | 0,22 | 0,62 | 0,37 | 0,41 | 0,41 |
| 2 | DiffAbXL-A-SG | 0,46 | 0,22 | 0,64 | -0,38 | 0,43 | 0,274 |
| 3 | DiffAbXL-H3-DN | 0,49 | 0 | 0,52 | -0,08 | 0,37 | 0,26 |
| 4 | IgBlend (struct. uniquement) | 0,40 | 0,21 | 0,54 | -0,30 | 0,31 | 0,232 |
| 5 | Antipliage | 0,43 | 0,22 | 0,4 | -0,47 | 0,38 | 0,192 |
| 6 | DiffAbXL-H3-SG | 0,48 | 0 | 0,4 | -0,41 | 0,29 | 0,152 |
| 7 | MES | 0,29 | 0 | 0 | 0,18 | 0,27 | 0,148 |
| 8 | Abdiff | 0,34 | 0,21 | 0 | -0,14 | 0,22 | 0,126 |
| 9 | AbLang2 | 0,3 | 0 | 0 | -0,07 | 0,36 | 0,118 |
| 10 | IgBlend (séquence uniquement) | 0,27 | 0 | 0 | -0,1 | 0,36 | 0,106 |
| 11 | AbLang | 0,3 | 0 | 0 | -0,13 | 0,35 | 0,104 |
| 12 | dyMEAN | 0,37 | 0,15 | 0 | 0 | 0 | 0,104 |
| 13 | AbX | 0,28 | 0,19 | 0 | 0 | 0 | 0,094 |
| 14 | AntiBERTY | 0,26 | 0 | 0 | -0,17 | 0,35 | 0,088 |
| 15 | SIGNIFIER | 0,36 | 0 | 0 | 0,02 | 0 | 0,076 |
| 16 | MES-FI | 0 | -0,27 | 0 | -0,53 | 0,42 | -0,076 |
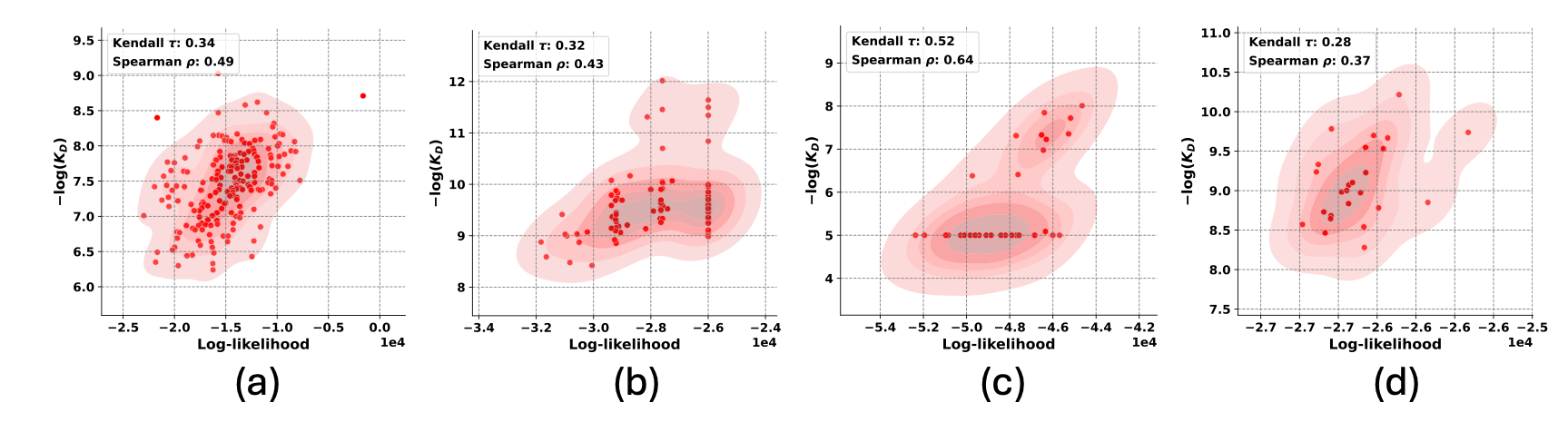
Figure 1 : Résultats pour DiffAbXL : a) DiffAbXL-H3-DN pour les données Absci zero-shot HER2 b) DiffAbXL-A-SG pour AZ Target-2, c) DiffAbXL-A-SG pour Nature HEL, d) DiffAbXL- A-DN pour Nature HER2.
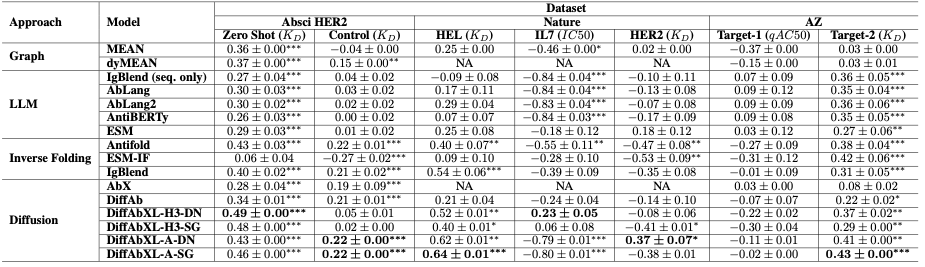
Tableau 1 : Résumé des résultats de la corrélation de Spearman. Abréviations : DN : mode De Novo, SG : mode Structure Guidance, NA : épitope ou structure complexe requis, mais non disponible. *, **, *** indiquent des valeurs p inférieures à 0,05, 0,01 et 1e-4 respectivement.
Pour nous permettre de comparer plus facilement votre modèle, nous vous recommandons d'implémenter une interface en tant que méthode Python dans une classe que nous pouvons facilement intégrer à notre pipeline d'évaluation. La méthode doit accepter les entrées suivantes :
La méthode doit renvoyer un dictionnaire contenant :
Voici un modèle de base en Python pour implémenter cette interface :
def benchmark ( self , sequences , structure = None , mask = None , ** kwargs ):
"""
Benchmark the model on provided antibody sequences and structures.
Parameters:
sequences (list of str): List of antibody sequences.
structure (optional): Path to a PDB file. Currently, only one PDB file is provided per target dataset.
The PDB file may contain either just the antibody or an antibody-antigen complex,
depending on the dataset.
mask (optional): Binary list or array indicating the regions of interest in the sequences for metric calculations.
kwargs (optional): Additional parameters required by the model.
Returns:
dict: A dictionary containing log-likelihood scores and other relevant metrics.
"""
passVeuillez vous assurer que votre modèle génère les scores de log-vraisemblance dans un format que nous pouvons utiliser directement pour comparer les conceptions de séquences d'anticorps. Cela nous aidera à comparer efficacement les performances de votre modèle sur nos ensembles de données.
Il existe un fichier de configuration : sabdab.yaml, qui peut être utilisé pour modifier n'importe quel paramètre. Vous pouvez entraîner le modèle en utilisant :
python train.py # For training.
- train.py
-src
|-model.py
- configuration
|-sabdab.yaml
- utilitaires
|-load_data.py
|-arguments.py
|-model_utils.py
|-loss_functions.py
...
- données
|-elle2
...
La pondération et les biais peuvent être utilisés pour suivre les expériences. Il est désactivé par défaut, mais peut être activé en modifiant l'option dans le fichier de configuration dans ./config/sabdab.yaml
@article {Ucar2024.10.07.617023,
author = {Ucar, Talip and Malherbe, Cedric and Gonzalez Hernandez, Ferran},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
elocation-id = {2024.10.07.617023},
year = {2024},
doi = {10.1101/2024.10.07.617023},
publisher = {Cold Spring Harbor Laboratory},
URL = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023},
eprint = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023.full.pdf},
journal = {bioRxiv}
}
Si vous utilisez DiffAbXL dans vos propres études et travail, veuillez le citer en utilisant ce qui suit :
@Misc{talip_ucar_2024_DiffAbXL,
author = {Talip Ucar},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
URL = {https://github.com/AstraZeneca/DiffAbXL},
month = {October},
year = {since 2024}
}