L'ensemble des données bien traitées et prêtes pour DL de 109 sujets de l'EEGMMIDB sont téléchargées !
Ce didacticiel contient des codes de notebook Python et Jupyter implémentables et des ensembles de données de référence pour apprendre à reconnaître les signaux cérébraux sur la base de modèles d'apprentissage en profondeur. Ce tutoriel associe notre enquête sur les signaux cérébraux non invasifs basés sur DL et notre livre sur le BCI basé sur DL : Représentations, algorithmes et applications.
| Distribution sur les signaux | Distribution sur les modèles DL |
|---|---|
 |  |
Une attention particulière a été accordée aux études de pointe sur l'apprentissage profond pour la recherche BCI basée sur l'EEG en termes d'algorithmes. Plus précisément, nous introduisons un certain nombre d'algorithmes et de cadres avancés d'apprentissage en profondeur visant à résoudre plusieurs problèmes majeurs de la BCI, notamment l'apprentissage robuste de la représentation des signaux cérébraux, la classification inter-scénarios et la classification semi-supervisée.
En outre, plusieurs nouveaux prototypes de systèmes BCI basés sur l'apprentissage profond sont proposés, qui mettent en lumière des applications réelles telles que l'authentification, la reconstruction visuelle, l'interprétation du langage et le diagnostic de troubles neurologiques. De telles applications peuvent bénéficier considérablement à la fois aux personnes en bonne santé et aux personnes handicapées dans la vie réelle.
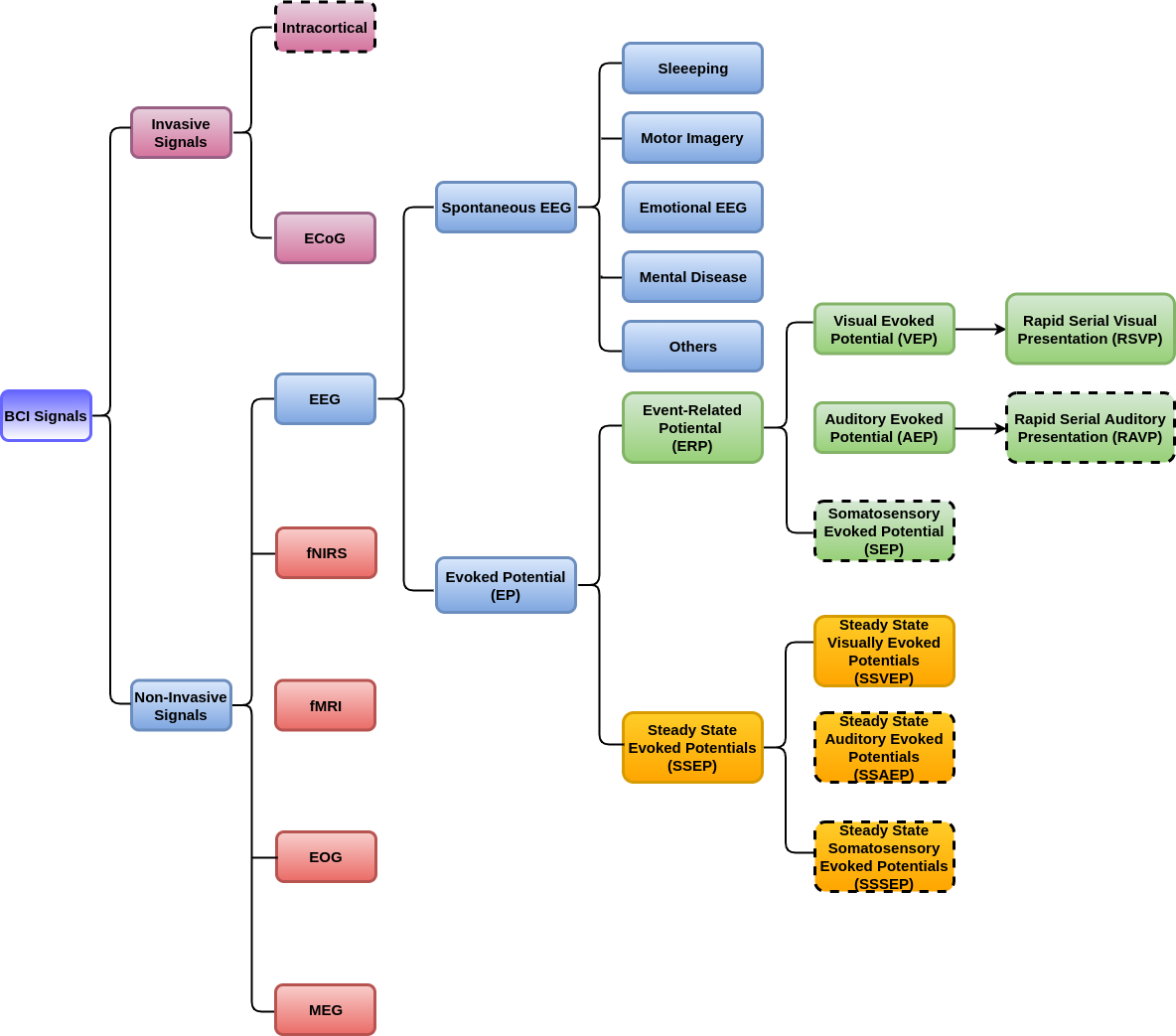
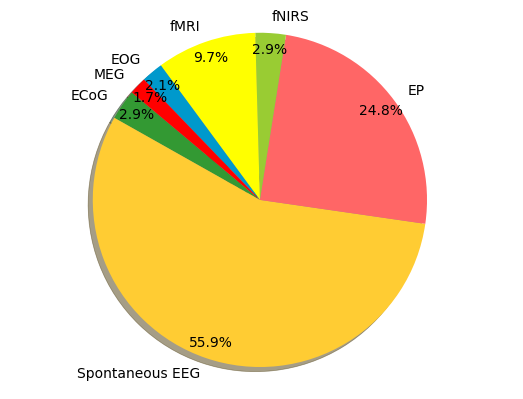
La collecte de signaux cérébraux est coûteuse à la fois financièrement et temporellement. Nous explorons en profondeur les ensembles de données de référence applicables à la recherche sur les signaux de pluie et fournissons 31 ensembles de données publiques avec des liens de téléchargement couvrant la plupart des types de signaux cérébraux.
| Signaux cérébraux | Ensemble de données | #-Sujet | #-Cours | Taux d'échantillonnage (Hz) | #-Canaux | Lien de téléchargement |
|---|---|---|---|---|---|---|
| FM EcoG | BCI-C IV, Ensemble de données IV | 3 | 5 | 1000 | 48 -- 64 | Lien |
| MI EcoG | BCI-C III Ensemble de données I | 1 | 2 | 1000 | 64 | Lien |
| EEG endormi | Télémétrie Sommeil-EDF | 22 | 6 | 100 | 2 EEG, 1 EOG, 1 EMG | Lien |
| EEG endormi | Sommeil-EDF : Cassette | 78 | 6 | 100, 1 | 2 EEG (100 Hz), 1 EOG (100 Hz), 1 EMG (1 Hz) | Lien |
| EEG endormi | MASSE-1 | 53 | 5 | 256 | 17/19 EEG, 2 EOG, 5 EMG | Lien |
| EEG endormi | MASSE-2 | 19 | 6 | 256 | 19 EEG, 4 EOG, 1EMG | Lien |
| EEG endormi | MASSE-3 | 62 | 5 | 256 | 20 EEG, 2 EOG, 3 EMG | Lien |
| EEG endormi | MASSE-4 | 40 | 6 | 256 | 4 EEG, 4 EOG, 1 EMG | Lien |
| EEG endormi | MASSE-5 | 26 | 6 | 256 | 20 EEG, 2 EOG, 3 EMG | Lien |
| EEG endormi | SHHS | 5804 | N / A | 125, 50 | 2 EEG (125 Hz), 1EOG (50 Hz), 1 EMG (125 Hz) | Lien |
| Saisie EEG | CHB-MIT | 22 | 2 | 256 | 18 | Lien |
| Saisie EEG | TUH | 315 | 2 | 200 | 19 | Lien |
| MI-EEG | EEGMMI | 109 | 4 | 160 | 64 | Lien |
| MI-EEG | BCI-C II, ensemble de données III | 1 | 2 | 128 | 3 | Lien |
| MI-EEG | BCI-C III, Ensemble de données III a | 3 | 4 | 250 | 60 | Lien |
| MI-EEG | BCI-C III, ensemble de données III b | 3 | 2 | 125 | 2 | Lien |
| MI-EEG | BCI-C III, ensemble de données IV a | 5 | 2 | 1000 | 118 | Lien |
| MI-EEG | BCI-C III, ensemble de données IV b | 1 | 2 | 1001 | 119 | Lien |
| MI-EEG | BCI-C III, ensemble de données IV c | 1 | 2 | 1002 | 120 | Lien |
| MI-EEG | BCI-C IV, Ensemble de données I | 7 | 2 | 1000 | 64 | Lien |
| MI-EEG | BCI-C IV, Ensemble de données II a | 9 | 4 | 250 | 22 EEG, 3 EOG | Lien |
| MI-EEG | BCI-C IV, ensemble de données II b | 9 | 2 | 250 | 3 EEG, 3 EOG | Lien |
| EEG émotionnel | AMIGOS | 40 | 4 | 128 | 14 | Lien |
| EEG émotionnel | GRAINE | 15 | 3 | 200 | 62 | Lien |
| EEG émotionnel | DEAP | 32 | 4 | 512 | 32 | Lien |
| D'autres EEG | Ouvrir le MIIR | 10 | 12 | 512 | 64 | Lien |
| PEV | BCI-C II, ensemble de données IIb | 1 | 36 | 240 | 64 | Lien |
| PEV | BCI-C III, ensemble de données II | 2 | 26 | 240 | 64 | Lien |
| IRMf | ADNI | 202 | 3 | N / A | N / A | Lien |
| IRMf | GROSSES | 65 | 4 | N / A | N / A | Lien |
| MEG | BCI-C IV, ensemble de données III | 2 | 4 | 400 | 10 | Lien |
Afin de permettre aux lecteurs d'avoir un accès rapide à l'ensemble de données et de pouvoir jouer avec, nous fournissons l'ensemble de données bien traité et prêt à l'emploi de la base de données de mouvements/images moteurs EEG (EEGMMIDB). Cet ensemble de données contient 109 sujets tandis que les signaux EEG sont enregistrés sur 64 canaux avec un taux d'échantillonnage de 160 Hz. Après notre nettoyage et notre tri, chaque fichier npy représente un sujet : la forme des données de chaque fichier npy est [N, 65], les 64 premières colonnes correspondent à 64 fonctionnalités de canal, la dernière colonne désigne l'étiquette de classe. Le N varie selon les sujets, mais N doit être soit 259 520, soit 255 680. Il s'agit de la différence inhérente à l'ensemble de données d'origine.
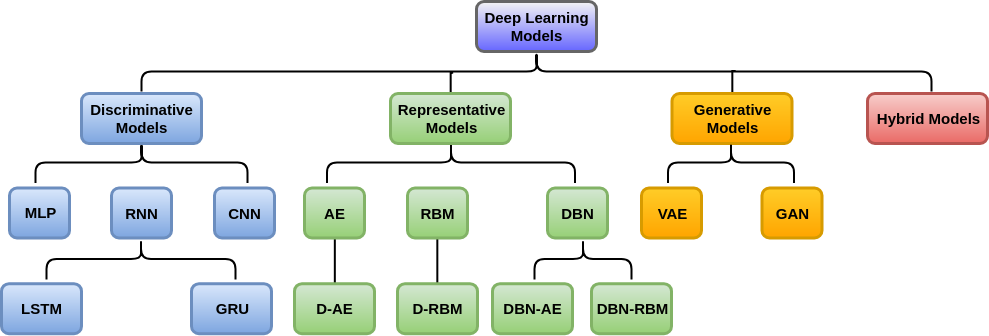
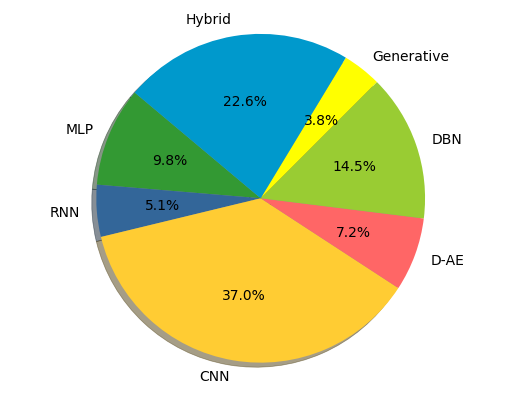
Dans nos fichiers de didacticiel, vous découvrirez le pipeline et le flux de travail du système BCI, y compris l'acquisition de données, le prétraitement, l'extraction de fonctionnalités (facultatif), la classification et l'évaluation. Nous présentons les références nécessaires et les codes exploitables des modèles d'apprentissage profond les plus typiques (GRU, LSTM, CNN, GNN) tout en tirant parti des dépendances temporelles, spatiales et topographiques. Nous fournissons également des codes python très pratiques. Par exemple, pour vérifier les performances de classification EEG de CNN, exécutez le code suivant :
python 4-2_CNN.py
Pour les débutants PyTorch, nous recommandons vivement les didacticiels PyTorch de Morvan Zhou.
Pour les algorithmes et applications présentés dans le livre, nous fournissons les codes d'implémentation nécessaires (version TensorFlow) :
Si vous trouvez que nos recherches sont utiles pour votre recherche, pensez à citer notre enquête ou notre livre :
@article{zhang2020survey,
title={A survey on deep learning-based non-invasive brain signals: recent advances and new frontiers},
author={Zhang, Xiang and Yao, Lina and Wang, Xianzhi and Monaghan, Jessica JM and Mcalpine, David and Zhang, Yu},
journal={Journal of Neural Engineering},
year={2020},
publisher={IOP Publishing}
}
@book{zhang2021deep,
title={Deep Learning for EEG-based Brain-Computer Interface: Representations, Algorithms and Applications},
author={Zhang, Xiang and Yao, Lina},
year={2021},
publisher={World Scientific Publishing}
}
Les codes du didacticiel sont testés pour fonctionner sous Python 3.7.
Des versions récentes de Pytorch, torch-geometric, numpy et scipy sont requises. Tous les packages de base requis peuvent être installés à l'aide de la commande suivante : ''' pip install -r Requirements.txt ''' Remarque : Pour toch-geometric et les dépendances associées (par exemple, cluster, scatter, sparse), la version supérieure peut Fonctionne mais n'a pas encore été testé.
Veuillez envoyer toutes vos questions sur le code et/ou l'algorithme à [email protected].
Ce didacticiel est sous licence MIT.