Terakhir diperbarui: 30/9/2024
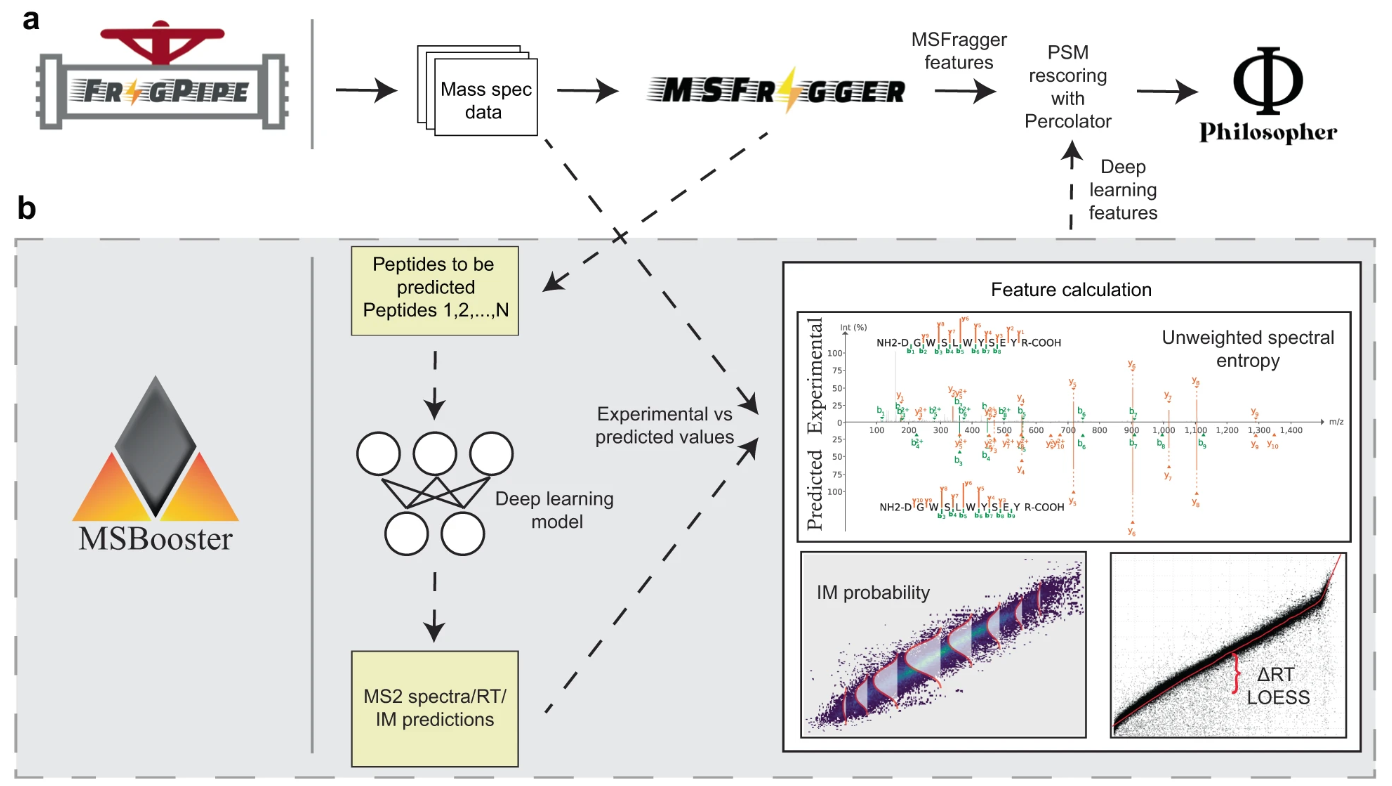
MSBooster adalah alat untuk menggabungkan prediksi perpustakaan spektral ke dalam pencocokan peptida-spektrum (PSM) yang menghitung ulang data proteomik kromatografi cair tandem cair tandem dari bawah ke atas. Ini secara kasar dipecah menjadi 4 langkah:
Ekstraksi peptida dari PSM dalam hasil pencarian, dan pemformatan untuk file input prediktor mesin/pembelajaran mendalam (ML/DL)
Memanggil model prediksi dan menyimpan hasilnya
Perhitungan fitur
Penambahan fitur baru pada file hasil pencarian
MSBooster kompatibel dengan banyak jenis pencarian basis data, termasuk imunopeptidomik HLA, DDA dan DIA, dan proteomik sel tunggal. Ini dimasukkan ke dalam FragPipe dan disertakan dalam banyak alur kerjanya. MSBooster dikembangkan dengan mempertimbangkan alat FragPipe lainnya, seperti FragPipe-PDV.
MSBooster dilengkapi untuk menangani berbagai format dan model file input:
| Keluaran spektrometer massa |
|---|
| .mzML |
| .mgf |
| berkas PSM |
|---|
| .pin |
| .pepXML (sedang berlangsung) |
| Model prediksi |
|---|
| DIA-NN |
| Model Koina |
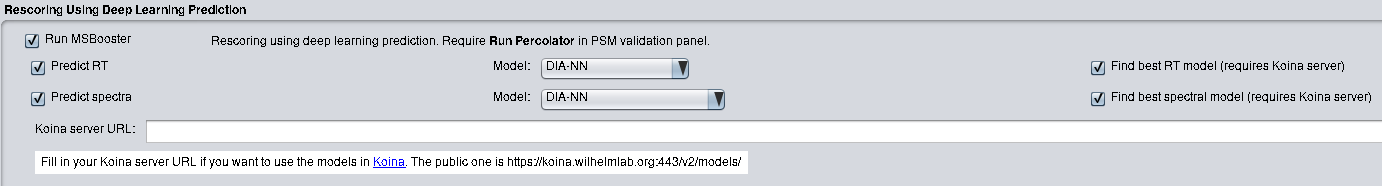
MSBooster dapat dijalankan di sistem Windows dan Linux. Jika menggunakan FragPipe, tidak diperlukan langkah instalasi lain selain menginstal FragPipe. MSBooster terletak di tab "Validasi". Pilih untuk mengaktifkan fitur waktu retensi dengan "Predict RT" dan fitur spektral MS/MS dengan "Predict spectra". Silakan merujuk ke dokumentasi FragPipe untuk mengetahui cara menjalankan analisis.
Jika menggunakan MSBooster mandiri untuk dijalankan di baris perintah, silakan unduh file jar terbaru dari Rilis. MSBooster juga memerlukan DIA-NN untuk prediksi MS/MS dan RT. Silakan instal DIA-NN dan catat jalur ke DIA-NN yang dapat dieksekusi (mis. DiaNN.exe untuk Windows, diann-1.8.1.8 untuk Linux).
Anda dapat menjalankan MSBooster menggunakan perintah yang mirip dengan berikut ini:
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
Parameter minimum yang harus dilewati adalah:
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
Meskipun Anda dapat meneruskan parameter ini satu per satu, akan lebih mudah untuk menempatkan satu parameter di setiap baris file paramsList. Silakan merujuk ke msbooster_params.txt untuk template.
Parameter di bawah ini untuk penggunaan umum. Parameter khusus Koina ada di dokumentasi Koina
paramsList (String) : lokasi ke file teks yang berisi parameter untuk proses ini
fragger (String) : jalur file fragger.params dari proses MSFragger. MSBooster akan membaca beberapa parameter dan menyesuaikan parameter internal berdasarkan parameter tersebut, seperti toleransi kesalahan massa fragmen dan offset massa
outputDirectory (String) : tempat mengeluarkan file baru
editedPin (String) : MSBooster akan memberi nama file baru berdasarkan yang disediakan. Misalnya, A.pin akan memiliki padanannya yang disebut A_edited.pin. Untuk mengubah dari default "diedit", berikan string baru di sini
renamePin (int) : apakah akan membuat file pin baru atau menulis ulang yang lama. Defaultnya di sini adalah 1, yang tidak akan ditimpa. Menyetelnya ke 0 akan menimpa file pin lama
deletePreds (boolean) : apakah akan menghapus file yang menyimpan prediksi model setelah menyelesaikan proses yang berhasil. Secara default, disetel ke salah. Setel ke true jika Anda ingin menghapusnya
loadingPercent (int) : seberapa sering melaporkan kemajuan tugas menggunakan pelapor kemajuan. Secara default, disetel ke 10, artinya pembaruan akan dicetak setiap 10%.
numThreads (int) : jumlah thread yang akan digunakan. Secara default disetel ke 0, yang menggunakan semua thread yang tersedia dikurangi 1
splitPredInputFile (int) : hanya digunakan ketika prediksi DIA-NN gagal karena kesalahan kehabisan memori (137). Secara default, disetel ke 1, namun Anda dapat meningkatkannya untuk menentukan berapa banyak file kecil yang harus dipecah menjadi file masukan DIA-NN. Setiap file kemudian akan diprediksi secara berurutan, meringankan beban memori
plotExtension (String) : format file apa yang seharusnya ada dalam plot.png secara default, dan pdf juga diperbolehkan
features (String) : daftar fitur yang akan dihitung. Peka huruf besar-kecil, dipisahkan komunikasi tanpa spasi di antaranya. Standarnya adalah "predRTrealUnits,SpectralEntropy tidak tertimbang,deltaRTLOESS"
spectraPredFile (String) : jika Anda menggunakan kembali prediksi spektral lama (misalnya dari DIA-NN atau Koina), Anda dapat menentukan lokasi file di sini
RTPredFile (String) : sama seperti spectraPredFile, tetapi untuk prediksi RT
IMPredFile (String) : sama seperti spectraPredFile, tetapi untuk prediksi IM
spectraModel (String) : model prediksi spektral mana yang akan digunakan
rtModel (String) : sama seperti spectraModel, tetapi untuk RT
imModel (String) : sama seperti spectraModel, tetapi untuk IM
useSpectra (boolean) : apakah akan menggunakan fitur berbasis prediksi spektral. Disetel ke benar secara default
useRT (boolean) : apakah akan menggunakan fitur berbasis prediksi RT. Disetel ke benar secara default
useIM (boolean) : apakah akan menggunakan fitur berbasis prediksi IM. Diatur ke salah secara default
ppmTolerance (float) : kesalahan fragmen toleransi ppm (default 20ppm)
matchWithDaltons (boolean) : apakah cocok dengan fragmen yang diprediksi dan diamati di Daltons (default salah)
DaTolerance (float) : berapa dalton di sekitar puncak yang diprediksi untuk mencari puncak eksperimental (default 0,05)
useTopFragments (boolean) : apakah akan memfilter prediksi spektral ke N puncak intensitas tertinggi (default benar)
topFragments (int) : hingga berapa banyak prediksi fragmen yang harus digunakan untuk penghitungan fitur (default 20). Hanya diterapkan jika useTopFragments benar
removeRankPeaks (boolean) : Ditetapkan ke true secara default, yang memfilter fragmen dari spektrum eksperimental setelah cocok. Jika salah, fragmen eksperimen dapat dicocokkan dengan beberapa PSM dari pemindaian yang sama
useBasePeak (boolean) : apakah batas bawah harus diterapkan pada prediksi MS2 untuk hanya menggunakan fragmen dengan intensitas lebih tinggi (default benar)
percentBasePeak (float) : persen di mana fragmen dengan intensitas beberapa persen dari intensitas puncak dasar dimasukkan dalam perhitungan kesamaan. Hanya diterapkan jika useBasePeak benar (default 1)
loessEscoreCutoff (float) : batas nilai ekspektasi yang digunakan pada tahap pertama pengumpulan PSM untuk kalibrasi RT/IM. Standarnya adalah 10^-3,5, atau sekitar 0,000316
rtLoessRegressionSize (int) : jumlah maksimum PSM yang digunakan untuk kalibrasi RT LOESS (default 5000)
imLoessRegressionSize (int) : sama seperti rtLoessRegressionSize tetapi untuk IM (default 1000)
minLoessRegressionSize (int) : jumlah minimum PSM yang diperlukan untuk mencoba kalibrasi LOESS RT/IM (default 100). Jika jumlah PSM yang tersedia kurang dari jumlah ini, regresi linier digunakan sebagai gantinya
minLinearRegressionSize (int) : jumlah minimum PSM yang diperlukan untuk mencoba kalibrasi RT/IM regresi linier (default 10). Jika PSM yang tersedia kurang dari jumlah ini, kalibrasi tidak dilakukan
loessBandwidth (String) : daftar bandwidth yang akan dicoba untuk kalibrasi RT/IM LOESS (default 0.01,0.05,0.1,0.2). Ini harus dipisahkan dengan koma tanpa spasi di antaranya
regressionSplits (int) : jumlah validasi silang yang digunakan untuk kalibrasi RT/IM LOESS (default 5)
massesForLoessCalibration (String) : massa untuk pergeseran massa yang harus sesuai dengan kurva kalibrasinya sendiri. Daftar dipisahkan dengan koma tanpa spasi di antaranya. Massa harus ditulis dengan jumlah digit yang sama seperti pada file PIN
loessScatterOpacity (float) : opasitas plot sebar dalam angka kalibrasi LOESS, dari 0 hingga 1 (default 0,35)
File .pin dengan fitur baru. Secara default, file pin baru akan diproduksi dengan akhiran "_edited.pin". Fitur default yang digunakan adalah "unweighted_spectral_entropy", "delta_RT_loess", dan "pred_RT_real_units". Jika fitur mobilitas ion diaktifkan, "delta_IM_loess" dan "ion_mobility" juga akan disertakan
spectraRT.tsv dan spectraRT_full.tsv: file masukan untuk model prediksi DIA-NN
spectraRT.predicted.bin: file biner dengan prediksi dari DIA-NN yang akan digunakan oleh MSBooster untuk perhitungan fitur. Jika menggunakan FragPipe-PDV, file ini digunakan untuk menghasilkan plot cermin spektrum eksperimental dan prediksi
MSBooster menghasilkan beberapa grafik yang dapat digunakan untuk memeriksa lebih lanjut bagaimana data Anda dibandingkan dengan prediksi model.
Folder MSBooster_plots:
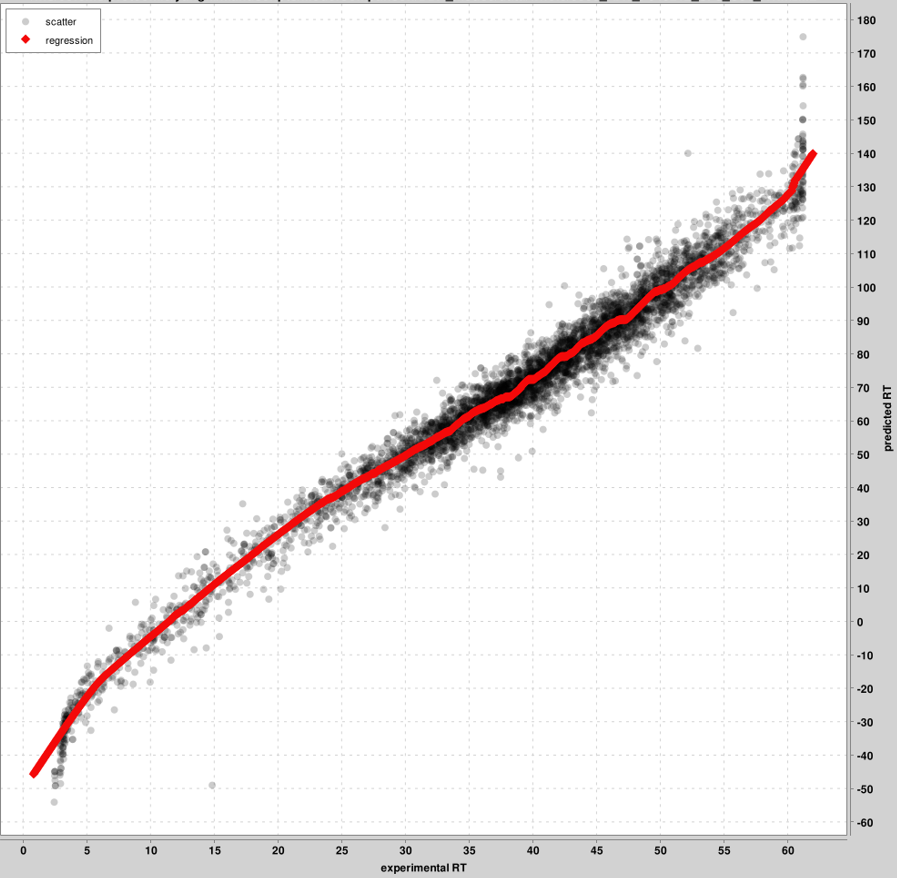
RT_calibration_curves: hingga 5000 PSM teratas akan digunakan untuk kalibrasi antara skala RT eksperimental dan prediksi. PSM teratas ini disajikan dalam grafik, tidak semua PSM. Satu grafik akan dihasilkan per file pin
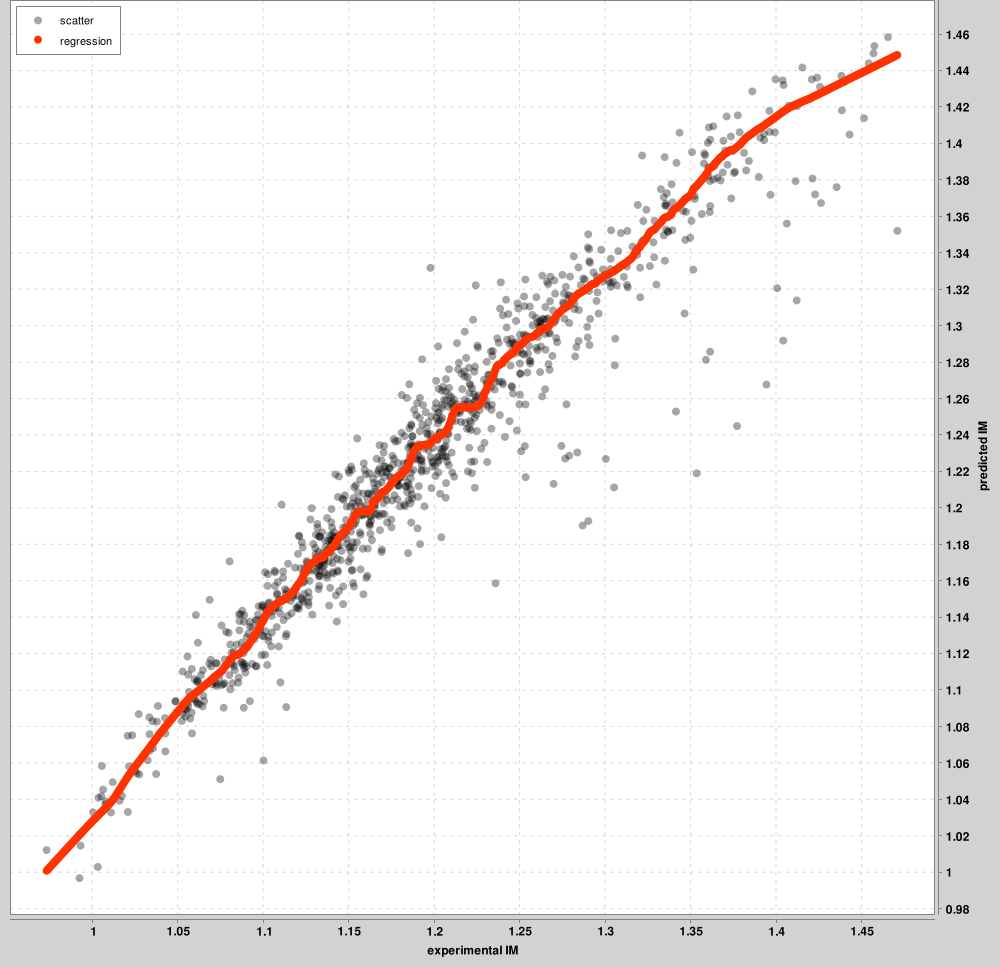
IM_calibration_curves: hingga 1000 PSM teratas akan digunakan untuk kalibrasi antara skala IM eksperimental dan prediksi. PSM teratas ini disajikan dalam grafik, tidak semua PSM. Kurva terpisah akan dipelajari untuk setiap status muatan. Gambar di bawah adalah contoh prekursor muatan 2
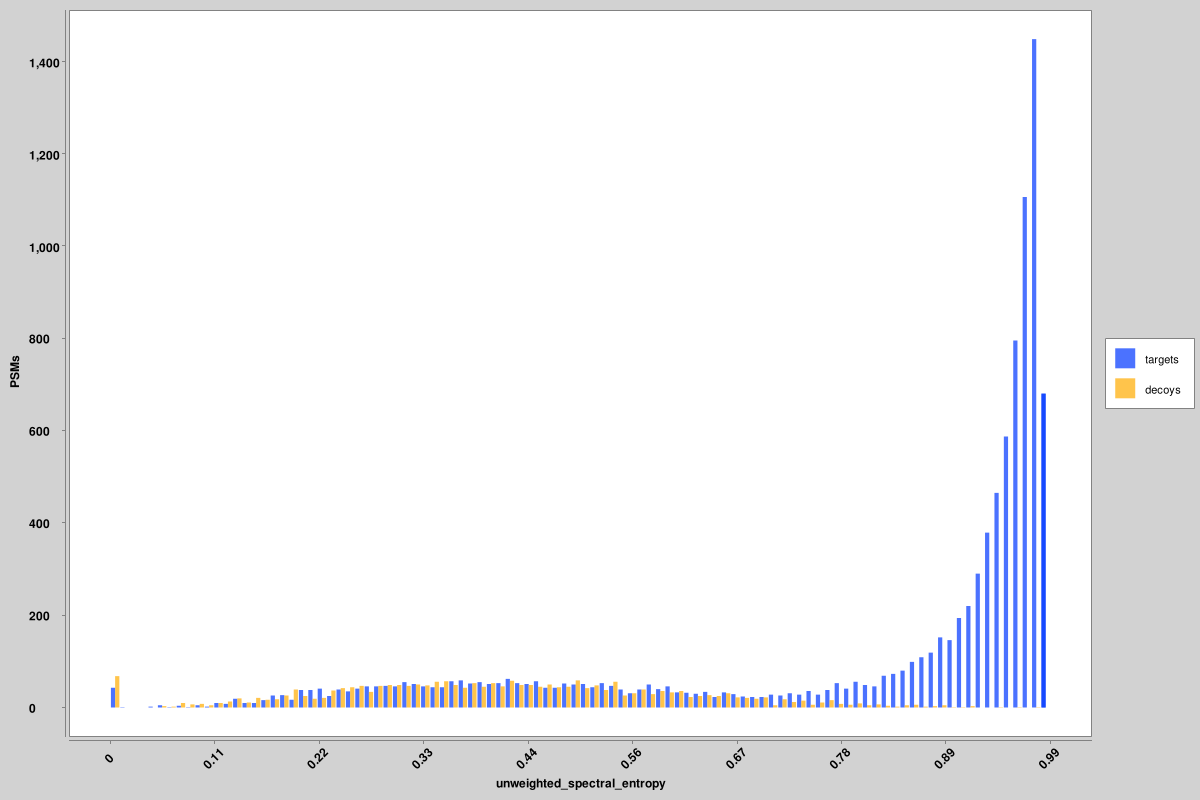
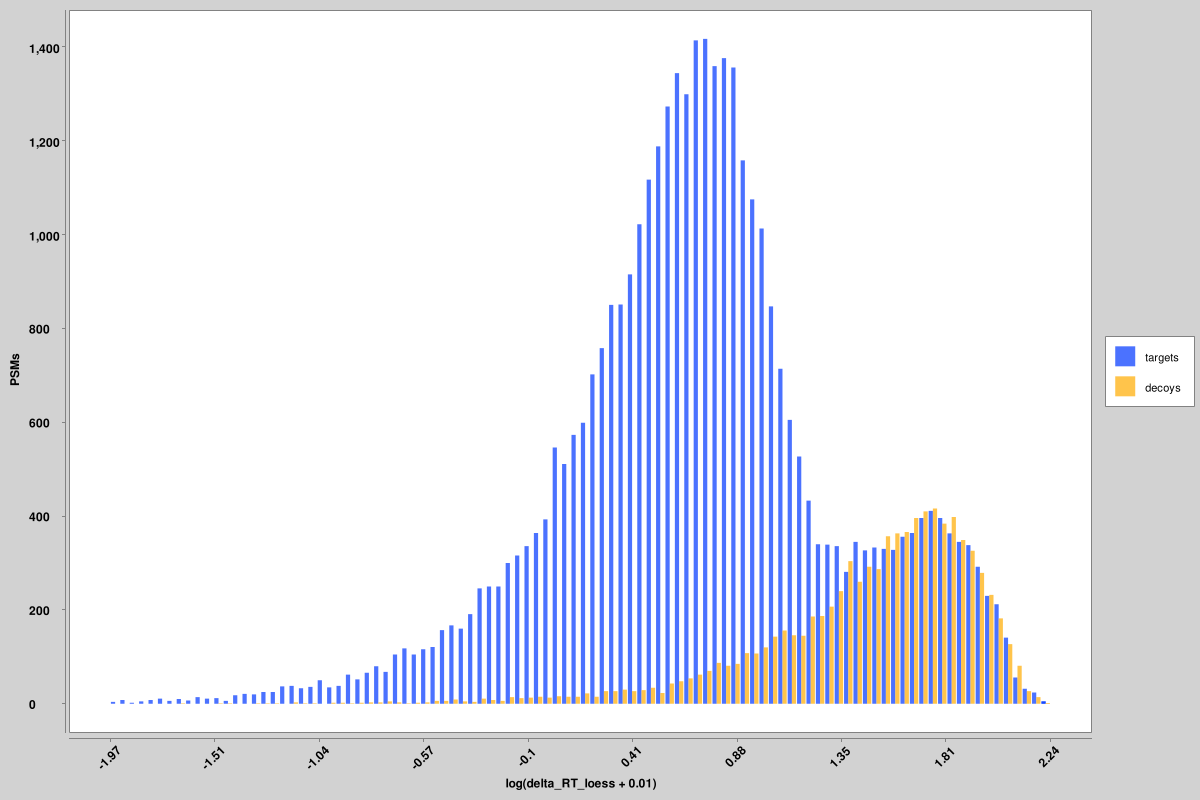
score_histograms: histogram yang dihamparkan dari semua PSM target dan umpan untuk setiap file pin. Beberapa fitur diplot di sini pada skala log untuk visualisasi yang lebih baik dari distribusi bimodal positif benar dan salah, tetapi nilai asli adalah apa yang digunakan dalam file pin, bukan versi skala log. Di sini ditampilkan histogram untuk entropi spektral tak berbobot dan skor delta RT, namun histogram serupa dihasilkan untuk semua fitur
Gunakan model prediksi peptida dari Koina untuk pembuatan fitur MSBooster: https://fragpipe.nesvilab.org/docs/tutorial_koina.html
Membaca prediksi dari model apa pun melalui file MGF
Dokumentasi tentang semua fitur yang diizinkan dan cara melakukan QC dengan output grafis
Harap kutip yang berikut ini saat menggunakan MSBooster: https://www.nature.com/articles/s41467-023-40129-9