Rangkaian alur kerja molekuler dan komputasi untuk Spatial Total RNA-Sequencing (STRS)
Repositori ini memiliki semua protokol, pipeline, dan skrip yang Anda perlukan untuk menjalankan dan menganalisis STRS di hampir semua sampel!
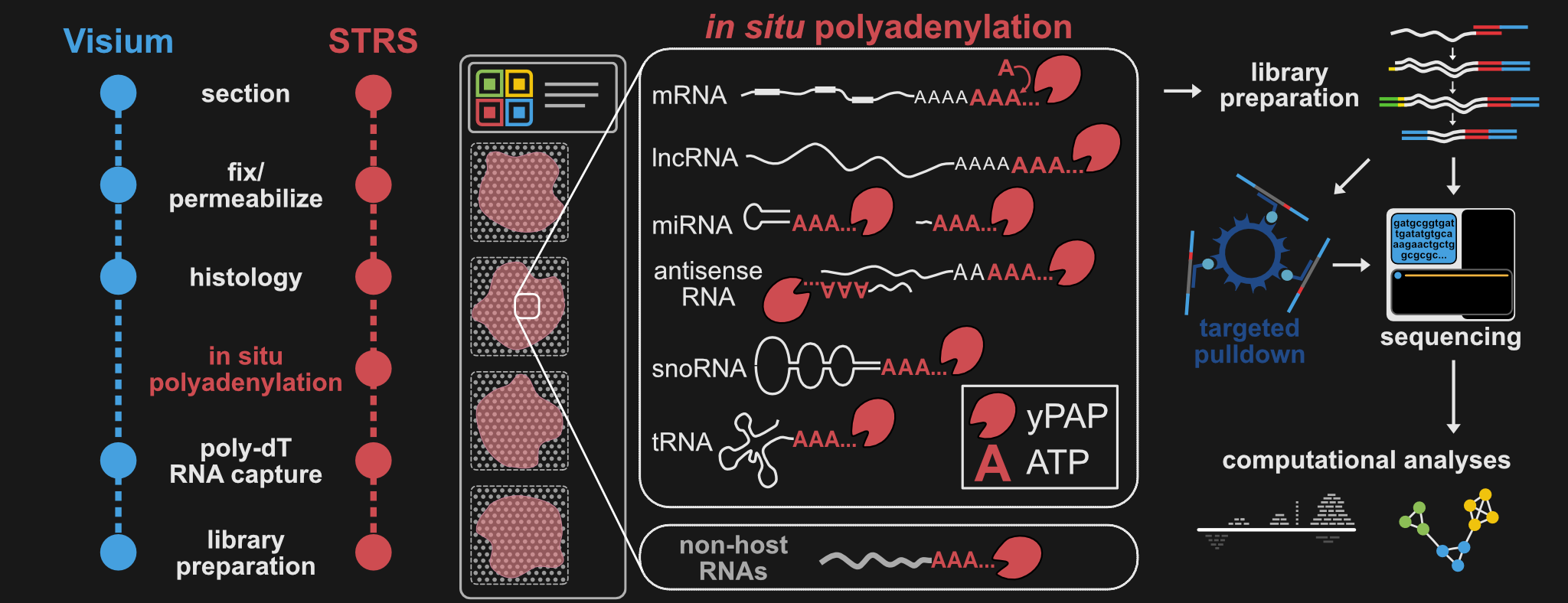
txg_snake , pipeline yang sedang dalam proses untuk 10x perpustakaan, terutama kumpulan data STRS!McKellar dkk, Bioteknologi Alam , 2022
tautan pracetak bioRxiv
manuscripts : file .pdf dari pracetak dan manuskrip selanjutnya (semoga!)
protocols : Dokumen Microsoft word dan .pdf dari protokol yang digunakan di sini
pipelines : Alur kerja Snakemake yang digunakan dalam penelitian kami. Ini termasuk jalur penyelarasan untuk data STRS (kallisto, STARsolo, & miRge3.0), data RNAseq kecil (STAR & miRge3.0), SmartSeqTotal (kallisto), dan VASAdrop (kallisto)
scripts : Semua kode lain yang kami gunakan! Sebagian besar berisi skrip R dan fungsi utilitas yang digunakan dalam analisis spasial kami.
references : info & skrip pada genom referensi & anotasi yang digunakan dalam penelitian ini
resources : berbagai macam metadata, daftar gen, dan informasi lain yang kami gunakan untuk menganalisis data kami , lihat file README di setiap subdirektori untuk lebih jelasnya
Kumpulan data STRS: GSE200481
Data RNAseq kecil SkM/hati: GSE200480
data RNAseq total inti tunggal (garis sel C2C12): GSE209780
Data RNA Atlas kecil (Isakova et al, PNAS , 2020): GSE119661
Data SmartSeqTotal (Isakova dkk, PNAS , 2021): GSE151334
Data VASAdrop (Salmen dkk, Bioteknologi Alam , 2022): GSE176588
Iwijn De Vlaminck ([email protected])
Benjamin D. Cosgrove ([email protected])
David W. McKellar ([email protected])