Perlu diketahui bahwa repositori ini hanya untuk keperluan arsip. Fungsi ANCOM yang berdiri sendiri di repositori ini tidak lagi dipertahankan. Pengguna sangat disarankan untuk menggunakan fungsi ancom atau ancombc dalam paket ANCOMBC R kami. Untuk laporan bug, silakan buka repositori ANCOMBC.
Kode saat ini mengimplementasikan ANCOM dalam kumpulan data cross-sectional dan longitudinal sekaligus memungkinkan penggunaan kovariat. Pustaka berikut perlu disertakan agar kode R dapat dijalankan:
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )Kami mengadopsi metodologi ANCOM-II sebagai langkah pra-pemrosesan untuk menangani berbagai jenis angka nol sebelum melakukan analisis kelimpahan diferensial.
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : Bingkai data atau matriks yang mewakili tabel OTU/SV yang diamati dengan taksa dalam baris ( rownames ) dan sampel dalam kolom ( colnames ). Perhatikan bahwa ini adalah tabel kelimpahan absolut , jangan diubah menjadi tabel kelimpahan relatif (yang total kolomnya sama dengan 1).meta_data : Kerangka data atau matriks semua variabel dan kovariat yang diminati.sample_var : Karakter. Nama kolom yang menyimpan ID sampel.group_var : Karakter. Nama indikator grup. group_var diperlukan untuk mendeteksi angka nol dan outlier struktural. Untuk definisi berbagai angka nol (nol struktural, nol outlier, dan nol pengambilan sampel), silakan merujuk ke ANCOM-II.out_cut Pecahan numerik antara 0 dan 1. Untuk setiap takson, pengamatan dengan proporsi distribusi campuran kurang dari out_cut akan terdeteksi sebagai outlier nol; sedangkan observasi dengan proporsi distribusi campuran lebih besar dari 1 - out_cut akan terdeteksi sebagai nilai outlier.zero_cut : Pecahan numerik antara 0 dan 1. Taksa dengan proporsi nol lebih besar dari zero_cut tidak disertakan dalam analisis.lib_cut : Numerik. Sampel dengan ukuran perpustakaan kurang dari lib_cut tidak disertakan dalam analisis.neg_lb : Logis. BENAR menunjukkan bahwa suatu takson akan diklasifikasikan sebagai struktur nol pada kelompok eksperimen yang bersangkutan menggunakan batas bawahnya yang asimtotik. Lebih khusus lagi, neg_lb = TRUE menunjukkan Anda menggunakan kedua kriteria yang dinyatakan di bagian 3.2 ANCOM-II untuk mendeteksi angka nol struktural; Jika tidak, neg_lb = FALSE hanya akan menggunakan persamaan 1 di bagian 3.2 ANCOM-II untuk mendeklarasikan nol struktural. feature_table : Bingkai data tabel OTU yang telah diproses sebelumnya.meta_data : Bingkai data metadata yang telah diproses sebelumnya.structure_zeros : Sebuah matriks terdiri dari 0 dan 1 dengan 1 menunjukkan takson diidentifikasi sebagai nol struktural dalam kelompok yang sesuai.ANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : Bingkai data yang mewakili tabel OTU/SV dengan taksa dalam baris ( rownames ) dan sampel dalam kolom ( colnames ). Ini bisa berupa nilai keluaran dari feature_table_pre_process . Perhatikan bahwa ini adalah tabel kelimpahan absolut , jangan diubah menjadi tabel kelimpahan relatif (yang total kolomnya sama dengan 1).meta_data : Kerangka data variabel. Dapat berupa nilai keluaran dari feature_table_pre_process .struc_zero : Sebuah matriks terdiri dari 0 dan 1 dengan 1 menunjukkan takson diidentifikasi sebagai nol struktural dalam kelompok yang sesuai. Dapat berupa nilai keluaran dari feature_table_pre_process .main_var : Karakter. Nama variabel utama yang diminati. ANCOM v2.1 saat ini mendukung main_var kategorikal.p_adjust_method : Karakter. Menentukan metode untuk menyesuaikan nilai p untuk beberapa perbandingan. Standarnya adalah “BH” (prosedur Benjamini-Hochberg).alpha : Tingkat signifikansi. Standarnya adalah 0,05.adj_formula : String karakter yang mewakili rumus penyesuaian (lihat contoh).rand_formula : String karakter yang mewakili rumus untuk efek acak di lme . Untuk detailnya, lihat ?lme .lme_control : Daftar yang menentukan nilai kontrol untuk lme fit. Untuk detailnya, lihat ?lmeControl . 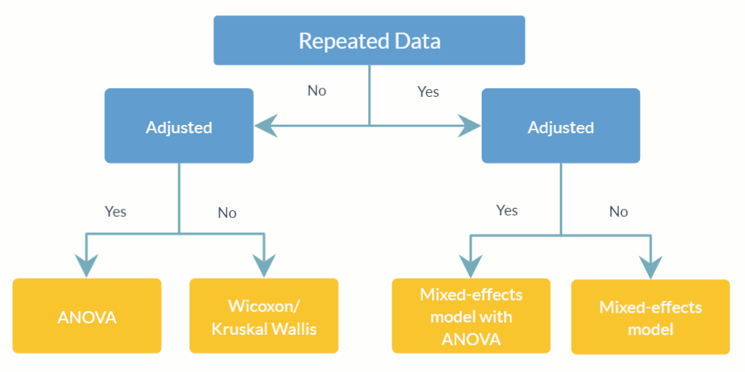
out : Kerangka data dengan statistik W untuk setiap taksa dan kolom berikutnya yang merupakan indikator logis apakah suatu OTU atau takson berlimpah secara berbeda dalam serangkaian potongan (0,9, 0,8, 0,7, dan 0,6). detected_0.7 umumnya digunakan. Namun, Anda dapat memilih detected_0.8 atau detected_0.9 jika Anda ingin lebih konservatif pada hasil Anda (FDR lebih kecil), atau menggunakan detected_0.6 jika Anda ingin menjelajahi lebih banyak penemuan (kekuatan lebih besar)
fig : Objek ggplot dari plot gunung berapi.
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Di sini kami ingin mengetahui apakah ada angka nol struktural di berbagai tingkat delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var . Di sini kami ingin mengetahui apakah ada angka nol struktural di berbagai tingkat delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Di sini kami ingin mengetahui apakah ada angka nol struktural di berbagai tingkat delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )