Pemuatan data batch yang cepat dari file BigWig yang berisi data trek epigentik dan urutan terkait yang didukung oleh GPU untuk aplikasi pembelajaran mendalam.
Bigwig-loader terutama bergantung pada perpustakaan rapidsai kvikio dan cupy, keduanya paling baik diinstal menggunakan conda/mamba. Bigwig-loader sekarang juga dapat diinstal menggunakan conda/mamba. Untuk membuat lingkungan baru dengan bigwig-loader terinstal:
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loaderAtau tambahkan ini ke file environment.yml Anda:
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loaderdan perbarui:
mamba env update -f environment.ymlBigwig-loader juga dapat diinstal menggunakan pip di lingkungan yang sudah menginstal perpustakaan rapidsai kvikio dan cupy:
pip install bigwig-loaderKami menggabungkan BigWigDataset dalam kumpulan data PyTorch yang dapat diubah yang dapat Anda gunakan langsung:
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step () Objek Dataset kerangka agnostik dapat diimpor dari bigwig_loader.dataset . Objek kumpulan data ini mengembalikan tensor cupy. Tensor cupy mematuhi antarmuka array cuda dan dapat ditransformasikan tanpa salinan menjadi tensor JAX atau tensorflow.
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)Lihat direktori contoh untuk contoh lainnya.
Pustaka ini dimaksudkan untuk memuat kumpulan data dengan dimensi yang sama, yang memungkinkan beberapa asumsi yang dapat mempercepat proses pemuatan. Seperti yang dapat dilihat dari plot di bawah, saat memuat sejumlah kecil data, pyBigWig sangat cepat, tetapi tidak memanfaatkan sifat pemuatan data yang bersifat batch untuk pembelajaran mesin.
Pada benchmark di bawah ini kami juga membuat dataloader PyTorch (dengan set_start_method('spawn')) menggunakan pyBigWig untuk membandingkan dengan skenario realistis di mana beberapa CPU akan digunakan per GPU. Kami melihat bahwa throughput pemuat data CPU tidak naik secara linier seiring dengan jumlah CPU, dan oleh karena itu menjadi sulit untuk mendapatkan throughput yang diperlukan untuk menjaga GPU, melatih jaringan saraf, tetap jenuh selama langkah-langkah pembelajaran.
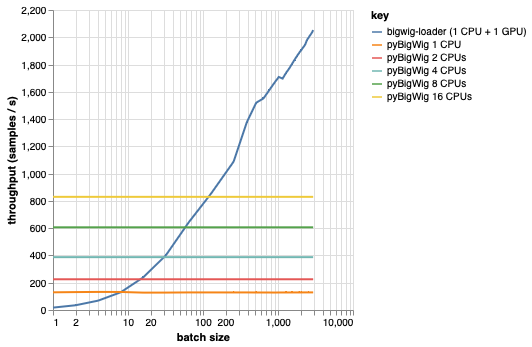
Ini adalah masalah yang dipecahkan oleh petinggi-loader. Ini adalah contoh cara menggunakan bigwig-loader:
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.yml Di lingkungan ini Anda seharusnya dapat menjalankan pytest -v dan melihat pengujiannya berhasil. CATATAN: Anda memerlukan GPU untuk menggunakan bigwig-loader!
Bagian ini memandu Anda melalui langkah-langkah yang diperlukan untuk menambahkan fungsionalitas baru. Jika ada yang kurang jelas, silakan buka isu.
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install untuk memasang hook pre-commitTes ada di direktori tes. Salah satu tes yang paling penting adalah test_against_pybigwig yang memastikan bahwa jika ada kesalahan di pyBigWIg, kesalahan itu juga ada di bigwig-loader.
pytest -vv .Saat pelari github dengan GPU tersedia, kami juga ingin menjalankan pengujian ini di CI. Namun untuk saat ini, Anda dapat menjalankannya secara lokal.
Jika Anda menggunakan perpustakaan ini, pertimbangkan untuk mengutip:
Retel, Joren Sebastian, Andreas Poehlmann, Josh Chiou, Andreas Steffen, dan Djork-Arné Clevert. “Pemuat Data Pembelajaran Mesin Cepat untuk Trek Epigenetik dari File BigWig.” Bioinformatika 40, no. 1 (1 Januari 2024): btad767. https://doi.org/10.1093/bioinformatics/btad767.
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}