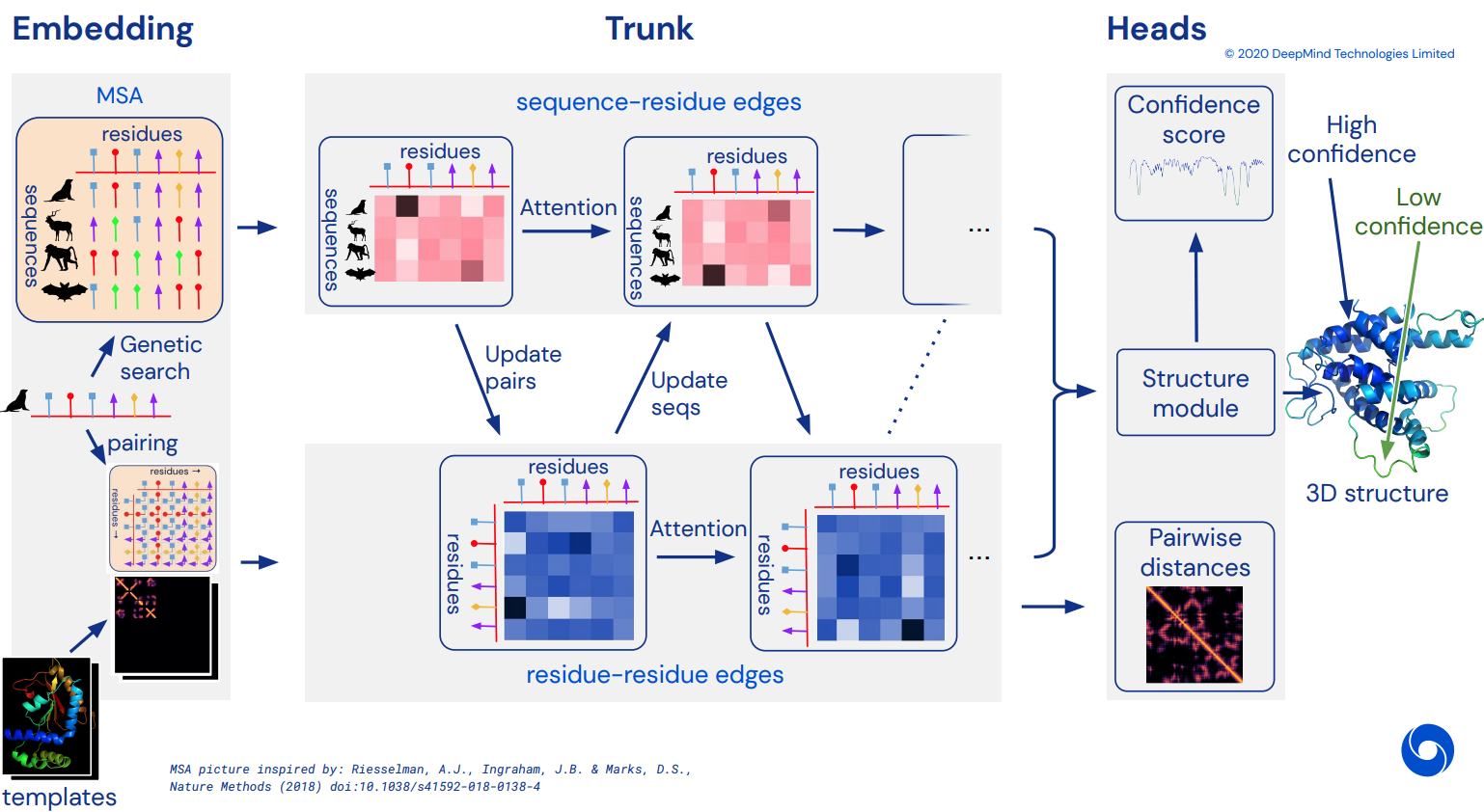
Untuk akhirnya menjadi implementasi Pytorch yang berfungsi tidak resmi dari Alphafold2, jaringan perhatian menakjubkan yang memecahkan CASP14. Akan diterapkan secara bertahap seiring dengan dirilisnya lebih banyak detail arsitektur.
Setelah ini direplikasi, saya bermaksud untuk melipat semua rangkaian asam amino yang tersedia di luar sana ke dalam silico dan merilisnya sebagai torrent akademis, untuk ilmu pengetahuan lebih lanjut. Jika Anda tertarik dengan upaya replikasi, silakan mampir #alphafold di saluran Discord ini
Pembaruan: Deepmind telah membuka kode resmi di Jax, beserta bobotnya! Repositori ini sekarang akan diarahkan ke terjemahan pytorch langsung dengan beberapa perbaikan pada pengkodean posisi
Video ArxivInsights
$ pip install alphafold2-pytorchlhatsk telah melaporkan pelatihan bagasi yang dimodifikasi dari repositori ini, menggunakan pengaturan yang sama seperti trRosetta, dengan hasil yang kompetitif
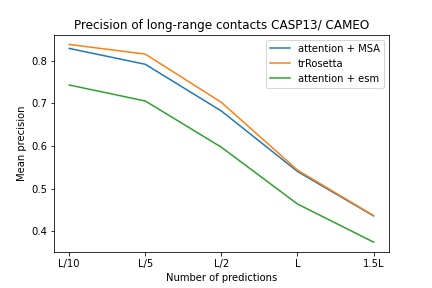
blue used the the trRosetta input (MSA -> potts -> axial attention), green used the ESM embedding (only sequence) -> tiling -> axial attention - lhatsk
Memprediksi distogram, seperti Alphafold-1, tetapi dengan perhatian
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
reversible = False # set this to True for fully reversible self / cross attention for the trunk
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda () # AA length of 128
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda () # MSA doesn't have to be the same length as primary sequence
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) Anda juga dapat mengaktifkan prediksi sudut, dengan meneruskan predict_angles = True pada init. Contoh di bawah ini akan setara dengan trRosetta tetapi dengan perhatian mandiri/silang.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_angles = True # set this to True
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram , theta , phi , omega = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
)
# distogram - (1, 128, 128, 37),
# theta - (1, 128, 128, 25),
# phi - (1, 128, 128, 13),
# omega - (1, 128, 128, 25) Makalah Fabian baru-baru ini menyarankan pemberian kembali koordinat ke SE3 Transformer secara berulang, dengan pembagian bobot, mungkin berhasil. Saya telah memutuskan untuk mengeksekusi berdasarkan ide ini, meskipun cara kerjanya sebenarnya masih belum jelas.
Anda juga dapat menggunakan E(n)-Transformer atau EGNN untuk penyempurnaan struktural.
Pembaruan: Lab Baker telah menunjukkan bahwa arsitektur end-to-end dari sequence dan embeddings MSA ke SE3 Transformers dapat melakukan trRosetta terbaik dan menutup kesenjangan ke Alphafold2. Kita akan menggunakan Graph Transformer, yang bekerja pada penyematan trunk, untuk menghasilkan kumpulan koordinat awal untuk dikirim ke jaringan ekuivalen. (Hal ini selanjutnya dikuatkan oleh Costa dkk dalam pekerjaan mereka yang mencari koordinat 3d dari penyematan MSA Transformer dalam makalah yang mendahului laboratorium Baker)
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
structure_module_type = 'se3' , # use SE3 Transformer - if set to False, will use E(n)-Transformer, Victor and Max Welling's new paper
structure_module_dim = 4 , # se3 transformer dimension
structure_module_depth = 1 , # depth
structure_module_heads = 1 , # heads
structure_module_dim_head = 16 , # dimension of heads
structure_module_refinement_iters = 2 , # number of equivariant coordinate refinement iterations
structure_num_global_nodes = 1 # number of global nodes for the structure module, only works with SE3 transformer
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 3, 3) <-- 3 atoms per residue Asumsi yang mendasarinya adalah bahwa trunk bekerja pada tingkat residu, dan kemudian membentuk tingkat atom untuk modul struktur, apakah itu SE3 Transformers, E(n)-Transformer, atau EGNN yang melakukan penyempurnaan. Pustaka ini defaultnya adalah 3 atom tulang punggung (C, Ca, N), tetapi Anda dapat mengonfigurasinya untuk menyertakan atom lain yang Anda suka, termasuk Cb dan rantai samping.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
atoms = 'backbone-with-cbeta'
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 4, 3) <-- 4 atoms per residue (C, Ca, N, Cb) Pilihan yang valid untuk atoms meliputi:
backbone - 3 atom tulang punggung (C, Ca, N) [default]backbone-with-cbeta - 3 atom tulang punggung dan C betabackbone-with-oxygen - 3 atom tulang punggung dan oksigen dari karboksilbackbone-with-cbeta-and-oxygen - 3 atom tulang punggung dengan C beta dan oksigenall - tulang punggung dan semua atom lain dari rantai sampingAnda juga dapat memasukkan bentuk tensor (14,) yang menentukan atom mana yang ingin Anda sertakan
mantan.
atoms = torch . tensor ([ 1 , 1 , 1 , 1 , 1 , 1 , 0 , 1 , 0 , 0 , 0 , 0 , 0 , 1 ])Repositori ini menawarkan Anda penambahan jaringan yang mudah dengan penyematan terlatih dari Facebook AI. Ini berisi pembungkus untuk ESM terlatih, MSA Transformers, atau Protein Transformer.
Ada beberapa prasyarat. Anda perlu memastikan bahwa Anda telah menginstal perpustakaan apex Nvidia, karena transformator yang telah dilatih sebelumnya menggunakan beberapa operasi gabungan.
Atau Anda dapat mencoba menjalankan skrip di bawah ini
git clone https://github.com/NVIDIA/apex
cd apex
pip install -v --disable-pip-version-check --no-cache-dir --global-option= " --cpp_ext " --global-option= " --cuda_ext " ./ Selanjutnya, Anda hanya perlu mengimpor dan membungkus instance Alphafold2 Anda dengan ESMEmbedWrapper , MSAEmbedWrapper , atau ProtTranEmbedWrapper dan itu akan menangani penyematan urutan dan penyelarasan beberapa urutan untuk Anda (dan memproyeksikannya ke dimensi seperti yang ditentukan pada Anda model). Tidak ada yang perlu diubah kecuali menambahkan pembungkus.
import torch
from alphafold2_pytorch import Alphafold2
from alphafold2_pytorch . embeds import MSAEmbedWrapper
alphafold2 = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64
)
model = MSAEmbedWrapper (
alphafold2 = alphafold2
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) Secara default, meskipun wrapper menyuplai trunk dengan urutan dan penyematan MSA, keduanya akan dijumlahkan dengan penyematan token biasa. Jika Anda ingin melatih Alphafold2 tanpa penyematan token (hanya mengandalkan penyematan yang telah dilatih sebelumnya), Anda perlu disable_token_embed ke True pada init Alphafold2 .
alphafold2 = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
disable_token_embed = True
) Sebuah makalah yang ditulis oleh Jinbo Xu menyarankan bahwa seseorang tidak perlu membuang jarak, melainkan dapat memprediksi mean dan deviasi standar secara langsung. Anda dapat menggunakan ini dengan mengaktifkan satu predict_real_value_distances , dalam hal ini, prediksi jarak yang dikembalikan akan memiliki dimensi 2 untuk mean dan deviasi standar masing-masing.
Jika predict_coords juga diaktifkan, maka MDS akan menerima prediksi mean dan deviasi standar secara langsung tanpa harus menghitungnya dari bin distogram.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
predict_real_value_distances = True , # set this to True
structure_module_type = 'se3' ,
structure_module_dim = 4 ,
structure_module_depth = 1 ,
structure_module_heads = 1 ,
structure_module_dim_head = 16 ,
structure_module_refinement_iters = 2
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 3, 3) <-- 3 atoms per residue Anda dapat menambahkan blok konvolusional, baik untuk urutan utama maupun MSA, hanya dengan menetapkan satu argumen kata kunci tambahan use_conv = True
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True # set this to True
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37)Kernel konvolusional mengikuti jejak makalah ini, menggabungkan kernel 1d dan 2d dalam satu blok mirip resnet. Anda dapat sepenuhnya menyesuaikan kernel.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True , # set this to True
conv_seq_kernels = (( 9 , 1 ), ( 1 , 9 ), ( 3 , 3 )), # kernels for N x N primary sequence
conv_msa_kernels = (( 1 , 9 ), ( 3 , 3 )), # kernels for {num MSAs} x N MSAs
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) Anda juga dapat melakukan dilatasi siklus dengan satu argumen kata kunci tambahan. Pelebaran default adalah 1 untuk semua lapisan.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True , # set this to True
dilations = ( 1 , 3 , 5 ) # cycle between dilations of 1, 3, 5
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) Terakhir, alih-alih mengikuti pola konvolusi, perhatian diri, perhatian silang per pengulangan kedalaman, Anda dapat menyesuaikan urutan apa pun yang Anda inginkan dengan kata kunci custom_block_types
mantan. Jaringan tempat Anda melakukan sebagian besar konvolusi terlebih dahulu, diikuti dengan blok perhatian mandiri + perhatian silang
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
heads = 8 ,
dim_head = 64 ,
custom_block_types = (
* (( 'conv' ,) * 6 ),
* (( 'self' , 'cross' ) * 6 )
)
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) Anda dapat berlatih dengan Sparse Attention Microsoft Deepspeed, tetapi Anda harus menanggung proses instalasi. Ini adalah dua langkah.
Pertama, Anda perlu menginstal Deepspeed dengan Sparse Attention
$ sh install_deepspeed.sh Selanjutnya, Anda perlu menginstal paket pip triton
$ pip install tritonJika kedua cara di atas berhasil, kini Anda bisa berlatih dengan Sparse Attention!
Sayangnya, perhatian yang jarang tersebut hanya didukung untuk perhatian pada diri sendiri, dan bukan perhatian silang. Saya akan memberikan solusi berbeda untuk membuat perhatian silang menjadi efektif.
model = Alphafold2 (
dim = 256 ,
depth = 12 ,
heads = 8 ,
dim_head = 64 ,
max_seq_len = 2048 , # the maximum sequence length, this is required for sparse attention. the input cannot exceed what is set here
sparse_self_attn = ( True , False ) * 6 # interleave sparse and full attention for all 12 layers
). cuda ()Saya juga menambahkan salah satu varian perhatian linier terbaik, dengan harapan dapat mengurangi beban kehadiran silang. Saya pribadi belum menemukan Performer yang bekerja dengan baik, tetapi karena di makalah mereka melaporkan beberapa angka yang baik untuk tolok ukur protein, saya pikir saya akan memasukkannya dan mengizinkan orang lain untuk bereksperimen.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_linear = True # simply set this to True to use Performer for all cross attention
). cuda ()Anda juga dapat menentukan lapisan tepat yang ingin Anda gunakan perhatian linier dengan meneruskan tupel dengan panjang yang sama dengan kedalamannya
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 6 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_linear = ( True , False ) * 3 # interleave linear and full attention
). cuda ()Makalah ini menyarankan bahwa jika Anda memiliki pertanyaan atau konteks yang telah menentukan sumbu (misalnya gambar), Anda dapat mengurangi jumlah perhatian yang diperlukan dengan membuat rata-rata pada sumbu tersebut (tinggi dan lebar) dan menggabungkan sumbu rata-rata menjadi satu urutan. Anda dapat mengaktifkan ini sebagai teknik penghemat memori untuk perhatian silang, khususnya untuk urutan primer.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 6 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_kron_primary = True # make sure primary sequence undergoes the kronecker operator during cross attention
). cuda () Anda juga dapat menerapkan operator yang sama ke MSA selama perhatian silang dengan tanda cross_attn_kron_msa , jika MSA Anda sejajar dan memiliki lebar yang sama.
Semua yang harus dilakukan
Untuk menghemat memori untuk perhatian silang, Anda dapat mengatur rasio kompresi untuk kunci/nilai, mengikuti skema yang dijelaskan dalam makalah ini. Rasio kompresi 2-4 biasanya dapat diterima.
model = Alphafold2 (
dim = 256 ,
depth = 12 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_compress_ratio = 3
). cuda ()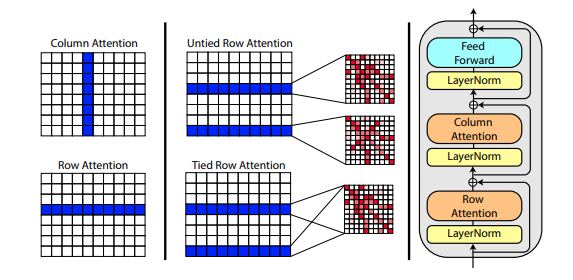
Sebuah makalah baru oleh Roshan Rao mengusulkan penggunaan perhatian aksial untuk pra-pelatihan MSA. Mengingat hasil yang kuat, repositori ini akan menggunakan skema yang sama di trunk, khususnya untuk perhatian mandiri MSA.
Anda juga dapat mengikat perhatian baris MSA dengan pengaturan msa_tie_row_attn = True pada inisialisasi Alphafold2 . Namun, untuk menggunakan ini, Anda harus memastikan bahwa jika Anda memiliki jumlah MSA yang tidak merata per urutan utama, bahwa masker MSA diatur dengan benar ke False untuk baris yang tidak digunakan.
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
msa_tie_row_attn = True # just set this to true
)Pemrosesan template juga sebagian besar dilakukan dengan perhatian aksial, dengan perhatian silang dilakukan sepanjang jumlah dimensi template. Hal ini sebagian besar mengikuti skema yang sama seperti pendekatan semua perhatian terhadap klasifikasi video seperti yang ditunjukkan di sini.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 5 ,
heads = 8 ,
dim_head = 64 ,
reversible = True ,
sparse_self_attn = False ,
max_seq_len = 256 ,
cross_attn_compress_ratio = 3
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 10 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
templates_seq = torch . randint ( 0 , 21 , ( 1 , 2 , 16 )). cuda ()
templates_coors = torch . randint ( 0 , 37 , ( 1 , 2 , 16 , 3 )). cuda ()
templates_mask = torch . ones_like ( templates_seq ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask ,
templates_seq = templates_seq ,
templates_coors = templates_coors ,
templates_mask = templates_mask
)Jika informasi sidechain juga ada, dalam bentuk vektor satuan antara koordinat C dan C-alpha setiap residu, Anda juga dapat meneruskannya sebagai berikut.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 5 ,
heads = 8 ,
dim_head = 64 ,
reversible = True ,
sparse_self_attn = False ,
max_seq_len = 256 ,
cross_attn_compress_ratio = 3
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 10 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
templates_seq = torch . randint ( 0 , 21 , ( 1 , 2 , 16 )). cuda ()
templates_coors = torch . randn ( 1 , 2 , 16 , 3 ). cuda ()
templates_mask = torch . ones_like ( templates_seq ). bool (). cuda ()
templates_sidechains = torch . randn ( 1 , 2 , 16 , 3 ). cuda () # unit vectors of difference of C and C-alpha coordinates
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask ,
templates_seq = templates_seq ,
templates_mask = templates_mask ,
templates_coors = templates_coors ,
templates_sidechains = templates_sidechains
)Saya telah menyiapkan implementasi ulang SE3 Transformer, seperti yang dijelaskan oleh Fabian Fuchs dalam postingan blog spekulasi.
Selain itu, makalah baru dari Victor dan Welling menggunakan fitur invarian untuk ekuivariansi E(n), mencapai SOTA dan mengungguli SE3 Transformer di sejumlah benchmark, sekaligus jauh lebih cepat. Saya telah mengambil ide utama dari makalah ini dan memodifikasinya menjadi transformator (menambahkan perhatian pada fitur dan mengoordinasikan pembaruan).
Ketiga jaringan ekivalen di atas telah terintegrasi dan tersedia untuk digunakan dalam repositori untuk penyempurnaan koordinat atom hanya dengan menyetel satu hyperparameter structure_module_type .
se3 SE3 Transformator
egnn EGNN
en E(n)-Transformator
Yang menarik bagi pembaca, masing-masing dari ketiga kerangka tersebut juga telah divalidasi oleh peneliti pada masalah terkait.
$ python setup.py test Perpustakaan ini akan menggunakan karya luar biasa dari Jonathan King di repositori ini. Terima kasih Jonatan!
Kami juga memiliki data MSA, semuanya senilai ~3,5 TB, diunduh dan dihosting oleh Archivist, pemilik proyek The-Eye. (Mereka juga menampung data dan model untuk Eleuther AI) Harap pertimbangkan untuk berdonasi jika Anda merasa terbantu.
$ curl -s https://the-eye.eu/eleuther_staging/globus_stuffs/tree.txthttps://xukui.cn/alphafold2.html
https://moalquraishi.wordpress.com/2020/12/08/alphafold2-casp14-it-feels-like-ones-child-has-left-home/

https://www.biorxiv.org/content/10.1101/2020.12.10.419994v1.full.pdf
https://pubmed.ncbi.nlm.nih.gov/33637700/
Presentasi lipat, dari laboratorium Tencent AI
cd downloads_folder > pip install pyrosetta_wheel_filename.whlBuka MM Kuning
@misc { unpublished2021alphafold2 ,
title = { Alphafold2 } ,
author = { John Jumper } ,
year = { 2020 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @article { Rao2021.02.12.430858 ,
author = { Rao, Roshan and Liu, Jason and Verkuil, Robert and Meier, Joshua and Canny, John F. and Abbeel, Pieter and Sercu, Tom and Rives, Alexander } ,
title = { MSA Transformer } ,
year = { 2021 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/02/13/2021.02.12.430858 } ,
journal = { bioRxiv }
} @article { Rives622803 ,
author = { Rives, Alexander and Goyal, Siddharth and Meier, Joshua and Guo, Demi and Ott, Myle and Zitnick, C. Lawrence and Ma, Jerry and Fergus, Rob } ,
title = { Biological Structure and Function Emerge from Scaling Unsupervised Learning to 250 Million Protein Sequences } ,
year = { 2019 } ,
doi = { 10.1101/622803 } ,
publisher = { Cold Spring Harbor Laboratory } ,
journal = { bioRxiv }
} @article { Elnaggar2020.07.12.199554 ,
author = { Elnaggar, Ahmed and Heinzinger, Michael and Dallago, Christian and Rehawi, Ghalia and Wang, Yu and Jones, Llion and Gibbs, Tom and Feher, Tamas and Angerer, Christoph and Steinegger, Martin and BHOWMIK, DEBSINDHU and Rost, Burkhard } ,
title = { ProtTrans: Towards Cracking the Language of Life{textquoteright}s Code Through Self-Supervised Deep Learning and High Performance Computing } ,
elocation-id = { 2020.07.12.199554 } ,
year = { 2021 } ,
doi = { 10.1101/2020.07.12.199554 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/05/04/2020.07.12.199554 } ,
eprint = { https://www.biorxiv.org/content/early/2021/05/04/2020.07.12.199554.full.pdf } ,
journal = { bioRxiv }
} @misc { king2020sidechainnet ,
title = { SidechainNet: An All-Atom Protein Structure Dataset for Machine Learning } ,
author = { Jonathan E. King and David Ryan Koes } ,
year = { 2020 } ,
eprint = { 2010.08162 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @misc { alquraishi2019proteinnet ,
title = { ProteinNet: a standardized data set for machine learning of protein structure } ,
author = { Mohammed AlQuraishi } ,
year = { 2019 } ,
eprint = { 1902.00249 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @misc { gomez2017reversible ,
title = { The Reversible Residual Network: Backpropagation Without Storing Activations } ,
author = { Aidan N. Gomez and Mengye Ren and Raquel Urtasun and Roger B. Grosse } ,
year = { 2017 } ,
eprint = { 1707.04585 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CV }
} @misc { fuchs2021iterative ,
title = { Iterative SE(3)-Transformers } ,
author = { Fabian B. Fuchs and Edward Wagstaff and Justas Dauparas and Ingmar Posner } ,
year = { 2021 } ,
eprint = { 2102.13419 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.LG }
} @misc { satorras2021en ,
title = { E(n) Equivariant Graph Neural Networks } ,
author = { Victor Garcia Satorras and Emiel Hoogeboom and Max Welling } ,
year = { 2021 } ,
eprint = { 2102.09844 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.LG }
} @misc { su2021roformer ,
title = { RoFormer: Enhanced Transformer with Rotary Position Embedding } ,
author = { Jianlin Su and Yu Lu and Shengfeng Pan and Bo Wen and Yunfeng Liu } ,
year = { 2021 } ,
eprint = { 2104.09864 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CL }
} @article { Gao_2020 ,
title = { Kronecker Attention Networks } ,
ISBN = { 9781450379984 } ,
url = { http://dx.doi.org/10.1145/3394486.3403065 } ,
DOI = { 10.1145/3394486.3403065 } ,
journal = { Proceedings of the 26th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining } ,
publisher = { ACM } ,
author = { Gao, Hongyang and Wang, Zhengyang and Ji, Shuiwang } ,
year = { 2020 } ,
month = { Jul }
} @article { Si2021.05.10.443415 ,
author = { Si, Yunda and Yan, Chengfei } ,
title = { Improved protein contact prediction using dimensional hybrid residual networks and singularity enhanced loss function } ,
elocation-id = { 2021.05.10.443415 } ,
year = { 2021 } ,
doi = { 10.1101/2021.05.10.443415 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/05/11/2021.05.10.443415 } ,
eprint = { https://www.biorxiv.org/content/early/2021/05/11/2021.05.10.443415.full.pdf } ,
journal = { bioRxiv }
} @article { Costa2021.06.02.446809 ,
author = { Costa, Allan and Ponnapati, Manvitha and Jacobson, Joseph M. and Chatterjee, Pranam } ,
title = { Distillation of MSA Embeddings to Folded Protein Structures with Graph Transformers } ,
year = { 2021 } ,
doi = { 10.1101/2021.06.02.446809 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/06/02/2021.06.02.446809 } ,
eprint = { https://www.biorxiv.org/content/early/2021/06/02/2021.06.02.446809.full.pdf } ,
journal = { bioRxiv }
} @article { Baek2021.06.14.448402 ,
author = { Baek, Minkyung and DiMaio, Frank and Anishchenko, Ivan and Dauparas, Justas and Ovchinnikov, Sergey and Lee, Gyu Rie and Wang, Jue and Cong, Qian and Kinch, Lisa N. and Schaeffer, R. Dustin and Mill{'a}n, Claudia and Park, Hahnbeom and Adams, Carson and Glassman, Caleb R. and DeGiovanni, Andy and Pereira, Jose H. and Rodrigues, Andria V. and van Dijk, Alberdina A. and Ebrecht, Ana C. and Opperman, Diederik J. and Sagmeister, Theo and Buhlheller, Christoph and Pavkov-Keller, Tea and Rathinaswamy, Manoj K and Dalwadi, Udit and Yip, Calvin K and Burke, John E and Garcia, K. Christopher and Grishin, Nick V. and Adams, Paul D. and Read, Randy J. and Baker, David } ,
title = { Accurate prediction of protein structures and interactions using a 3-track network } ,
year = { 2021 } ,
doi = { 10.1101/2021.06.14.448402 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/06/15/2021.06.14.448402 } ,
eprint = { https://www.biorxiv.org/content/early/2021/06/15/2021.06.14.448402.full.pdf } ,
journal = { bioRxiv }
}