Antarmuka R ke keluarga alat meme, yang menyediakan beberapa utilitas untuk melakukan analisis motif pada DNA, RNA, dan urutan protein. meme bekerja dengan mendeteksi instalasi lokal dari meme suite, menjalankan perintah, kemudian mengimpor hasil langsung ke R.
meme saat ini tersedia di cabang devel biokonduktor:
if ( ! requireNamespace( " BiocManager " , quietly = TRUE ))
install.packages( " BiocManager " )
# The following initializes usage of Bioc devel
BiocManager :: install( version = ' devel ' )
BiocManager :: install( " memes " )Anda dapat menginstal versi pengembangan meme dari GitHub dengan:
if ( ! requireNamespace( " remotes " , quietly = TRUE ))
install.packages( " remotes " )
remotes :: install_github( " snystrom/memes " )
# To temporarily bypass the R version 4.1 requirement, you can pull from the following branch:
remotes :: install_github( " snystrom/memes " , ref = " no-r-4 " ) # Get development version from dockerhub
docker pull snystrom/memes_docker:devel
# the -v flag is used to mount an analysis directory,
# it can be excluded for demo purposes
docker run -e PASSWORD= < password > -p 8787:8787 -v < path > / < to > / < project > :/mnt/ < project > snystrom/memes_docker:develMeme bergantung pada instalasi lokal dari meme suite. Untuk instruksi pemasangan untuk meme suite, lihat Panduan Instalasi Meme Suite.
Meme perlu mengetahui lokasi meme/bin/ direktori pada mesin lokal Anda. Anda dapat memberi tahu meme lokasi pemasangan meme suite Anda dalam 4 cara. Meme akan selalu lebih suka definisi yang lebih spesifik jika itu adalah jalur yang valid. Di sini mereka berada di peringkat dari sebagian besar hingga paling tidak spesifik:
meme_path dari semua fungsi memeoptions(meme_bin = "/path/to/meme/bin/") di dalam skrip r AndaMEME_BIN=/path/to/meme/bin/ di file .Renviron Anda~/meme/bin/Jika meme gagal mendeteksi instalasi Anda di lokasi yang ditentukan, itu akan kembali ke opsi berikutnya.
Untuk memverifikasi meme dapat mendeteksi instalasi meme Anda, gunakan check_meme_install() yang menggunakan herirarki pencarian di atas untuk menemukan instalasi meme yang valid. Ini akan melaporkan apakah ada alat yang hilang, dan mencetak jalan menuju meme yang dilihatnya. Ini bisa berguna untuk masalah pemecahan masalah dengan instalasi Anda.
library( memes )
# Verify that memes detects your meme install
# (returns all green checks if so)
check_meme_install()
# > checking main install
# > ✓ /opt/meme/bin
# > checking util installs
# > ✓ /opt/meme/bin/dreme
# > ✓ /opt/meme/bin/ame
# > ✓ /opt/meme/bin/fimo
# > ✓ /opt/meme/bin/tomtom
# > ✓ /opt/meme/bin/meme
# > x /opt/meme/bin/streme # You can manually input a path to meme_path
# If no meme/bin is detected, will return a red X
check_meme_install( meme_path = ' bad/path ' )
# > checking main install
# > x bad/path | Nama fungsi | Menggunakan | Input urutan | Input motif | Keluaran |
|---|---|---|---|---|
runStreme() | Penemuan motif (motif pendek) | Ya | TIDAK | universalmotif_df |
runDreme() | Penemuan motif (motif pendek) | Ya | TIDAK | universalmotif_df |
runAme() | Pengayaan Motif | Ya | Ya | Data.frame (Opsional: Kolom sequences ) |
runFimo() | Pemindaian motif | Ya | Ya | GRUT POSISI MOTIF |
runTomTom() | Perbandingan motif | TIDAK | Ya | universalmotif_df w/ best_match_motif dan kolom tomtom * |
runMeme() | Penemuan motif (motif panjang) | Ya | TIDAK | universalmotif_df |
* Catatan: Jika runTomTom() dijalankan menggunakan universalmotif_df hasilnya akan bergabung dengan hasil universalmotif_df sebagai kolom tambahan. Ini memungkinkan perbandingan mudah motif yang ditemukan De-Novo dengan pertandingan mereka.
Input urutan bisa saja:
Biostrings::XStringSet (dapat dihasilkan dari grange menggunakan fungsi helper get_sequence() )Biostrings::XStringSet Objects (dihasilkan oleh get_sequence() )Input motif bisa jadi salah satu dari:
universalmotif , atau daftar objek universalmotifrunDreme() (ini memungkinkan hasil runDreme() untuk diteruskan langsung ke runTomTom() )list() (misalnya list("path/to/database.meme", "dreme_results" = dreme_res) )Jenis keluaran :
runDreme() , runStreme() , runMeme() dan runTomTom() mengembalikan objek universalmotif_df yang merupakan data.frames dengan kolom khusus. Kolom motif berisi objek universalmotif , dengan 1 entri per baris. Kolom yang tersisa menggambarkan sifat dari setiap motif yang dikembalikan. Nama -nama kolom berikut adalah khusus karena nilainya digunakan saat menjalankan update_motifs() dan to_list() untuk mengubah properti motif yang disimpan di kolom motif . Berhati -hatilah tentang mengubah nilai -nilai ini karena perubahan ini akan merambat ke kolom motif saat memanggil update_motifs() atau to_list() .
meme dibangun di sekitar paket universalmotif yang menyediakan kerangka kerja untuk memanipulasi motif dalam objek R. universalmotif_df dapat menafsirkan antara data.frame dan format daftar universalmotif masing -masing menggunakan fungsi to_df() dan to_list() . Ini memungkinkan penggunaan hasil memes dengan semua paket motif biokonduktor lainnya, karena objek universalmotif dapat dikonversi ke jenis motif lainnya menggunakan convert_motifs() .
runTomTom() Mengembalikan kolom khusus: tomtom yang merupakan data.frame dari semua data kecocokan untuk setiap motif input. Ini dapat diperluas menggunakan tidyr::unnest(tomtom_results, "tomtom") , dan diganti dengan nest_tomtom() . Kolom best_match_ Prefixed yang dikembalikan oleh runTomTom() menunjukkan nilai untuk motif yang merupakan kecocokan terbaik dengan motif input.
suppressPackageStartupMessages(library( magrittr ))
suppressPackageStartupMessages(library( GenomicRanges ))
# Example transcription factor peaks as GRanges
data( " example_peaks " , package = " memes " )
# Genome object
dm.genome <- BSgenome.Dmelanogaster.UCSC.dm6 :: BSgenome.Dmelanogaster.UCSC.dm6 Fungsi get_sequence mengambil GRanges atau GRangesList sebagai input dan mengembalikan urutan sebagai BioStrings::XStringSet , atau daftar objek XStringSet , masing -masing. get_sequence akan menyebutkan setiap entri FASTA oleh koordinat genom dari setiap urutan dari.
# Generate sequences from 200bp about the center of my peaks of interest
sequences <- example_peaks % > %
resize( 200 , " center " ) % > %
get_sequence( dm.genome ) runDreme() menerima xStringset atau jalur ke file FASTA sebagai input. Anda dapat menggunakan sekuens lain atau sekuens input terseret sebagai dataset kontrol.
# runDreme accepts all arguments that the commandline version of dreme accepts
# here I set e = 50 to detect motifs in the limited example peak list
# In a real analysis, e should typically be < 1
dreme_results <- runDreme( sequences , control = " shuffle " , e = 50 ) meme dibangun di sekitar paket UniversalMotif. Hasilnya dikembalikan dalam format universalmotif_df , yang merupakan data R.Frame yang dapat dengan mulus antara data.frame dan format universalmotif menggunakan to_list() untuk dikonversi ke format daftar universalmotif , dan to_df() untuk mengonversi kembali ke data. Format frame. Menggunakan to_list() memungkinkan menggunakan hasil memes dengan semua fungsi universalmotif seperti itu:
library( universalmotif )
dreme_results % > %
to_list() % > %
view_motifs()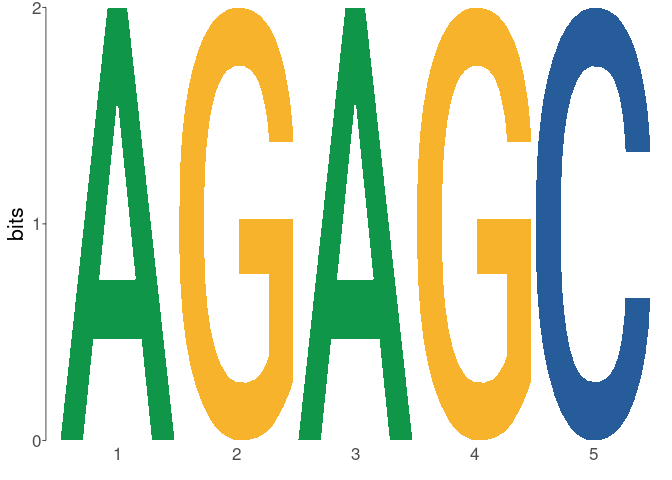
Motif yang ditemukan dapat dicocokkan dengan motif TF yang diketahui menggunakan runTomTom() , yang dapat menerima sebagai input jalur ke file .meme yang diformat, daftar universalmotif , atau hasil runDreme() .
TomTom menggunakan database motif yang diketahui yang dapat diteruskan ke parameter database sebagai jalur ke file format .meme, atau objek universalmotif .
Secara opsional, Anda dapat mengatur variabel lingkungan MEME_DB di .Renviron ke file pada disk, atau nilai meme_db dalam options ke file format .meme yang valid dan meme akan menggunakan file tersebut sebagai database. Meme akan selalu lebih suka input pengguna daripada panggilan fungsi daripada pengaturan variabel global.
options( meme_db = system.file( " extdata/flyFactorSurvey_cleaned.meme " , package = " memes " ))
m <- create_motif( " CMATTACN " , altname = " testMotif " )
tomtom_results <- runTomTom( m ) tomtom_results
# > motif name altname consensus alphabet strand icscore type
# > 1 <mot:motif> motif testMotif CMATTACN DNA +- 13 PPM
# > bkg best_match_name best_match_altname
# > 1 0.25, 0.25, 0.25, 0.25 prd_FlyReg prd
# > best_db_name best_match_offset best_match_pval best_match_eval
# > 1 flyFactorSurvey_cleaned 0 9.36e-05 0.052
# > best_match_qval best_match_strand
# > 1 0.0353 +
# > best_match_motif
# > 1 <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>
#> tomtom
#> 1 prd_FlyReg, tup_SOLEXA_10, CG13424_Cell, CG11085_Cell, BH2_Cell, CG13424_SOLEXA_2, Tup_Cell, Tup_SOLEXA, Bsh_Cell, Exex_SOLEXA, Odsh_SOLEXA, Unc4_Cell, Ubx_FlyReg, Unc4_SOLEXA, E5_Cell, inv_SOLEXA_5, BH2_SOLEXA, Zen_SOLEXA, CG33980_SOLEXA_2_10, BH1_SOLEXA, CG33980_SOLEXA_2_0, Hgtx_Cell, NK7.1_Cell, Slou_Cell, CG13424_SOLEXA, Zen2_Cell, AbdA_SOLEXA, Antp_SOLEXA, Btn_Cell, Dfd_SOLEXA, Eve_SOLEXA, Ftz_Cell, Hmx_SOLEXA, Hmx_Cell, CG34031_Cell, zen2_SOLEXA_2, En_Cell, Pb_SOLEXA, Slou_SOLEXA, Unpg_Cell, inv_SOLEXA_2, ovo_FlyReg, lim_SOLEXA_2, C15_SOLEXA, Ems_Cell, Btn_SOLEXA, Unpg_SOLEXA, Pb_Cell, Bsh_SOLEXA, Scr_SOLEXA, Zen2_SOLEXA, CG34031_SOLEXA, Eve_Cell, Pph13_Cell, BH1_Cell, CG11085_SOLEXA, CG32532_Cell, en_FlyReg, Dll_SOLEXA, Dfd_Cell, Dr_SOLEXA, Ap_Cell, Ro_Cell, CG4136_SOLEXA, CG33980_SOLEXA, Hbn_SOLEXA, Lbl_Cell, Otp_Cell, Rx_Cell, CG32532_SOLEXA, NK7.1_SOLEXA, Dr_Cell, Odsh_Cell, Al_SOLEXA, Antp_Cell, Hgtx_SOLEXA, Ftz_SOLEXA, Lab_SOLEXA, Dfd_FlyReg, Ap_SOLEXA, Awh_SOLEXA, CG11294_SOLEXA, CG4136_Cell, E5_SOLEXA, Ro_SOLEXA, PhdP_SOLEXA, CG12361_SOLEXA_2, Ind_Cell, Scr_Cell, CG9876_Cell, CG18599_Cell, CG9876_SOLEXA, Otp_SOLEXA, Lbl_SOLEXA, Ubx_Cell, Ubx_SOLEXA, en_SOLEXA_2, Pph13_SOLEXA, Rx_SOLEXA, CG15696_SOLEXA, CG18599_SOLEXA, Ems_SOLEXA, Repo_Cell, Dll_Cell, C15_Cell, CG12361_SOLEXA, Abd-A_FlyReg, Repo_SOLEXA, Zen_Cell, Inv_Cell, En_SOLEXA, Lim3_Cell, Lim1_SOLEXA, CG15696_Cell, Crc_CG6272_SANGER_5, Lab_Cell, CG32105_SOLEXA, Bap_SOLEXA, CG9437_SANGER_5, AbdA_Cell, pho_FlyReg, CG33980_Cell, Cad_SOLEXA, CG4328_SOLEXA, CG4328_Cell, Gsc_Cell, vri_SANGER_5, AbdB_SOLEXA, Xrp1_CG6272_SANGER_5, Al_Cell, Exex_Cell, br-Z4_FlyReg, CG11294_Cell, Aef1_FlyReg, CG7745_SANGER_5, PhdP_Cell, Awh_Cell, prd, tup, lms, CG11085, B-H2, lms, tup, tup, bsh, exex, OdsH, unc-4, Ubx, unc-4, E5, inv, B-H2, zen, CG33980, B-H1, CG33980, HGTX, NK7.1, slou, lms, zen2, abd-A, Antp, btn, Dfd, eve, ftz, Hmx, Hmx, CG34031, zen2, en, pb, slou, unpg, inv, ovo, Lim1, C15, ems, btn, unpg, pb, bsh, Scr, zen2, CG34031, eve, Pph13, B-H1, CG11085, CG32532, en, Dll, Dfd, Dr, ap, ro, CG4136, CG33980, hbn, lbl, otp, Rx, CG32532, NK7.1, Dr, OdsH, al, Antp, HGTX, ftz, lab, Dfd, ap, Awh, CG11294, CG4136, E5, ro, PHDP, CG12361, ind, Scr, CG9876, CG18599, CG9876, otp, lbl, Ubx, Ubx, en, Pph13, Rx, CG15696, CG18599, ems, repo, Dll, C15, CG12361, abd-A, repo, zen, inv, en, Lim3, Lim1, CG15696, crc, lab, CG32105, bap, CG9437, abd-A, pho, CG33980, cad, CG4328, CG4328, Gsc, vri, Abd-B, Xrp1, al, exex, br, CG11294, Aef1, CG7745, PHDP, Awh, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, 0, 1, 0, 1, 1, 1, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 1, 0, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 1, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 3, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, -1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, -2, 0, 0, 0, -3, 0, 4, 1, 0, 0, 0, 0, -2, 0, -2, 1, 0, -2, 1, 0, 2, -1, 0, 9.36e-05, 0.000129, 0.00013, 0.000194, 0.000246, 0.000247, 0.000319, 0.000364, 0.000427, 0.000435, 0.000473, 0.000473, 0.000556, 0.000643, 0.000694, 0.000728, 0.000748, 0.000748, 0.000785, 0.000828, 0.000849, 0.000868, 0.000868, 0.000868, 0.00102, 0.00113, 0.00116, 0.00116, 0.00116, 0.00116, 0.00116, 0.00116, 0.00118, 0.00126, 0.00134, 0.00141, 0.00144, 0.00145, 0.00145, 0.00154, 0.00161, 0.00165, 0.00173, 0.00177, 0.00177, 0.00179, 0.00179, 0.0019, 0.00191, 0.00191, 0.00191, 0.00203, 0.00203, 0.00203, 0.00212, 0.00214, 0.00219, 0.00232, 0.00242, 0.00247, 0.0026, 0.00264, 0.00264, 0.00268, 0.00282, 0.00282, 0.00282, 0.00282, 0.00282, 0.00286, 0.00286, 0.00296, 0.00305, 0.00316, 0.0032, 0.0032, 0.00326, 0.00326, 0.00341, 0.00348, 0.00348, 0.00348, 0.00348, 0.00348, 0.00348, 0.0035, 0.00359, 0.00359, 0.00359, 0.00364, 0.00372, 0.00372, 0.00372, 0.00387, 0.00387, 0.00412, 0.00415, 0.00424, 0.00424, 0.00439, 0.00452, 0.00452, 0.00467, 0.00483, 0.00497, 0.00497, 0.00528, 0.00549, 0.0059, 0.00597, 0.00624, 0.00635, 0.00709, 0.00761, 0.0079, 0.00856, 0.00859, 0.00912, 0.00935, 0.0097, 0.00974, 0.0101, 0.0109, 0.0116, 0.0123, 0.0123, 0.0127, 0.0138, 0.0138, 0.014, 0.014, 0.0147, 0.0148, 0.0155, 0.0165, 0.0166, 0.0177, 0.052, 0.0718, 0.0725, 0.108, 0.137, 0.137, 0.177, 0.202, 0.238, 0.242, 0.263, 0.263, 0.309, 0.357, 0.386, 0.405, 0.416, 0.416, 0.436, 0.46, 0.472, 0.483, 0.483, 0.483, 0.566, 0.629, 0.646, 0.646, 0.646, 0.646, 0.646, 0.646, 0.654, 0.702, 0.745, 0.785, 0.8, 0.808, 0.808, 0.858, 0.897, 0.92, 0.961, 0.985, 0.985, 0.993, 0.993, 1.05, 1.06, 1.06, 1.06, 1.13, 1.13, 1.13, 1.18, 1.19, 1.22, 1.29, 1.34, 1.38, 1.45, 1.47, 1.47, 1.49, 1.57, 1.57, 1.57, 1.57, 1.57, 1.59, 1.59, 1.65, 1.7, 1.76, 1.78, 1.78, 1.81, 1.81, 1.9, 1.94, 1.94, 1.94, 1.94, 1.94, 1.94, 1.95, 2, 2, 2, 2.02, 2.07, 2.07, 2.07, 2.15, 2.15, 2.29, 2.31, 2.36, 2.36, 2.44, 2.52, 2.52, 2.6, 2.68, 2.76, 2.76, 2.94, 3.05, 3.28, 3.32, 3.47, 3.53, 3.94, 4.23, 4.39, 4.76, 4.77, 5.07, 5.2, 5.39, 5.41, 5.61, 6.06, 6.42, 6.81, 6.81, 7.03, 7.66, 7.65, 7.78, 7.78, 8.15, 8.26, 8.59, 9.18, 9.2, 9.85, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0368, 0.0369, 0.0369, 0.0369, 0.0369, 0.0369, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0379, 0.0379, 0.0381, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0404, 0.0404, 0.0424, 0.0424, 0.0424, 0.0424, 0.0435, 0.0439, 0.0439, 0.0449, 0.046, 0.0464, 0.0464, 0.0489, 0.0504, 0.0536, 0.0538, 0.0557, 0.0562, 0.0622, 0.0662, 0.0681, 0.0727, 0.0727, 0.0765, 0.0778, 0.0797, 0.0797, 0.0819, 0.0877, 0.0923, 0.0964, 0.0964, 0.0987, 0.106, 0.106, 0.106, 0.106, 0.11, 0.111, 0.114, 0.121, 0.121, 0.128, +, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, +, -, -, -, +, -, -, -, -, -, -, -, +, -, +, -, -, -, +, +, +, -, -
# >
# > [Hidden empty columns: family, organism, nsites, bkgsites, pval, qval,
# > eval.] runTomTom() akan menambahkan hasilnya sebagai kolom ke data hasil runDreme() .
full_results <- dreme_results % > %
runTomTom() AME digunakan untuk menguji pengayaan motif yang diketahui dalam urutan target. runAme() akan menggunakan entri MEME_DB di .Renviron atau options(meme_db = "path/to/database.meme") sebagai database motif. Bergantian, itu akan menerima semua input yang valid yang mirip dengan runTomTom() .
# here I set the evalue_report_threshold = 30 to detect motifs in the limited example sequences
# In a real analysis, evalue_report_threshold should be carefully selected
ame_results <- runAme( sequences , control = " shuffle " , evalue_report_threshold = 30 )
ame_results
# > # A tibble: 2 x 17
# > rank motif_db motif_id motif_alt_id consensus pvalue adj.pvalue evalue tests
# > <int> <chr> <chr> <chr> <chr> <dbl> <dbl> <dbl> <int>
# > 1 1 /usr/lo… Eip93F_… Eip93F ACWSCCRA… 5.14e-4 0.0339 18.8 67
# > 2 2 /usr/lo… Cf2-PB_… Cf2 CSSHNKDT… 1.57e-3 0.0415 23.1 27
# > # … with 8 more variables: fasta_max <dbl>, pos <int>, neg <int>,
# > # pwm_min <dbl>, tp <int>, tp_percent <dbl>, fp <int>, fp_percent <dbl> view_tomtom_hits memungkinkan membandingkan motif input dengan hit teratas dari TomTom. Inspeksi manual pertandingan ini adalah penting, karena terkadang pertandingan teratas tidak selalu merupakan tugas yang benar. Mengubah top_n memungkinkan Anda untuk menunjukkan kecocokan tambahan dalam urutan peringkat mereka.
full_results % > %
view_tomtom_hits( top_n = 1 )
# > $m01_AGAGC 
Dapat bermanfaat untuk melihat hasil dari runAme() sebagai pemanas. plot_ame_heatmap() dapat membuat visualisasi yang kompleks untuk analisis pengayaan antara berbagai jenis wilayah (lihat sketsa untuk detailnya). Berikut ini adalah contoh panas yang sederhana.
ame_results % > %
plot_ame_heatmap()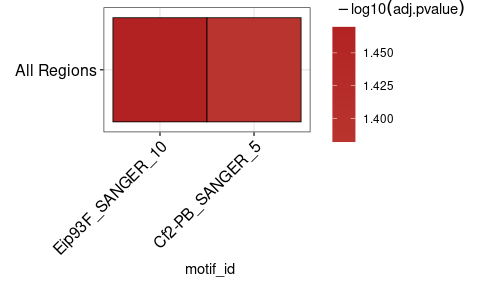
Alat FIMO digunakan untuk mengidentifikasi kecocokan dengan motif yang diketahui. runFimo akan mengembalikan hits ini sebagai objek GRanges yang mengandung koordinat genom dari pertandingan motif.
# Query MotifDb for a motif
e93_motif <- MotifDb :: query( MotifDb :: MotifDb , " Eip93F " ) % > %
universalmotif :: convert_motifs()
# > See system.file("LICENSE", package="MotifDb") for use restrictions.
# Scan for the E93 motif within given sequences
fimo_results <- runFimo( sequences , e93_motif , thresh = 1e-3 )
# Visualize the sequences matching the E93 motif
plot_sequence_heatmap( fimo_results $ matched_sequence ) 
Meme juga mendukung mengimpor hasil yang dihasilkan menggunakan meme suite di luar R (misalnya, menjalankan pekerjaan di meme-uite.org, atau berjalan pada garis komando). Ini memungkinkan penggunaan hasil meme suite yang sudah ada sebelumnya dengan fungsi meme hilir.
| Alat Meme | Nama fungsi | Jenis file |
|---|---|---|
| Streme | importStremeXML() | streme.xml |
| Dreme | importDremeXML() | dreme.xml |
| Tomtom | importTomTomXML() | tomtom.xml |
| Ame | importAme() | ame.tsv* |
| Fimo | importFimo() | fimo.tsv |
| Meme | importMeme() | meme.txt |
* importAME() juga dapat menggunakan output "Sequences.tsv" ketika AME menggunakan method = "fisher" , ini opsional.
MEME Suite saat ini tidak mendukung Windows, meskipun dapat diinstal di bawah Cygwin atau Windows Linux Subsytem (WSL). Harap dicatat bahwa jika meme dipasang di Cygwin atau WSL, Anda juga harus menjalankan R di dalam Cygwin atau WSL untuk menggunakan meme.
Solusi alternatif adalah menggunakan Docker untuk menjalankan lingkungan virtual dengan meme suite terpasang. Kami menyediakan wadah Docker meme
Itu dikirimkan dengan meme suite, r studio, dan semua ketergantungan memes yang telah diinstal sebelumnya.
Meme adalah pembungkus untuk beberapa alat tertentu dari meme suite, yang dikembangkan oleh kelompok lain. Selain mengutip meme, silakan mengutip alat meme suite yang sesuai dengan alat yang Anda gunakan.
Jika Anda menggunakan runDreme() dalam analisis Anda, silakan kutip:
Timothy L. Bailey, "Dreme: Penemuan Motif dalam Faktor Transkripsi Data Chip-Seq", Bioinformatika, 27 (12): 1653-1659, 2011. Teks Lengkap
Jika Anda menggunakan runTomTom() dalam analisis Anda, silakan kutip:
Shobhit Gupta, Ja Stamatoyannopolous, Timothy Bailey dan William Stafford Noble, “Mengukur Kesamaan Antara Motif”, Genome Biology, 8 (2): R24, 2007. Teks Lengkap
Jika Anda menggunakan runAme() dalam analisis Anda, silakan kutip:
Robert McLeay dan Timothy L. Bailey, “Analisis Pengayaan Motif: Kerangka Kerja Terpadu dan Evaluasi Metode”, BMC Bioinformatics, 11: 165, 2010, doi: 10.1186/1471-2105-11-165. teks lengkap
Jika Anda menggunakan runFimo() dalam analisis Anda, silakan kutip:
Charles E. Grant, Timothy L. Bailey, dan William Stafford Noble, "Fimo: Pemindaian untuk kejadian motif yang diberikan", Bioinformatika, 27 (7): 1017-1018, 2011. Teks Lengkap
Meme Suite gratis untuk penggunaan nirlaba, tetapi pengguna nirlaba harus membeli lisensi. Lihat halaman Hak Cipta meme Suite untuk detailnya.