ANCOM Code Archive
Third release of ANCOM
이 저장소는 보관 목적으로만 사용됩니다. 이 저장소의 독립형 ANCOM 기능은 더 이상 유지되지 않습니다. 사용자는 ANCOMBC R 패키지에서 ancom 또는 ancombc 기능을 사용하는 것이 좋습니다. 버그 보고서를 보려면 ANCOMBC 저장소로 이동하세요.
현재 코드는 공변량 사용을 허용하면서 단면 및 종단 데이터 세트에서 ANCOM을 구현합니다. R 코드를 실행하려면 다음 라이브러리가 포함되어야 합니다.
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )우리는 차등 존재비 분석을 수행하기 전에 다양한 유형의 0을 처리하기 위한 전처리 단계로 ANCOM-II의 방법론을 채택했습니다.
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : 행( rownames )에 분류군이 있고 열( colnames )에 샘플이 있는 관찰된 OTU/SV 테이블을 나타내는 데이터 프레임 또는 행렬입니다. 이것은 절대 존재비 표이므로 상대 존재비 표(열 합계가 1과 같음)로 변환하지 마십시오.meta_data : 관심 있는 모든 변수 및 공변량의 데이터 프레임 또는 행렬.sample_var : 문자. 샘플 ID를 저장하는 열의 이름입니다.group_var : 캐릭터. 그룹 표시기의 이름입니다. group_var 는 구조적 0과 이상값을 감지하는 데 필요합니다. 다양한 0(구조적 0, 이상값 0, 샘플링 0)에 대한 정의는 ANCOM-II를 참조하세요.out_cut 0과 1 사이의 숫자 분수입니다. 각 분류군에 대해 out_cut 보다 작은 혼합 분포 비율의 관찰은 이상치 0으로 감지됩니다. 혼합 분포 비율이 1 - out_cut 이상값으로 감지됩니다.zero_cut : 0과 1 사이의 숫자 분수. zero_cut 보다 큰 0의 비율을 가진 분류군은 분석에 포함되지 않습니다.lib_cut : 숫자. lib_cut 보다 작은 라이브러리 크기를 가진 샘플은 분석에 포함되지 않습니다.neg_lb : 논리적입니다. TRUE는 분류군이 점근적 하한을 사용하여 해당 실험 그룹에서 구조적 0으로 분류된다는 것을 나타냅니다. 보다 구체적으로 neg_lb = TRUE 구조적 0을 감지하기 위해 ANCOM-II의 섹션 3.2에 명시된 두 기준을 모두 사용하고 있음을 나타냅니다. 그렇지 않으면 neg_lb = FALSE 구조적 0을 선언하기 위해 ANCOM-II 섹션 3.2의 방정식 1만 사용합니다. feature_table : 전처리된 OTU 테이블의 데이터 프레임입니다.meta_data : 전처리된 메타데이터의 데이터 프레임입니다.structure_zeros : 매트릭스는 0과 1로 구성되며 1은 분류군이 해당 그룹에서 구조적 0으로 식별됨을 나타냅니다.ANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : 행( rownames )에 분류가 있고 열( colnames )에 샘플이 있는 OTU/SV 테이블을 나타내는 데이터 프레임입니다. feature_table_pre_process 의 출력 값일 수 있습니다. 이것은 절대 존재비 표이므로 상대 존재비 표(열 합계가 1과 같음)로 변환하지 마십시오.meta_data : 변수의 데이터 프레임입니다. feature_table_pre_process 의 출력 값일 수 있습니다.struc_zero : 매트릭스는 0과 1로 구성되며 1은 분류군이 해당 그룹에서 구조적 0으로 식별됨을 나타냅니다. feature_table_pre_process 의 출력 값일 수 있습니다.main_var : 캐릭터. 관심 있는 주요 변수의 이름입니다. ANCOM v2.1은 현재 범주형 main_var 지원합니다.p_adjust_method : 문자. 다중 비교를 위해 p-값을 조정하는 방법을 지정합니다. 기본값은 "BH"(Benjamini-Hochberg 절차)입니다.alpha : 유의 수준. 기본값은 0.05입니다.adj_formula : 조정 공식을 나타내는 문자열입니다(예제 참조).rand_formula : lme 의 무작위 효과 공식을 나타내는 문자열입니다. 자세한 내용은 ?lme 참조하세요.lme_control : lme fit에 대한 제어 값을 지정하는 목록입니다. 자세한 내용은 ?lmeControl 참조하세요. 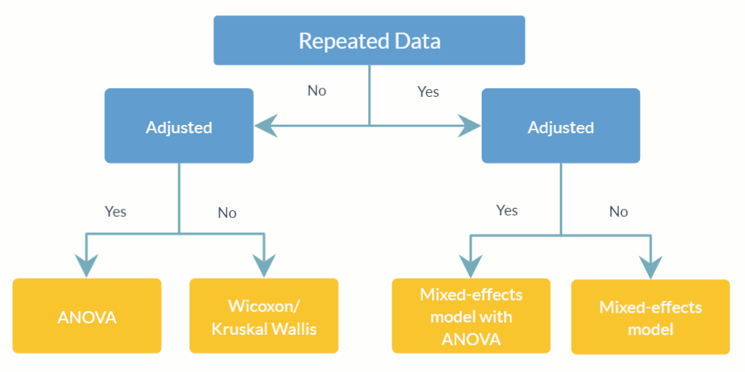
out : OTU 또는 분류군이 일련의 컷오프(0.9, 0.8, 0.7 및 0.6)에서 차별적으로 풍부한지 여부를 논리적으로 나타내는 각 분류군 및 후속 열에 대한 W 통계가 포함된 데이터 프레임입니다. detected_0.7 이 일반적으로 사용됩니다. 그러나 결과를 더 보수적으로(더 작은 FDR) 원할 경우 detected_0.8 또는 detected_0.9 선택할 수 있고, 더 많은 발견(더 큰 검정력)을 탐색하려면 detected_0.6 사용할 수 있습니다.
fig : 화산 플롯의 ggplot 객체.
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var 지정하여 구조적 0을 식별합니다. 여기서 우리는 다양한 delivery 수준에 걸쳐 구조적 제로가 있는지 알고 싶습니다. library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var 지정하여 구조적 0을 식별합니다. 여기서 우리는 다양한 delivery 수준에 걸쳐 구조적 제로가 있는지 알고 싶습니다. library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var 지정하여 구조적 0을 식별합니다. 여기서 우리는 다양한 delivery 수준에 걸쳐 구조적 제로가 있는지 알고 싶습니다. library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )