논문에서 벤치마킹된 DiffAbXL의 구현: 항체 서열 디자인 순위 지정을 위한 로그 우도 점수 탐색.
| 계급 | 모델 | 앱시 HER2 | 자연 | AZ 타겟-2 | ? | ||
|---|---|---|---|---|---|---|---|
| 제로샷 | SPR 제어 | 헬 | HER2 | ||||
| 1 | DiffAbXL-A-DN | 0.43 | 0.22 | 0.62 | 0.37 | 0.41 | 0.41 |
| 2 | DiffAbXL-A-SG | 0.46 | 0.22 | 0.64 | -0.38 | 0.43 | 0.274 |
| 3 | DiffAbXL-H3-DN | 0.49 | 0 | 0.52 | -0.08 | 0.37 | 0.26 |
| 4 | IgBlend(구조체에만 해당) | 0.40 | 0.21 | 0.54 | -0.30 | 0.31 | 0.232 |
| 5 | 안티폴드 | 0.43 | 0.22 | 0.4 | -0.47 | 0.38 | 0.192 |
| 6 | DiffAbXL-H3-SG | 0.48 | 0 | 0.4 | -0.41 | 0.29 | 0.152 |
| 7 | ESM | 0.29 | 0 | 0 | 0.18 | 0.27 | 0.148 |
| 8 | DiffAb | 0.34 | 0.21 | 0 | -0.14 | 0.22 | 0.126 |
| 9 | 앱랭2 | 0.3 | 0 | 0 | -0.07 | 0.36 | 0.118 |
| 10 | IgBlend(순서에만 해당) | 0.27 | 0 | 0 | -0.1 | 0.36 | 0.106 |
| 11 | 앱랭 | 0.3 | 0 | 0 | -0.13 | 0.35 | 0.104 |
| 12 | dyMEAN | 0.37 | 0.15 | 0 | 0 | 0 | 0.104 |
| 13 | AbX | 0.28 | 0.19 | 0 | 0 | 0 | 0.094 |
| 14 | 안티버티 | 0.26 | 0 | 0 | -0.17 | 0.35 | 0.088 |
| 15 | 평균 | 0.36 | 0 | 0 | 0.02 | 0 | 0.076 |
| 16 | ESM-IF | 0 | -0.27 | 0 | -0.53 | 0.42 | -0.076 |
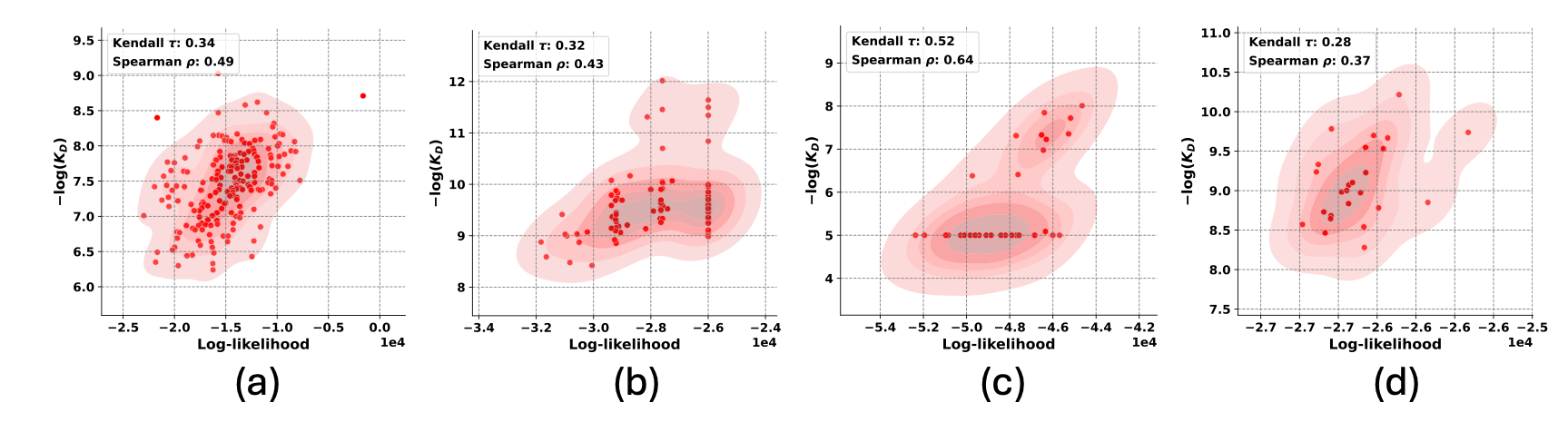
그림-1: DiffAbXL에 대한 결과: a) Absci 제로샷 HER2 데이터에 대한 DiffAbXL-H3-DN b) AZ Target-2에 대한 DiffAbXL-A-SG, c) Nature HEL에 대한 DiffAbXL-A-SG, d) DiffAbXL- Nature HER2의 경우 A-DN입니다.
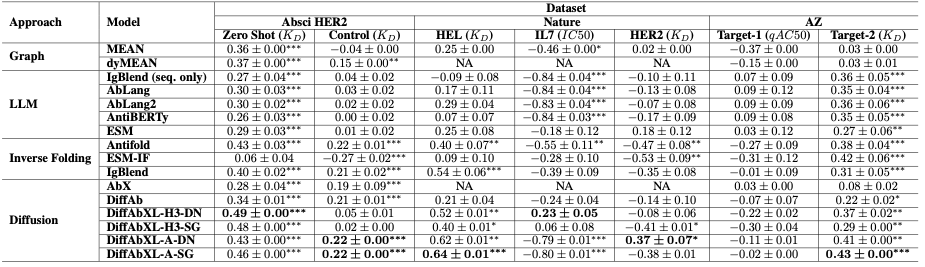
표-1: Spearman 상관관계에 대한 결과 요약. 약어: DN: De Novo 모드, SG: 구조 안내 모드, NA: 에피토프 또는 복합 구조가 필요하지만 사용할 수 없습니다. *, **, ***는 각각 0.05, 0.01 및 1e-4 미만의 p-값을 나타냅니다.
모델을 더 쉽게 벤치마킹할 수 있도록 평가 파이프라인과 쉽게 통합할 수 있는 클래스에서 인터페이스를 Python 메서드로 구현하는 것이 좋습니다. 이 메서드는 다음 입력을 허용해야 합니다.
이 메서드는 다음을 포함하는 사전을 반환해야 합니다.
다음은 이 인터페이스를 구현하기 위한 Python의 기본 템플릿입니다.
def benchmark ( self , sequences , structure = None , mask = None , ** kwargs ):
"""
Benchmark the model on provided antibody sequences and structures.
Parameters:
sequences (list of str): List of antibody sequences.
structure (optional): Path to a PDB file. Currently, only one PDB file is provided per target dataset.
The PDB file may contain either just the antibody or an antibody-antigen complex,
depending on the dataset.
mask (optional): Binary list or array indicating the regions of interest in the sequences for metric calculations.
kwargs (optional): Additional parameters required by the model.
Returns:
dict: A dictionary containing log-likelihood scores and other relevant metrics.
"""
pass모델이 항체 서열 설계 벤치마킹에 직접 사용할 수 있는 형식으로 로그 우도 점수를 출력하는지 확인하십시오. 이를 통해 데이터 세트 전체에서 모델 성능을 효율적으로 비교할 수 있습니다.
매개변수를 변경하는 데 사용할 수 있는 구성 파일 sabdab.yaml이 하나 있습니다. 다음을 사용하여 모델을 훈련할 수 있습니다.
python train.py # For training.
- train.py
- 소스
|-model.py
- 구성
|-sabdab.yaml
- 유틸리티
|-load_data.py
|-arguments.py
|-model_utils.py
|-loss_functions.py
...
- 데이터
|-그녀2
...
가중치 및 편향을 사용하여 실험을 추적할 수 있습니다. 기본적으로 꺼져 있지만 ./config/sabdab.yaml 의 구성 파일에서 옵션을 변경하여 켤 수 있습니다.
@article {Ucar2024.10.07.617023,
author = {Ucar, Talip and Malherbe, Cedric and Gonzalez Hernandez, Ferran},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
elocation-id = {2024.10.07.617023},
year = {2024},
doi = {10.1101/2024.10.07.617023},
publisher = {Cold Spring Harbor Laboratory},
URL = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023},
eprint = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023.full.pdf},
journal = {bioRxiv}
}
자신의 연구 및 작업에서 DiffAbXL을 사용하는 경우 다음을 사용하여 인용하십시오.
@Misc{talip_ucar_2024_DiffAbXL,
author = {Talip Ucar},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
URL = {https://github.com/AstraZeneca/DiffAbXL},
month = {October},
year = {since 2024}
}