
Isling은 짝을 이루는 독서에서 바이러스 또는 벡터 통합을 감지하는 도구입니다. 자세한 내용은 논문을 읽으십시오.
conda 및 snakemake 설치된 경우 (포함 된) 테스트 데이터와 함께 로컬로 실행합니다.
git clone https://github.com/aehrc/isling.git && cd isling
snakemake --configfile test/config/test.yml --cores <cores> --use-conda
snakemake 및 singularity 설치되어 있으면 대신 사용할 수 있습니다.
snakemake --configfile test/config/test.yml --cores <cores> --use-singularity
또는 Docker를 설치 한 경우 MacOS에서는 다음을 수행 할 수 있습니다.
docker run --rm -it -v"$(pwd)"/out:/opt/isling/out szsctt/isling:latest snakemake --configfile test/config/test.yml --cores 1
이렇게하면 컨테이너 내부의 구성 파일과 데이터가 사용되며 결과는 현재 작업 디렉토리에서 out 된 폴더에 나타납니다. Linux에서는이 명령을 루트로 실행해야하며 Windows에서는 Bind -Mount 구문 ( -v argument)을 조정해야합니다.
입력 데이터 (읽기 및 호스트 및 바이러스 성 참조)는 ni 구성 파일을 지정합니다. 자체 데이터의 경우 자체 데이터를 가리 키려면 예제 구성 파일 ( test/config/test.yml )을 수정해야합니다. 구성 파일 형식에 대한 자세한 내용은 configfile.md 참조하십시오.
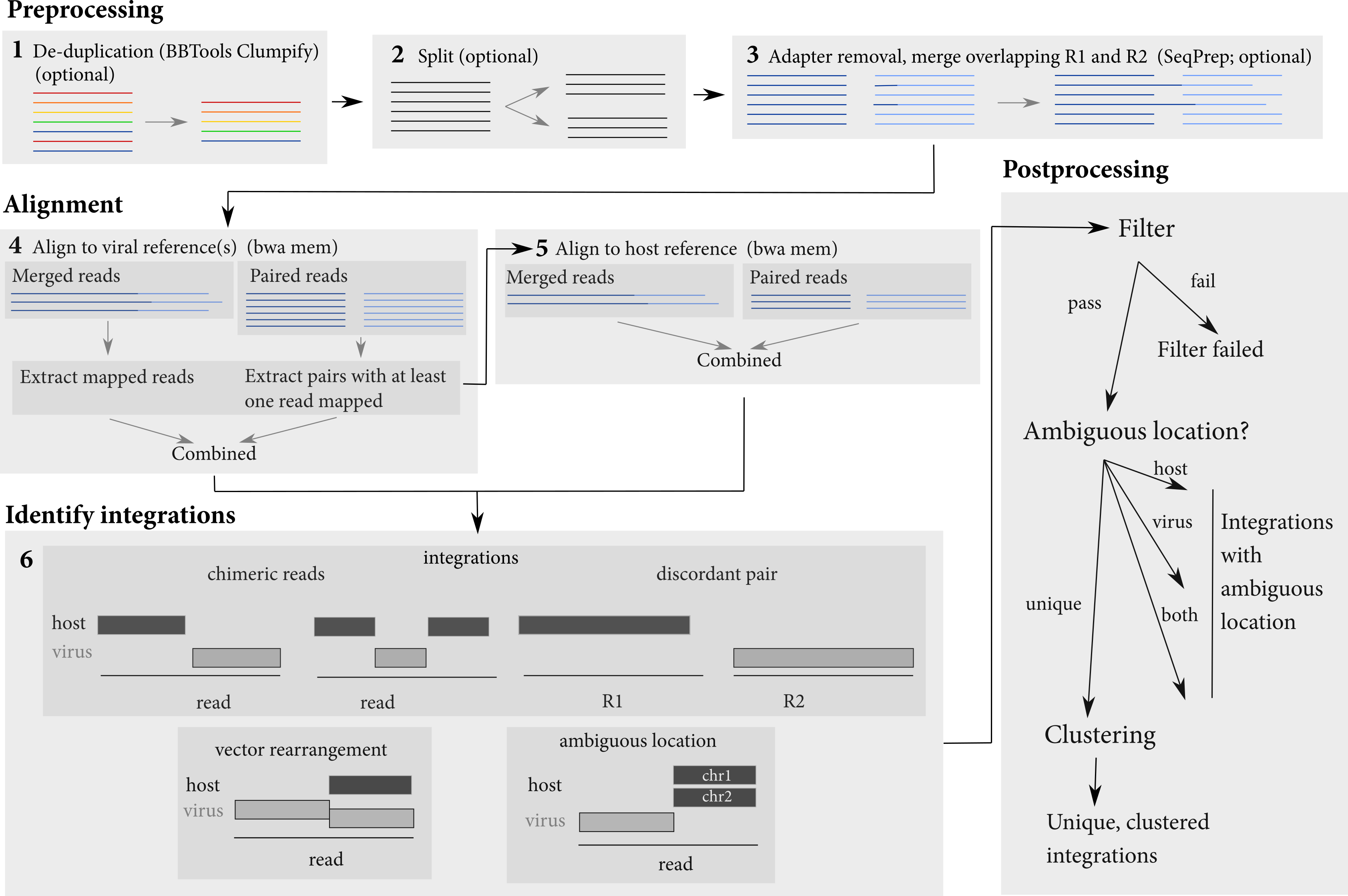
파이프 라인은 통합 사이트를 식별하기 위해 여러 단계를 수행합니다. FASTQ 파일 또는 BAM 파일로 구성된 입력 데이터 세트로 사용됩니다. 읽기 (중첩 읽기 병합, 선택 사항)의 사전 처리를 수행 한 다음 호스트 및 바이러스 시퀀스 모두에 정렬합니다. 읽기는 먼저 바이러스 서열 (들)에 정렬 된 다음, 정렬 된 판독 값이 추출되어 숙주에 정렬된다. 이러한 정렬은 바이러스 통합을 식별하는 데 사용됩니다.
Isling은 snakemake 와 singularity (권장) 또는 conda 필요합니다. 추가, python 버전 3.5 이상 및 pandas 필요합니다 ( conda 와 함께 snakemake 설치하는 경우 자동으로 설치해야합니다.
Alternativley, Isling 및 모든 종속성을 포함하는 Docker 버전을 사용하십시오.
필요한 입력은 구성 파일로 호스트 및 바이러스/벡터 참조를 명시하고 읽기가 필요합니다. 구성 파일에서 모든 입력을 지정합니다. Isling은 현재 짝을 이루는 읽기에만 작동합니다.
이 구성 파일의 형식에 대한 설명은 file configfile.md 참조하십시오.
Isling은 구성 파일에 지정된 출력 디렉토리의 탭 구분 형식으로 통합 사이트를 출력합니다.
출력 폴더 내에서 구성 파일의 각 데이터 세트에 대해 하나의 폴더가 생성되고 각 데이터 세트에 대해 ints Directory에서 통합을 찾을 수 있습니다. 각 샘플에 대해 하나의 출력 파일 세트가 있습니다.
각 샘플에 대해 특정 사용 사례에 관심이있을 수있는 여러 출력 파일이 있습니다.
<sample>.<host>.<virus>.integrations.txt : 필터를 통과했는지 여부에 관계없이 감지 된 모든 접합부에 대한 정보<sample>.<host>.<virus>.integrations.post.txt : 모든 필터를 통과 한 감지 된 접합에 대한 정보<sample>.<host>.<virus>.integrations.post.unique.txt<sample>.<host>.<virus>.integrations.post.unique.merged.txt : 호스트 및 바이러스/벡터 모두에서 동일한 좌표로 겹치는 접합 또는 접합을 병합 한 결과. 병합은 호스트 및 바이러스/벡터 모두에서 모호하지 않은 위치를 가진 통합 접합으로 만 수행됩니다.<sample>.<host>.<virus>.integrations.post.host_ambig.txt : 모든 필터를 전달하고 모호하지 않은 위치 벡터/바이러스가 있지만 호스트에서 모호한 위치를 갖는 감지 된 접합에 대한 정보<sample>.<host>.<virus>.integrations.post.virus_ambig.txt<sample>.<host>.<virus>.integrations.post.both_ambig.txt : 모든 필터를 통과하고 호스트 및 벡터/바이러스 모두에서 모호한 위치를 가진 감지 된 접합에 대한 정보 출력 파일은 식별 된 통합의 위치와 그 속성을 제공합니다. 통합 접합의 좌표는 모호한베이스 측면에서 지정됩니다. 즉, 종종 읽기의 숙주와 바이러스 부분 사이에 틈이 있거나 중복됩니다. 
이 경우 통합의 위치를 고유하게 결정할 수 없기 때문에, Isling은 호스트 및 벡터/바이러스 게놈에서 이러한 염기의 좌표를 통합의 위치로 출력합니다.
병합 된 클러스터 출력 파일을 제외하고 모든 파일에는 다음과 같은 열이 포함됩니다.
clean 키미어 읽기 (동일한 읽기에서 식별 된 숙주 및 바이러스 부품)에 대한 gap overlap 때문에 모호한 염기가 발생했는지 여부를 지정합니다. 또는 Jucntion이 불일치 한 쌍으로 식별 된 경우, OverlapType 은 discordanthv ) 또는 바이러스/숙주 ( vh )+ 또는 -bwa 에서)bwa 에서) 편집 거리 편집OverlapType 이 gap 인 경우 모호한베이스의 수chimeric 읽기, discordant 쌍에서 관찰 된 접합부는 또는 동일한 읽기에서 두 가지 접합부가 관찰되는 short 통합입니까?bwa 에서)bwa 에서)'xxx' 로 분리됩니다. Isling 원고의 수치를 재현하려면 benchmarking 디렉토리의 readme를 참조하십시오.