A implementação do DiffAbXL avaliada no artigo: Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs.
| Classificação | Modelos | Absci HER2 | Natureza | Alvo AZ-2 | Avenida. ? | ||
|---|---|---|---|---|---|---|---|
| Tiro Zero | Controle SPR | HEL | HER2 | ||||
| 1 | DiffAbXL-A-DN | 0,43 | 0,22 | 0,62 | 0,37 | 0,41 | 0,41 |
| 2 | DiffAbXL-A-SG | 0,46 | 0,22 | 0,64 | -0,38 | 0,43 | 0,274 |
| 3 | DiffAbXL-H3-DN | 0,49 | 0 | 0,52 | -0,08 | 0,37 | 0,26 |
| 4 | IgBlend (somente estrutura) | 0,40 | 0,21 | 0,54 | -0,30 | 0,31 | 0,232 |
| 5 | Antidobra | 0,43 | 0,22 | 0,4 | -0,47 | 0,38 | 0,192 |
| 6 | DiffAbXL-H3-SG | 0,48 | 0 | 0,4 | -0,41 | 0,29 | 0,152 |
| 7 | MEE | 0,29 | 0 | 0 | 0,18 | 0,27 | 0,148 |
| 8 | DifAb | 0,34 | 0,21 | 0 | -0,14 | 0,22 | 0,126 |
| 9 | AbLang2 | 0,3 | 0 | 0 | -0,07 | 0,36 | 0,118 |
| 10 | IgBlend (apenas seq.) | 0,27 | 0 | 0 | -0,1 | 0,36 | 0,106 |
| 11 | AbLang | 0,3 | 0 | 0 | -0,13 | 0,35 | 0,104 |
| 12 | dyMEAN | 0,37 | 0,15 | 0 | 0 | 0 | 0,104 |
| 13 | AbX | 0,28 | 0,19 | 0 | 0 | 0 | 0,094 |
| 14 | AntiBERTy | 0,26 | 0 | 0 | -0,17 | 0,35 | 0,088 |
| 15 | SIGNIFICAR | 0,36 | 0 | 0 | 0,02 | 0 | 0,076 |
| 16 | ESM-IF | 0 | -0,27 | 0 | -0,53 | 0,42 | -0,076 |
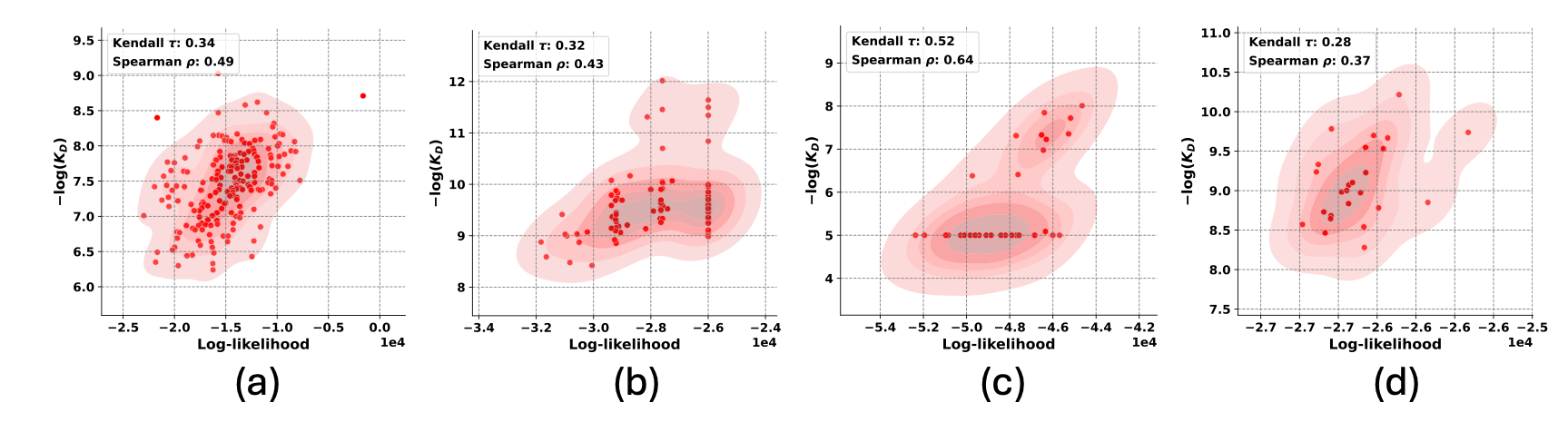
Figura 1: Resultados para DiffAbXL: a) DiffAbXL-H3-DN para dados Absci zero-shot HER2 b) DiffAbXL-A-SG para AZ Target-2, c) DiffAbXL-A-SG para Nature HEL, d) DiffAbXL- A-DN para Natureza HER2.
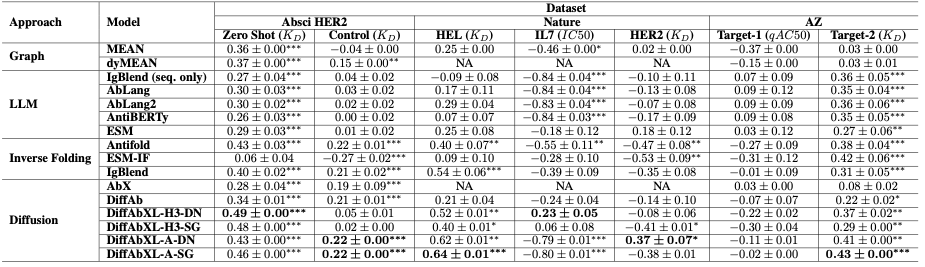
Tabela 1: Resumo dos resultados da correlação de Spearman. Abreviaturas: DN: modo De Novo, SG: modo Structure Guidance, NA: epítopo ou estrutura complexa necessária, mas não disponível. *, **, *** indicam valores de p abaixo de 0,05, 0,01 e 1e-4 respectivamente.
Para facilitar o benchmarking do seu modelo, recomendamos que você implemente uma interface como um método Python em uma classe que possamos integrar facilmente ao nosso pipeline de avaliação. O método deve aceitar as seguintes entradas:
O método deve retornar um dicionário contendo:
Aqui está um modelo básico em Python para implementar esta interface:
def benchmark ( self , sequences , structure = None , mask = None , ** kwargs ):
"""
Benchmark the model on provided antibody sequences and structures.
Parameters:
sequences (list of str): List of antibody sequences.
structure (optional): Path to a PDB file. Currently, only one PDB file is provided per target dataset.
The PDB file may contain either just the antibody or an antibody-antigen complex,
depending on the dataset.
mask (optional): Binary list or array indicating the regions of interest in the sequences for metric calculations.
kwargs (optional): Additional parameters required by the model.
Returns:
dict: A dictionary containing log-likelihood scores and other relevant metrics.
"""
passCertifique-se de que seu modelo produza as pontuações de log-verossimilhança em um formato que possamos usar diretamente para avaliar designs de sequências de anticorpos. Isso nos ajudará a comparar o desempenho do seu modelo em nossos conjuntos de dados de forma eficiente.
Existe um arquivo de configuração: sabdab.yaml, que pode ser usado para alterar qualquer um dos parâmetros. Você pode treinar o modelo usando:
python train.py # For training.
- trem.py
-src
|-modelo.py
- configuração
|-sabdab.yaml
- utilitários
|-load_data.py
|-argumentos.py
|-model_utils.py
|-loss_functions.py
...
- dados
|-her2
...
Peso e preconceitos podem ser usados para rastrear experimentos. Ele está desativado por padrão, mas pode ser ativado alterando a opção no arquivo de configuração em ./config/sabdab.yaml
@article {Ucar2024.10.07.617023,
author = {Ucar, Talip and Malherbe, Cedric and Gonzalez Hernandez, Ferran},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
elocation-id = {2024.10.07.617023},
year = {2024},
doi = {10.1101/2024.10.07.617023},
publisher = {Cold Spring Harbor Laboratory},
URL = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023},
eprint = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023.full.pdf},
journal = {bioRxiv}
}
Se você usa DiffAbXL em seus próprios estudos e trabalhos, cite-o usando o seguinte:
@Misc{talip_ucar_2024_DiffAbXL,
author = {Talip Ucar},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
URL = {https://github.com/AstraZeneca/DiffAbXL},
month = {October},
year = {since 2024}
}