Реализация DiffAbXL оценивалась в документе: Изучение показателей логарифмического правдоподобия для ранжирования дизайнов последовательностей антител.
| Классифицировать | Модели | Абсци HER2 | Природа | АЗ Таргет-2 | пр.? | ||
|---|---|---|---|---|---|---|---|
| Нулевой выстрел | СПР-контроль | ХЕЛ | HER2 | ||||
| 1 | ДиффАбXL-A-DN | 0,43 | 0,22 | 0,62 | 0,37 | 0,41 | 0,41 |
| 2 | DiffAbXL-A-SG | 0,46 | 0,22 | 0,64 | -0,38 | 0,43 | 0,274 |
| 3 | ДиффАбXL-H3-DN | 0,49 | 0 | 0,52 | -0,08 | 0,37 | 0,26 |
| 4 | IgBlend (только структура) | 0,40 | 0,21 | 0,54 | -0,30 | 0,31 | 0,232 |
| 5 | Антифолд | 0,43 | 0,22 | 0,4 | -0,47 | 0,38 | 0,192 |
| 6 | ДиффАбXL-H3-SG | 0,48 | 0 | 0,4 | -0,41 | 0,29 | 0,152 |
| 7 | ЭСМ | 0,29 | 0 | 0 | 0,18 | 0,27 | 0,148 |
| 8 | ДиффАб | 0,34 | 0,21 | 0 | -0,14 | 0,22 | 0,126 |
| 9 | АбЛанг2 | 0,3 | 0 | 0 | -0,07 | 0,36 | 0,118 |
| 10 | IgBlend (только последующий) | 0,27 | 0 | 0 | -0,1 | 0,36 | 0,106 |
| 11 | АбЛанг | 0,3 | 0 | 0 | -0,13 | 0,35 | 0,104 |
| 12 | dyMEAN | 0,37 | 0,15 | 0 | 0 | 0 | 0,104 |
| 13 | АбХ | 0,28 | 0,19 | 0 | 0 | 0 | 0,094 |
| 14 | АнтиБЕРТи | 0,26 | 0 | 0 | -0,17 | 0,35 | 0,088 |
| 15 | ИМЕТЬ В ВИДУ | 0,36 | 0 | 0 | 0,02 | 0 | 0,076 |
| 16 | ЕСМ-ИФ | 0 | -0,27 | 0 | -0,53 | 0,42 | -0,076 |
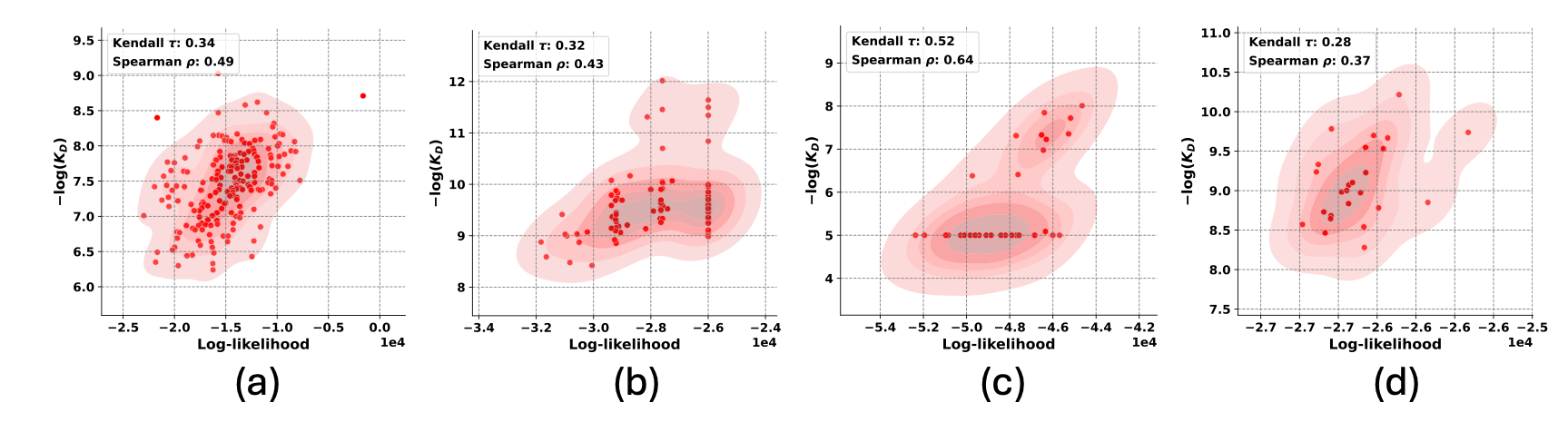
Рисунок 1: Результаты для DiffAbXL: a) DiffAbXL-H3-DN для данных HER2 с нулевым выстрелом Absci b) DiffAbXL-A-SG для AZ Target-2, c) DiffAbXL-A-SG для Nature HEL, d) DiffAbXL- A-DN для природы HER2.
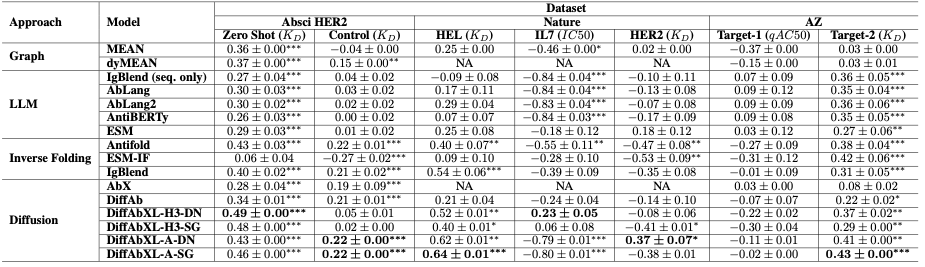
Таблица-1: Сводка результатов корреляции Спирмена. Сокращения: DN: режим De Novo, SG: режим наведения по структуре, NA: требуется эпитоп или сложная структура, но она недоступна. *, **, *** обозначают значения p ниже 0,05, 0,01 и 1e-4 соответственно.
Чтобы нам было проще протестировать вашу модель, мы рекомендуем вам реализовать интерфейс в виде метода Python в классе, который мы можем легко интегрировать с нашим конвейером оценки. Метод должен принимать следующие входные данные:
Метод должен возвращать словарь, содержащий:
Вот базовый шаблон на Python для реализации этого интерфейса:
def benchmark ( self , sequences , structure = None , mask = None , ** kwargs ):
"""
Benchmark the model on provided antibody sequences and structures.
Parameters:
sequences (list of str): List of antibody sequences.
structure (optional): Path to a PDB file. Currently, only one PDB file is provided per target dataset.
The PDB file may contain either just the antibody or an antibody-antigen complex,
depending on the dataset.
mask (optional): Binary list or array indicating the regions of interest in the sequences for metric calculations.
kwargs (optional): Additional parameters required by the model.
Returns:
dict: A dictionary containing log-likelihood scores and other relevant metrics.
"""
passУбедитесь, что ваша модель выводит показатели логарифмического правдоподобия в формате, который мы можем использовать непосредственно для сравнительного анализа конструкций последовательностей антител. Это поможет нам эффективно сравнить производительность вашей модели в наших наборах данных.
Существует один файл конфигурации: sabdab.yaml, который можно использовать для изменения любого параметра. Вы можете обучить модель, используя:
python train.py # For training.
- поезд.py
- источник
|-model.py
- конфигурация
|-sabdab.yaml
- утилиты
|-load_data.py
|-arguments.py
|-model_utils.py
|-loss_functions.py
...
- данные
|-her2
...
Вес и предвзятость можно использовать для отслеживания экспериментов. По умолчанию он отключен, но его можно включить, изменив параметр в файле конфигурации в ./config/sabdab.yaml
@article {Ucar2024.10.07.617023,
author = {Ucar, Talip and Malherbe, Cedric and Gonzalez Hernandez, Ferran},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
elocation-id = {2024.10.07.617023},
year = {2024},
doi = {10.1101/2024.10.07.617023},
publisher = {Cold Spring Harbor Laboratory},
URL = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023},
eprint = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023.full.pdf},
journal = {bioRxiv}
}
Если вы используете DiffAbXL в своих исследованиях и работе, пожалуйста, укажите его, используя следующее:
@Misc{talip_ucar_2024_DiffAbXL,
author = {Talip Ucar},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
URL = {https://github.com/AstraZeneca/DiffAbXL},
month = {October},
year = {since 2024}
}