ชุดเวิร์กโฟลว์ระดับโมเลกุลและการคำนวณสำหรับการจัดลำดับ RNA รวมเชิงพื้นที่ (STRS)
พื้นที่เก็บข้อมูลนี้มีโปรโตคอล ไปป์ไลน์ และสคริปต์ทั้งหมดที่คุณจำเป็นต้องใช้เพื่อเรียกใช้และวิเคราะห์ STRS ในตัวอย่างเกือบทั้งหมด!
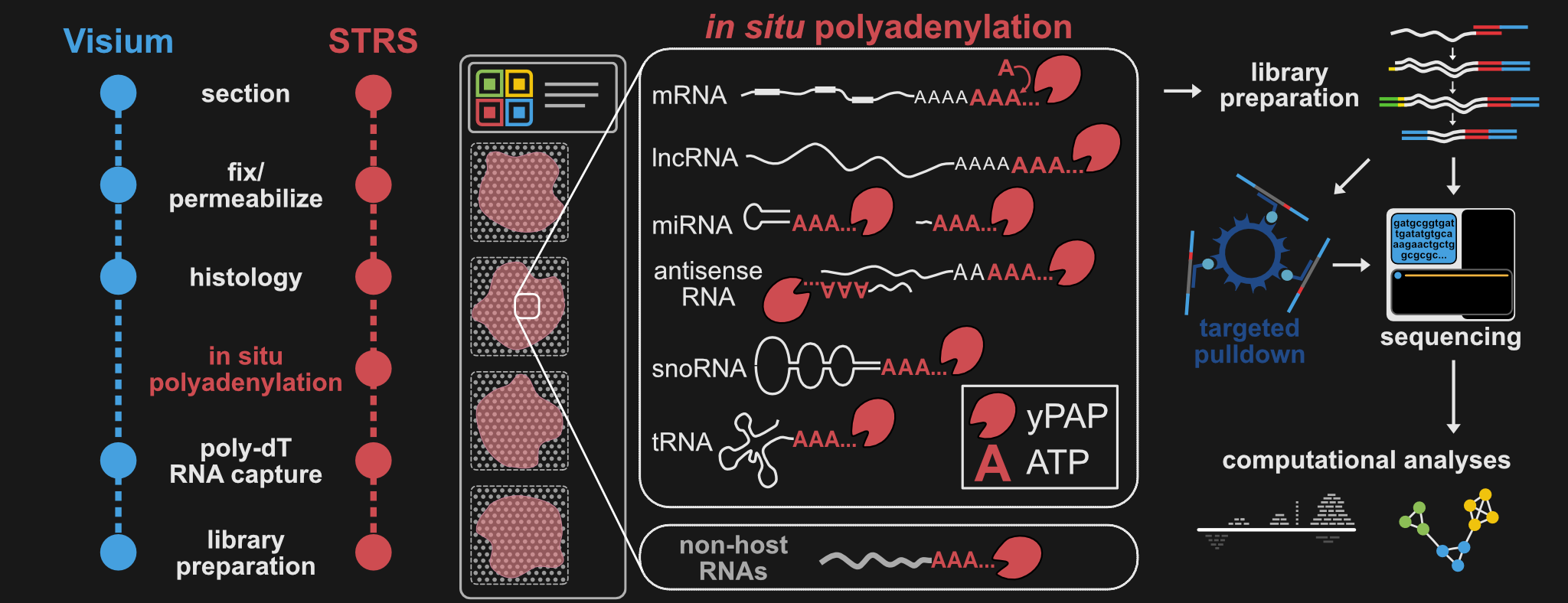
txg_snake ซึ่งเป็นไปป์ไลน์ที่อยู่ระหว่างดำเนินการสำหรับไลบรารี 10x โดยเฉพาะชุดข้อมูล STRS!McKellar และคณะ เทคโนโลยีชีวภาพธรรมชาติ 2022
ลิงก์ก่อนพิมพ์ bioRxiv
manuscripts : ไฟล์ .pdf ของฉบับพิมพ์ล่วงหน้าและต้นฉบับที่ตามมา (หวังว่า!)
protocols : เอกสาร Microsoft word และ .pdf ของโปรโตคอลที่ใช้ที่นี่
pipelines : เวิร์กโฟลว์ Snakemake ที่ใช้ในการศึกษาของเรา ซึ่งรวมถึงไปป์ไลน์การจัดตำแหน่งสำหรับข้อมูล STRS (kallisto, STARsolo และ miRge3.0), ข้อมูล RNAseq ขนาดเล็ก (STAR & miRge3.0), SmartSeqTotal (kallisto) และ VASAdrop (kallisto)
scripts : โค้ดอื่นๆ ทั้งหมดที่เราใช้! ส่วนใหญ่ประกอบด้วยสคริปต์ R และฟังก์ชันยูทิลิตี้ที่ใช้ในการวิเคราะห์เชิงพื้นที่ของเรา
references : ข้อมูลและสคริปต์เกี่ยวกับจีโนมอ้างอิงและคำอธิบายประกอบที่ใช้ในการศึกษานี้
resources : เมตาดาต้าสารพัน รายการยีน และข้อมูลอื่น ๆ ที่เราใช้ในการวิเคราะห์ข้อมูลของเรา โปรดดูไฟล์ README ในแต่ละไดเรกทอรีย่อยเพื่อดูรายละเอียดเพิ่มเติม
ชุดข้อมูล STRS: GSE200481
ข้อมูล RNAseq ขนาดเล็กของ SkM/หัวใจ: GSE200480
ข้อมูล RNAseq รวมนิวเคลียสเดี่ยว (สายเซลล์ C2C12): GSE209780
ข้อมูล RNA Atlas ขนาดเล็ก (Isakova et al, PNAS , 2020): GSE119661
ข้อมูล SmartSeqTotal (Isakova และคณะ PNAS , 2021): GSE151334
ข้อมูล VASAdrop (Salmen et al, Nature Biotechnology , 2022): GSE176588
อิไวจ์น เดอ วลามินค์ ([email protected])
เบนจามิน ดี. คอสโกรฟ ([email protected])
เดวิด ดับเบิลยู. แมคเคลลาร์ ([email protected])