การซื้อคืนนี้จัดเตรียมสคริปต์ของเราเพื่อสร้างความสัมพันธ์ทางพันธุกรรม corrplot (รูปที่ 2) โดยอิงตามผลลัพธ์การถดถอยของคะแนน LD แบบไบวาเรียต (Kanai, M. et al ., Nat. Genet. 2018)
install.packages("dplyr")
devtools::install_github("mkanai/corrplot")
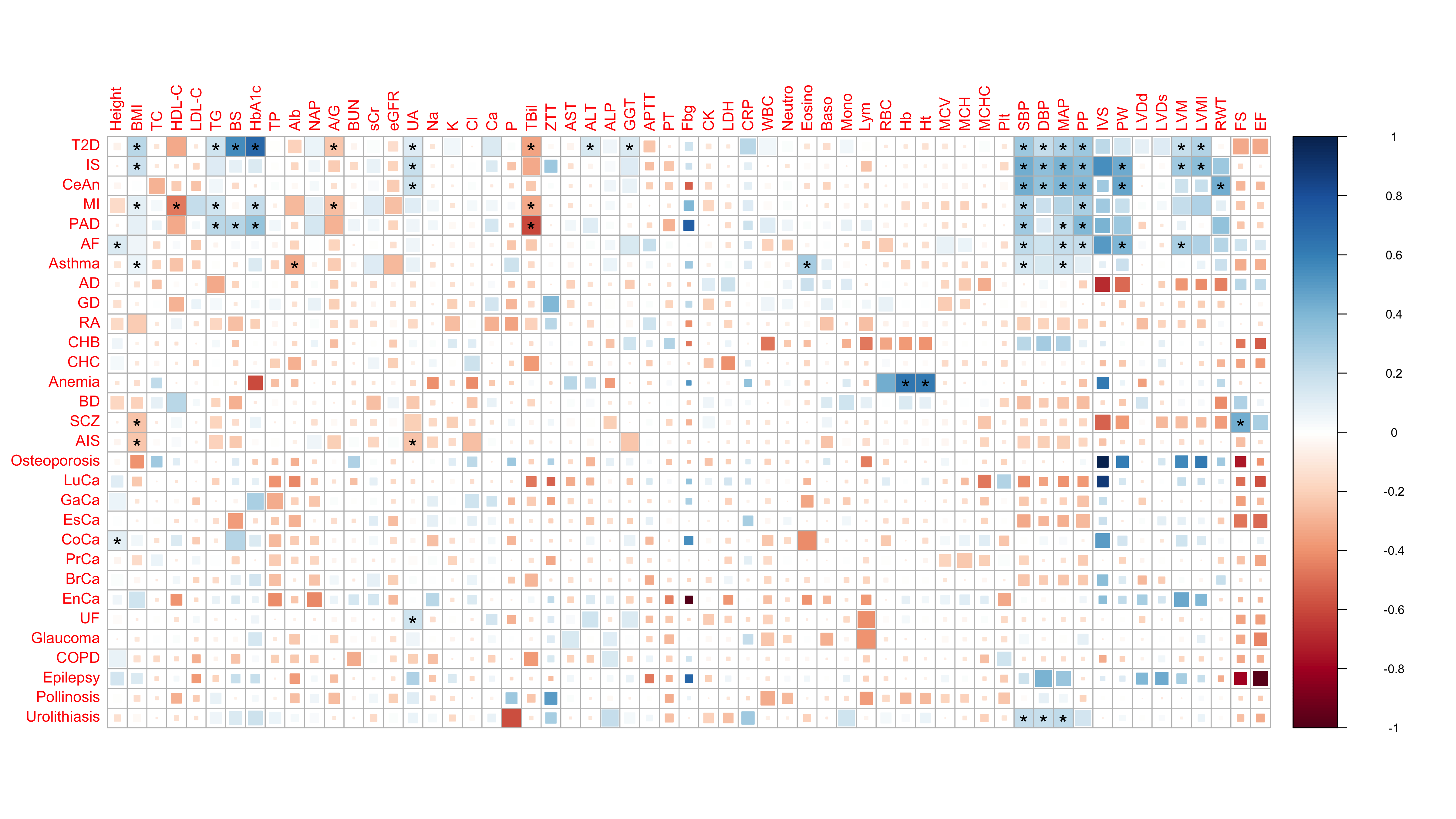
เราแก้ไข corrplot เพื่อให้เห็นภาพความสัมพันธ์ทางพันธุกรรมแบบคู่ที่ประเมินผ่านการถดถอยของคะแนน LD แบบไบวาเรียต สี่เหลี่ยมที่ใหญ่กว่าสอดคล้องกับ FDR ที่สำคัญกว่า ( corrplot(method = 'psquare', p.mat = p.mat, ...) ) ความสัมพันธ์ที่มีนัยสำคัญ (FDR < 0.05) ระบุด้วยเครื่องหมายดอกจัน ( corrplot(sig = 'pch', sig.level = 0.05, pch = '*') )
Rscript plot_corrplot_rg.R input_example/input_rg.txt input_example/traitlist.txt
input_rg.txt )ไฟล์นี้แสดงรายการความสัมพันธ์ทางพันธุกรรมแบบคู่ทั้งหมดที่ประเมินผ่านซอฟต์แวร์ ldsc สคริปต์คาดว่าแถวทั้งหมดจะไม่ซ้ำกัน ( เช่น หนึ่งแถวต่อคู่คุณลักษณะแต่ละคู่) ช่องที่ต้องกรอกมีดังนี้:
p1_category : หมวดหมู่ลักษณะของลักษณะที่ 1p1 : ลักษณะ 1p2_category : หมวดหมู่ลักษณะของลักษณะที่ 2p2 : ลักษณะ 2rg : ความสัมพันธ์ทางพันธุกรรมp : ค่า Pq : FDR ค่า qtraitlist.txt )ไฟล์นี้แสดงรายการลักษณะและหมวดหมู่ โดยจะกำหนดสีของแต่ละหมวดหมู่ในรูป ช่องที่ต้องกรอกมีดังนี้:
CATEGORY : หมวดลักษณะTRAIT : ชื่อลักษณะCOLOR : สีหมวดหมู่ตัวอย่างผลลัพธ์แสดงไว้ด้านล่าง เพื่อให้ได้ภาพที่เผยแพร่ เราได้แก้ไขเอาต์พุต pdf โดยใช้ Adobe Illustrator
แพ็คเกจ corrplot ดั้งเดิม:
ข้อมูลตัวอย่างและตัวเลขที่เผยแพร่:
มาซาฮิโระ คานาอิ ([email protected])
http://mkanai.github.io/