โปรดทราบว่าพื้นที่เก็บข้อมูลนี้มีไว้เพื่อวัตถุประสงค์ในการเก็บถาวรเท่านั้น ฟังก์ชัน ANCOM แบบสแตนด์อโลนในพื้นที่เก็บข้อมูลนี้ไม่ได้รับการดูแลรักษาอีกต่อไป ขอแนะนำอย่างยิ่งให้ผู้ใช้ใช้ฟังก์ชัน ancom หรือ ancombc ในแพ็คเกจ ANCOMBC R ของเรา สำหรับรายงานข้อผิดพลาด โปรดไปที่พื้นที่เก็บข้อมูล ANCOMBC
โค้ดปัจจุบันใช้ ANCOM ในชุดข้อมูลแบบตัดขวางและตามยาวในขณะที่อนุญาตให้ใช้ตัวแปรร่วมได้ จำเป็นต้องรวมไลบรารีต่อไปนี้เพื่อให้โค้ด R ทำงาน:
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )เรานำวิธีการของ ANCOM-II มาใช้เป็นขั้นตอนก่อนการประมวลผลเพื่อจัดการกับศูนย์ประเภทต่างๆ ก่อนที่จะทำการวิเคราะห์ความอุดมสมบูรณ์เชิงอนุพันธ์
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : กรอบข้อมูลหรือเมทริกซ์ที่แสดงถึงตาราง OTU/SV ที่สังเกตได้โดยมีแท็กซ่าในแถว ( rownames ) และตัวอย่างในคอลัมน์ ( colnames ) โปรดทราบว่านี่คือตารางความอุดม สมบูรณ์สัมบูรณ์ อย่าแปลงเป็นตารางความอุดม สมบูรณ์ สัมพัทธ์ (โดยที่ผลรวมของคอลัมน์เท่ากับ 1)meta_data : กรอบข้อมูลหรือเมทริกซ์ของตัวแปรทั้งหมดและตัวแปรร่วมที่น่าสนใจsample_var : ตัวละคร ชื่อของคอลัมน์ที่เก็บ ID ตัวอย่างgroup_var : ตัวละคร ชื่อของตัวบ่งชี้กลุ่ม group_var จำเป็นสำหรับการตรวจจับค่าศูนย์และค่าผิดปกติของโครงสร้าง สำหรับคำจำกัดความของศูนย์ต่างๆ (ศูนย์โครงสร้าง ศูนย์ค่าผิดปกติ และศูนย์สุ่มตัวอย่าง) โปรดดูที่ ANCOM-IIout_cut เศษส่วนที่เป็นตัวเลขระหว่าง 0 ถึง 1 สำหรับแต่ละอนุกรมวิธาน การสังเกตที่มีสัดส่วนการกระจายของส่วนผสมน้อยกว่า out_cut จะถูกตรวจพบว่าเป็นศูนย์ที่มีค่าผิดปกติ ในขณะที่การสังเกตที่มีสัดส่วนการกระจายตัวของส่วนผสมมากกว่า 1 - out_cut จะถูกตรวจพบว่าเป็นค่าผิดปกติzero_cut : เศษส่วนที่เป็นตัวเลขระหว่าง 0 ถึง 1 แท็กซ่าที่มีสัดส่วนของศูนย์มากกว่า zero_cut จะไม่รวมอยู่ในการวิเคราะห์lib_cut : ตัวเลข ตัวอย่างที่มีขนาดไลบรารีน้อยกว่า lib_cut จะไม่รวมอยู่ในการวิเคราะห์neg_lb : เชิงตรรกะ TRUE บ่งชี้ว่าอนุกรมวิธานจะถูกจัดประเภทเป็นศูนย์โครงสร้างในกลุ่มทดลองที่เกี่ยวข้องโดยใช้ขอบเขตล่างกำกับของมัน โดยเฉพาะอย่างยิ่ง neg_lb = TRUE บ่งชี้ว่าคุณกำลังใช้ทั้งสองเกณฑ์ที่ระบุไว้ในส่วน 3.2 ของ ANCOM-II เพื่อตรวจหาศูนย์โครงสร้าง มิฉะนั้น neg_lb = FALSE จะใช้สมการ 1 ในส่วนที่ 3.2 ของ ANCOM-II เท่านั้นในการประกาศค่าศูนย์เชิงโครงสร้าง feature_table : กรอบข้อมูลของตาราง OTU ที่ประมวลผลล่วงหน้าmeta_data : กรอบข้อมูลของข้อมูลเมตาที่ประมวลผลล่วงหน้าstructure_zeros : เมทริกซ์ประกอบด้วย 0 และ 1 โดยที่ 1 ระบุว่าอนุกรมวิธานถูกระบุเป็นศูนย์โครงสร้างในกลุ่มที่เกี่ยวข้องANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : กรอบข้อมูลที่แสดงถึงตาราง OTU/SV โดยมีแท็กซ่าในแถว ( rownames ) และตัวอย่างในคอลัมน์ ( colnames ) อาจเป็นค่าเอาต์พุตจาก feature_table_pre_process โปรดทราบว่านี่คือตารางความอุดม สมบูรณ์สัมบูรณ์ อย่าแปลงเป็นตารางความอุดม สมบูรณ์ สัมพัทธ์ (โดยที่ผลรวมของคอลัมน์เท่ากับ 1)meta_data : กรอบข้อมูลของตัวแปร สามารถเป็นค่าเอาต์พุตจาก feature_table_pre_processstruc_zero : เมทริกซ์ประกอบด้วย 0 และ 1s โดยที่ 1 ระบุว่าอนุกรมวิธานถูกระบุเป็นศูนย์โครงสร้างในกลุ่มที่เกี่ยวข้อง สามารถเป็นค่าเอาต์พุตจาก feature_table_pre_processmain_var : ตัวละคร ชื่อของตัวแปรหลักที่สนใจ ปัจจุบัน ANCOM v2.1 รองรับ main_var ตามหมวดหมู่p_adjust_method : ตัวละคร การระบุวิธีการปรับค่า p สำหรับการเปรียบเทียบหลายรายการ ค่าเริ่มต้นคือ “BH” (ขั้นตอนของ Benjamini-Hochberg)alpha : ระดับนัยสำคัญ ค่าเริ่มต้นคือ 0.05adj_formula : สตริงอักขระที่แสดงถึงสูตรสำหรับการปรับปรุง (ดูตัวอย่าง)rand_formula : สตริงอักขระที่แสดงถึงสูตรสำหรับเอฟเฟกต์แบบสุ่มใน lme สำหรับรายละเอียด โปรดดูที่ ?lmelme_control : รายการที่ระบุค่าควบคุมสำหรับ lme fit สำหรับรายละเอียด โปรดดูที่ ?lmeControl 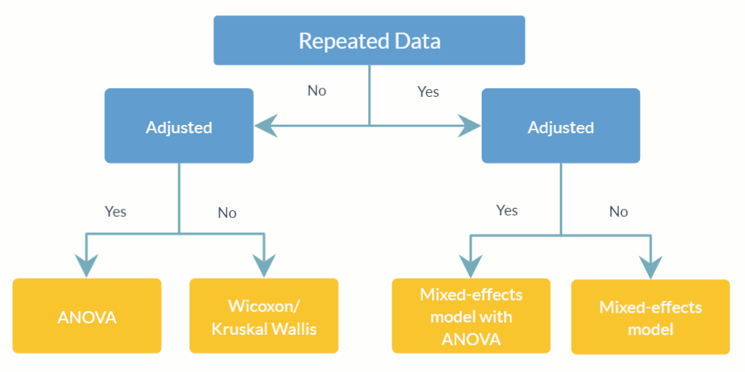
out : กรอบข้อมูลที่มีสถิติ W สำหรับแต่ละแท็กซ่าและคอลัมน์ต่อๆ ไป ซึ่งเป็นตัวบ่งชี้เชิงตรรกะว่า OTU หรือแท็กซอนมีความอุดมสมบูรณ์แตกต่างกันภายใต้ชุดของจุดตัด (0.9, 0.8, 0.7 และ 0.6) detected_0.7 เป็นที่นิยมใช้ อย่างไรก็ตาม คุณสามารถเลือก detected_0.8 หรือ detected_0.9 หากคุณต้องการอนุรักษ์ผลลัพธ์ของคุณมากขึ้น (FDR น้อยลง) หรือใช้ detected_0.6 หากคุณต้องการสำรวจการค้นพบเพิ่มเติม (กำลังมากขึ้น)
fig : วัตถุ ggplot ของแปลงภูเขาไฟ
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var ที่นี่เราต้องการทราบว่ามีศูนย์โครงสร้างบางส่วนในระดับต่างๆ ของ delivery หรือไม่ library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var ที่นี่เราต้องการทราบว่ามีศูนย์โครงสร้างบางส่วนในระดับต่างๆ ของ delivery หรือไม่ library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var ที่นี่เราต้องการทราบว่ามีศูนย์โครงสร้างบางส่วนในระดับต่างๆ ของ delivery หรือไม่ library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )