Repo นี้ประกอบด้วย:
sepal ต้องการ python3 โดยควรเป็นเวอร์ชันที่ใหม่กว่าหรือเท่ากับ 3.5 หากต้องการดาวน์โหลดและติดตั้ง ให้เปิดเทอร์มินัลแล้วเปลี่ยนเป็นไดเร็กทอรีที่คุณต้องการให้ sepal ดาวน์โหลดและทำดังนี้
git clone https://github.com/almaan/sepal.git
cd sepal
chmod +x setup.py
./setup.py install
ขึ้นอยู่กับสิทธิ์ผู้ใช้ของคุณ คุณอาจต้องเพิ่ม --user เป็นอาร์กิวเมนต์ใน setup.py การเรียกใช้การตั้งค่าจะทำให้คุณมีการติดตั้งขั้นต่ำที่จำเป็นในการคำนวณเวลาการแพร่กระจาย อย่างไรก็ตาม หากคุณต้องการใช้โมดูลการวิเคราะห์ คุณจะต้องติดตั้งแพ็คเกจที่แนะนำด้วย หากต้องการทำสิ่งนี้ เพียง (ในไดเร็กทอรีเดียวกัน) ให้รัน:
pip install -e " .[full] " อีกครั้ง --user อาจจำเป็นต้องรวมไว้ด้วย นอกจากนี้ คุณอาจต้องใช้ pip3 หากนี่คือวิธีการตั้งค่าอินเทอร์เฟซ python-pip ของคุณ หากคุณใช้ conda หรือสภาพแวดล้อมเสมือน ให้ทำตามคำแนะนำในการติดตั้งแพ็คเกจ
สิ่งนี้ควรติดตั้งทั้งอินเทอร์เฟซบรรทัดคำสั่ง (CLI) และแพ็คเกจมาตรฐาน หากต้องการทดสอบและดูว่าการติดตั้งสำเร็จหรือไม่ คุณสามารถลองใช้คำสั่ง:
sepal -h
ซึ่งควรพิมพ์ข้อความช่วยเหลือที่เกี่ยวข้องกับ sepal หากทุกอย่างได้ผลสำหรับคุณจนถึงตอนนี้ คุณสามารถไปที่ส่วนตัวอย่างเพื่อดูการทำงานของ sepal !
การใช้งาน sepal ที่แนะนำคือโดยอินเทอร์เฟซบรรทัดคำสั่ง ทั้งการจำลองเพื่อคำนวณเวลาการแพร่กระจายตลอดจนการวิเคราะห์ในภายหลังหรือการตรวจสอบผลลัพธ์สามารถทำได้ง่าย ๆ โดยพิมพ์ sepal ตามด้วย run หรือ analyze โมดูล analyze มีตัวเลือกที่แตกต่างกัน ในการแสดงภาพผลลัพธ์ ( inspect ) จัดเรียงโปรไฟล์เป็นตระกูลรูปแบบ ( family ) หรือกำหนดให้ตระกูลที่ระบุวิเคราะห์การเพิ่มคุณค่าการทำงาน ( fea ) สำหรับรายการคำสั่งทั้งหมดที่มี ให้ทำ sepal module -h โดยที่ module เป็นหนึ่งใน run and analyze ด้านล่างนี้ เราแสดงให้เห็นว่าอาจใช้ sepal เพื่อค้นหาโปรไฟล์การถอดเสียงด้วยรูปแบบเชิงพื้นที่ได้อย่างไร
เราจะสร้างโฟลเดอร์เพื่อเก็บผลลัพธ์ ซึ่งจะถือเป็นไดเร็กทอรีการทำงานของเราด้วย จากไดเร็กทอรีหลักของ repo ให้ทำดังนี้
cd res
mkdir example
cd exampleตัวอย่าง MOB จะถูกนำมาใช้เป็นตัวอย่างในการวิเคราะห์ของเรา เราเริ่มต้นด้วยการคำนวณเวลาการแพร่กระจายสำหรับแต่ละโปรไฟล์การถอดเสียง:
sepal run -c ../../data/real/mob.tsv.gz -mo 10 -mc 5 -o . -ar 1ด้านล่างนี้คือตัวอย่าง (พร้อมการแสดงคำสั่งช่วยเหลือเพิ่มเติม) ว่าจะมีลักษณะอย่างไร

เมื่อคำนวณเวลาการแพร่กระจายแล้ว เราต้องการตรวจสอบผลลัพธ์ เช่นเดียวกับในการศึกษา เราจะดูโปรไฟล์ 20 อันดับแรก เราสามารถสร้างภาพจากผลลัพธ์ของเราได้อย่างง่ายดายโดยการรันคำสั่ง:
sepal analyze -c ../../data/real/mob.tsv.gz
-r 20200409173043610345-top-diffusion-times.tsv
-ar 1k -o . inspect -ng 20 -nc 5ซึ่งจะมีลักษณะบางอย่างในบรรทัดนี้:

ผลลัพธ์จะเป็นรูปภาพต่อไปนี้:
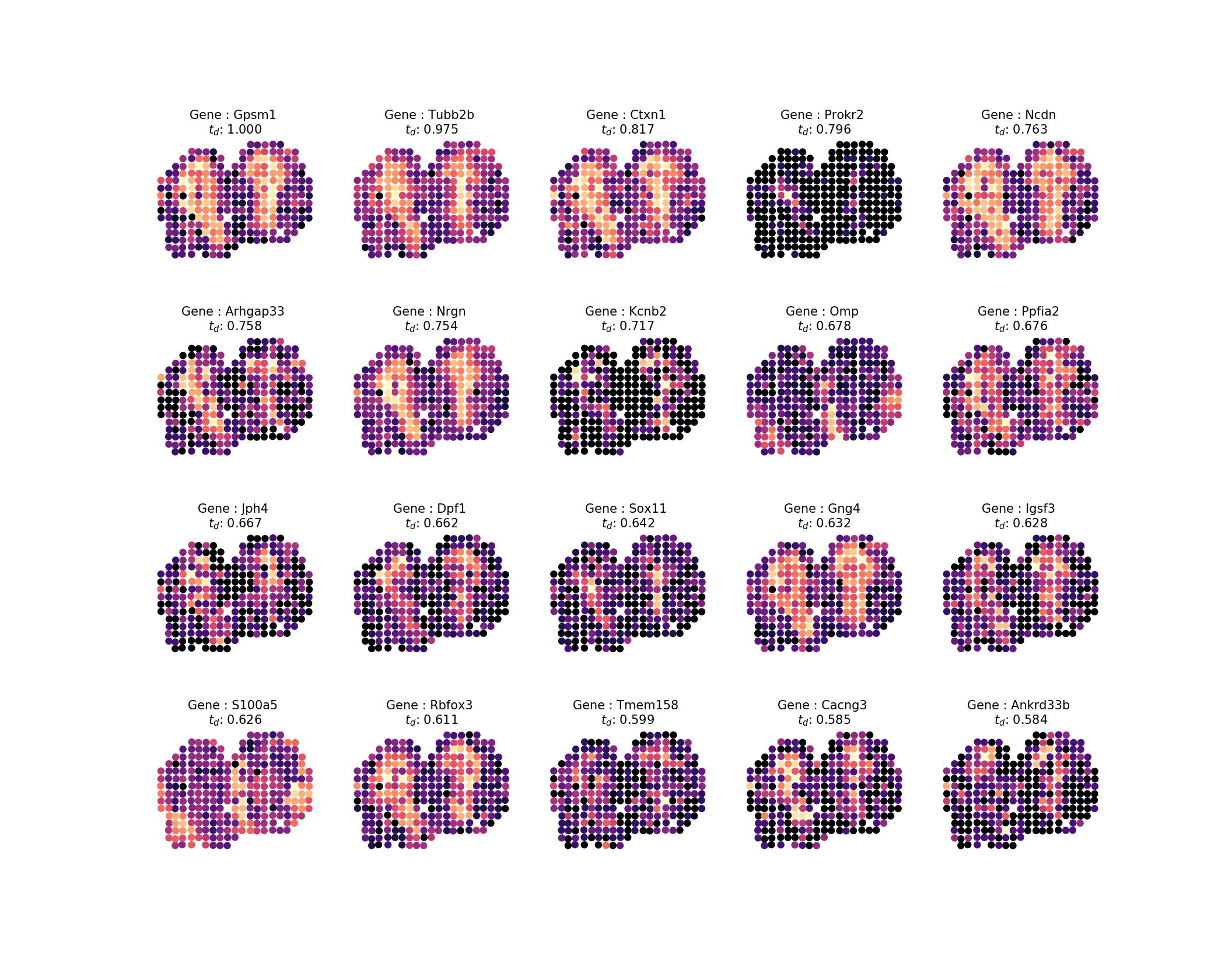
จากนั้น เพื่อจัดเรียงยีนอันดับสูงสุด 100 ยีนให้อยู่ในกลุ่มตระกูลรูปแบบ โดยที่ 85% ของความแปรปรวนในรูปแบบของเราควรอธิบายด้วยรูปแบบลักษณะเฉพาะ ให้ทำดังนี้
sepal analyze -c ../../data/real/mob.tsv.gz
-r 20200409173043610345-top-diffusion-times.tsv
-ar 1k -o . family -ng 100 -nbg 100 -eps 0.85 --plot -nc 3จากนี้ เราได้รับแรงจูงใจที่เป็นตัวแทนสามประการต่อไปนี้สำหรับแต่ละครอบครัว:
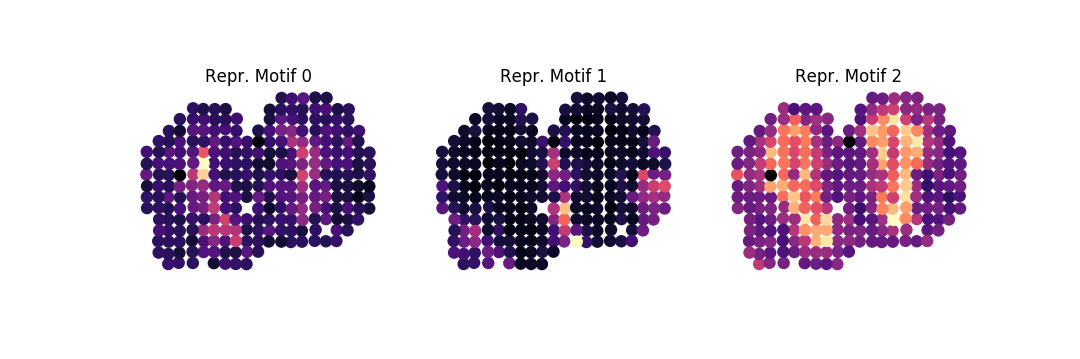
เราอาจให้ครอบครัวของเราได้รับการวิเคราะห์เสริมคุณค่าโดยดำเนินการ:
sepal analyze -c ../../data/real/mob.tsv.gz
-r 20200409173043610345-top-diffusion-times.tsv
-ar 1k -o . fea -fl mob.tsv-family-index.tsv -or " mmusculus "โดยที่เราเห็นว่าแฟมิลี่ 2 ได้รับการเสริมสมรรถนะสำหรับกระบวนการต่างๆ ที่เกี่ยวข้องกับการทำงานของเซลล์ประสาท การสร้าง และการควบคุม:
| ตระกูล | พื้นเมือง | ชื่อ | p_value | แหล่งที่มา | ทางแยก_ขนาด | |
|---|---|---|---|---|---|---|
| 2 | 2 | ไป:0007399 | การพัฒนาระบบประสาท | 0.00035977 | ไป:บีพี | 26 |
| 3 | 2 | ไป:0050773 | การควบคุมการพัฒนาเดนไดรต์ | 0.000835883 | ไป:บีพี | 8 |
| 4 | 2 | ไป:0048167 | การควบคุมความเป็นพลาสติกแบบซินแนปติก | 0.00196494 | ไป:บีพี | 8 |
| 5 | 2 | ไป:0016358 | การพัฒนาเดนไดรต์ | 0.00217167 | ไป:บีพี | 9 |
| 6 | 2 | ไป:0048813 | การสร้างสัณฐานวิทยาของเดนไดรต์ | 0.00741589 | ไป:บีพี | 7 |
| 7 | 2 | ไป:0048814 | การควบคุมการสร้างสัณฐานวิทยาของเดนไดรต์ | 0.00800399 | ไป:บีพี | 6 |
| 8 | 2 | ไป:0048666 | การพัฒนาเซลล์ประสาท | 0.0114088 | ไป:บีพี | 16 |
| 9 | 2 | ไป:0099004 | เส้นทางการส่งสัญญาณไคเนสที่ขึ้นอยู่กับความสงบ | 0.0159572 | ไป:บีพี | 3 |
| 10 | 2 | ไป:0050804 | การปรับการส่งผ่านซินแนปติกเคมี | 0.0341913 | ไป:บีพี | 10 |
| 11 | 2 | ไป:0099177 | การควบคุมการส่งสัญญาณทรานส์ซินแนปติก | 0.0347783 | ไป:บีพี | 10 |
แน่นอนว่าการวิเคราะห์นี้ไม่ได้ละเอียดถี่ถ้วนนัก แต่เป็นตัวอย่างสั้นๆ เพื่อแสดงวิธีการทำงานของ CLI สำหรับ sepal
แม้ว่า sepal ได้รับการออกแบบให้เป็นเครื่องมือแบบสแตนด์อโลน แต่เรายังสร้างให้ทำงานเป็นแพ็คเกจ Python มาตรฐานซึ่งสามารถนำเข้าและใช้งานฟังก์ชันในเวิร์กโฟลว์แบบรวมได้ เพื่อแสดงให้เห็นว่าสามารถทำได้อย่างไร เราได้ยกตัวอย่างโดยจำลองการวิเคราะห์มะเร็งผิวหนัง อาจมีการเพิ่มตัวอย่างเพิ่มเติมในภายหลัง
อินพุตไปยัง sepal จะต้องอยู่ในรูปแบบ n_locations x n_genes อย่างไรก็ตาม หากข้อมูลของคุณมีโครงสร้างในทางตรงกันข้าม ( n_genes x n_locations ) เพียงแค่ระบุแฟล็ก --transpose เมื่อรันการจำลองหรือการวิเคราะห์ และสิ่งนี้จะได้รับการดูแล ของ.
ขณะนี้เรารองรับรูปแบบ .csv , .tsv และ .h5ad ประการหลัง ไฟล์ของคุณควรได้รับการจัดโครงสร้างตามรูปแบบนี้ เราคาดหวังว่าจะมีการเผยแพร่จากทีมงาน scanpy ในอนาคตอันใกล้นี้ โดยมีการนำเสนอรูปแบบมาตรฐานสำหรับข้อมูลเชิงพื้นที่ แต่จนกว่าจะถึงตอนนั้น เราจะใช้มาตรฐานดังกล่าว
ข้อมูลจริงทั้งหมดที่เราใช้นั้นเป็นข้อมูลสาธารณะ และสามารถเข้าถึงได้จากลิงก์ต่อไปนี้:
ข้อมูลสังเคราะห์ถูกสร้างขึ้นโดย:
synthetic/img2cnt.pysynthetic/turing.pysynthetic/ablation.py ผลลัพธ์ทั้งหมดที่นำเสนอในการศึกษานี้สามารถพบได้ในโฟลเดอร์ res ทั้งข้อมูลจริงและข้อมูลสังเคราะห์ สำหรับแต่ละตัวอย่าง เราได้จัดโครงสร้างผลลัพธ์ตามนั้น:
res/sample-name/X-diffusion-times.tsv : เวลาการแพร่กระจายของยีนที่ได้รับการจัดอันดับทั้งหมดanalysis/ : มีเอาต์พุตของการวิเคราะห์รอง