การโหลดข้อมูลแบบแบตช์อย่างรวดเร็วของไฟล์ BigWig ที่มีข้อมูลแทร็ก epigentic และลำดับที่สอดคล้องกันซึ่งขับเคลื่อนโดย GPU สำหรับแอปพลิเคชันการเรียนรู้เชิงลึก
Bigwig-loader ส่วนใหญ่ขึ้นอยู่กับไลบรารี่ Rapidsai kvikio และ Cupy ซึ่งทั้งสองอย่างนี้ติดตั้งได้ดีที่สุดโดยใช้ conda/mamba Bigwig-loader สามารถติดตั้งได้โดยใช้ conda/mamba หากต้องการสร้างสภาพแวดล้อมใหม่โดยติดตั้ง bigwig-loader:
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loaderหรือเพิ่มสิ่งนี้ลงในไฟล์ environment.yml ของคุณ:
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loaderและอัปเดต:
mamba env update -f environment.ymlBigwig-loader สามารถติดตั้งได้โดยใช้ pip ในสภาพแวดล้อมที่มีไลบรารี่ Rapidsai kvikio และ Cupy ติดตั้งอยู่แล้ว:
pip install bigwig-loaderเรารวม BigWigDataset ไว้ในชุดข้อมูลที่ทำซ้ำได้ของ PyTorch ซึ่งคุณสามารถใช้ได้โดยตรง:
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step () ออบเจ็กต์ Dataset ที่ไม่เชื่อเรื่องกรอบงานสามารถนำเข้าได้จาก bigwig_loader.dataset ออบเจ็กต์ชุดข้อมูลนี้ส่งคืนเทนเซอร์แบบถ้วย Cupy tensors ยึดติดกับอินเทอร์เฟซอาร์เรย์ cuda และสามารถแปลงเป็นศูนย์คัดลอกเป็น JAX หรือเทนเซอร์โฟลว์ได้
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)ดูไดเร็กทอรีตัวอย่างสำหรับตัวอย่างเพิ่มเติม
ไลบรารีนี้มีไว้สำหรับการโหลดชุดข้อมูลที่มีมิติเท่ากัน ซึ่งช่วยให้เกิดข้อสันนิษฐานบางประการที่สามารถทำให้กระบวนการโหลดเร็วขึ้นได้ ดังที่เห็นได้จากแผนภาพด้านล่าง เมื่อโหลดข้อมูลจำนวนเล็กน้อย pyBigWig จะทำงานได้เร็วมาก แต่ไม่ได้ใช้ประโยชน์จากลักษณะการโหลดข้อมูลแบบแบตช์สำหรับการเรียนรู้ของเครื่อง
ในเกณฑ์มาตรฐานด้านล่าง เรายังสร้างตัวโหลดข้อมูล PyTorch (ด้วย set_start_method('spawn')) โดยใช้ pyBigWig เพื่อเปรียบเทียบกับสถานการณ์จริงที่ต้องใช้ CPU หลายตัวต่อ GPU เราเห็นว่าปริมาณงานของตัวโหลดข้อมูล CPU ไม่ได้เพิ่มขึ้นเป็นเส้นตรงกับจำนวน CPU ดังนั้นจึงเป็นเรื่องยากที่จะได้รับปริมาณงานที่จำเป็นเพื่อรักษา GPU ฝึกโครงข่ายประสาทเทียมให้อิ่มตัวในระหว่างขั้นตอนการเรียนรู้
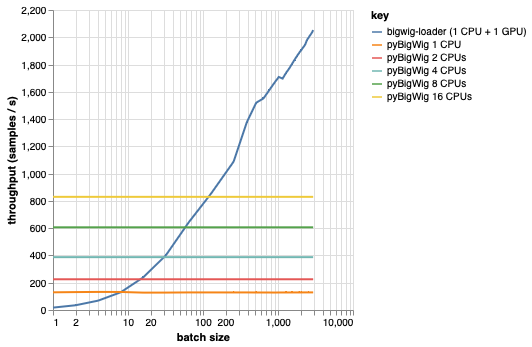
นี่คือปัญหาที่ bigwig-loader แก้ไขได้ นี่คือตัวอย่างวิธีใช้ bigwig-loader:
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.yml ในสภาพแวดล้อมนี้ คุณควรจะสามารถรัน pytest -v และเห็นว่าการทดสอบสำเร็จ หมายเหตุ: คุณต้องมี GPU เพื่อใช้ bigwig-loader!
ส่วนนี้จะแนะนำคุณตลอดขั้นตอนที่จำเป็นในการเพิ่มฟังก์ชันการทำงานใหม่ หากมีอะไรไม่ชัดเจนกรุณาเปิดประเด็น
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install เพื่อติดตั้ง hooks ล่วงหน้าการทดสอบอยู่ในไดเร็กทอรีการทดสอบ หนึ่งในการทดสอบที่สำคัญที่สุดคือ test_against_pybigwig ซึ่งจะทำให้แน่ใจได้ว่าหากมีข้อผิดพลาดใน pyBigWIg มันจะอยู่ใน bigwig-loader ด้วย
pytest -vv .เมื่อ GitHub Runner ที่มี GPU จะพร้อมใช้งาน เราก็อยากจะทำการทดสอบเหล่านี้ใน CI ด้วย แต่สำหรับตอนนี้ คุณสามารถเรียกใช้งานได้ในเครื่อง
หากคุณใช้ไลบรารีนี้ โปรดพิจารณาการอ้างอิง:
รีเทล, ยอเรน เซบาสเตียน, อันเดรียส โพห์ลมันน์, จอช ชิอู, อันเดรียส สเตฟเฟน และยอร์ค-อาร์เน เคลเวิร์ต “ตัวโหลดข้อมูลการเรียนรู้ของเครื่องจักรที่รวดเร็วสำหรับแทร็ก Epigenetic จากไฟล์ BigWig” ชีวสารสนเทศศาสตร์ 40, no. 1 (1 มกราคม 2024): btad767. https://doi.org/10.1093/bioinformatics/btad767.
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}